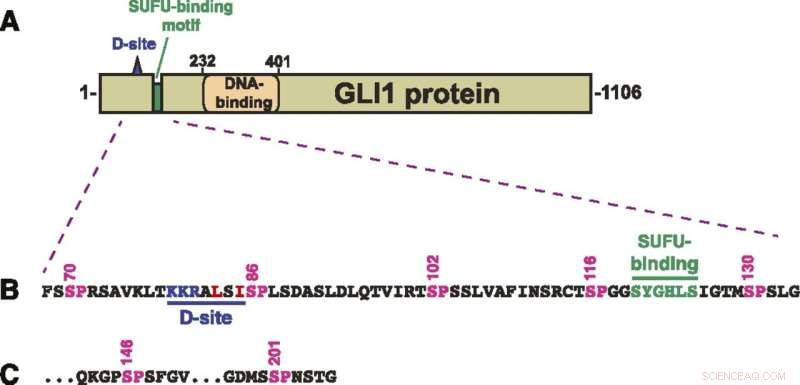
Estrutura da proteína GLI1. (A) Esquema da estrutura primária da proteína GLI1 humana, mostrando o sítio de ancoragem MAPK (sítio D), o motivo de ligação SUFU e o domínio de ligação ao DNA do dedo de zinco. O domínio de ativação transcricional constitui uma grande fração da metade C-terminal da proteína. (B) Sequência de aminoácidos dos resíduos 68-134, mostrando o sítio D, o motivo de ligação SUFU e um agrupamento de cinco sítios de fosforilação MAPK canônicos (SP). Os números acima mostram a posição dos resíduos de serina nos sítios SP. (C) Os dois últimos dos sete sítios de fosforilação SP MAPK contidos em resíduos de 68-232 GLI1. Após S201, o próximo site SP ou TP ocorre em S441. Crédito:A. Jane Bardwell et al, Life Science Alliance (2022). DOI:10.26508/lsa.202101353
Melhores tratamentos para algumas das formas mais mortais de câncer podem estar mais próximos devido a uma descoberta liderada pela Universidade da Califórnia, Irvine, sobre como uma certa proteína é ativada em células tumorais. A descoberta, liderada por pesquisadores da Escola de Ciências Biológicas, pode eventualmente levar a possíveis terapias para o melanoma especialmente perigoso e o adenocarcinoma pancreático, bem como o tipo mais comum de câncer cerebral infantil e câncer de pele adulto. Um artigo descrevendo o projeto aparece na
Life Science Alliance .
A descoberta diz respeito à proteína GLI1, que é importante no desenvolvimento celular, mas também foi encontrada ativada em vários tipos de câncer. GLI1 é tipicamente ativado pela via de sinalização Hedgehog, conhecida como HH. No entanto, os cientistas sabem há cerca de uma década que o crosstalk (interação) entre HH e a via da proteína quinase ativada por mitógeno tem um papel nos cânceres.
"Em alguns casos, as proteínas em uma via podem ativar proteínas em outra", disse o principal autor A. Jane Bardwell, cientista do projeto no Departamento de Desenvolvimento e Biologia Celular da UCI. "É um sistema complexo. Queríamos entender o mecanismo molecular que leva o GLI1 a ser ativado por proteínas na via MAPK."
GLI1 normalmente se liga firmemente a uma proteína chamada SUFU. Essa proteína suprime o GLI1, impedindo-o de penetrar nos núcleos das células e ativar os genes. Os cientistas examinaram sete locais na proteína GLI1 que poderiam ser fosforilados, ou ter um grupo fosfato transferido para ela.
"Identificamos três que podem ser fosforilados e estão envolvidos no enfraquecimento da ligação entre GLI1 e SUFU", disse Lee Bardwell, professor de desenvolvimento e biologia celular, cujo laboratório conduziu o projeto. "Esse processo ativa o GLI1, permitindo que ele entre no núcleo das células, onde pode causar um crescimento descontrolado, resultando em câncer".
Ele observou que a fosforilação de todos os três locais causa um nível significativamente maior de escape de GLI1 do SUFU do que se apenas um ou mesmo dois deles receberem grupos fosfato.
A descoberta é um passo significativo para tratamentos de câncer mais eficazes e personalizados. "Se pudermos entender exatamente o que está acontecendo em um determinado câncer ou tumor em particular, pode ser possível desenvolver uma droga específica para um tumor específico ou paciente individual", disse Bardwell. "Isso nos permitiria tratar essas doenças sem a toxicidade da quimioterapia básica". Além disso, muitos tumores do mesmo câncer apresentam mutações diferentes entre os indivíduos. Eventualmente, pode ser viável rastrear tumores para desenvolver a melhor abordagem para cada um.
A pesquisa foi realizada em colaboração com o Departamento de Microbiologia e Genética Molecular da Faculdade de Medicina da UCI e o Departamento de Dermatologia da Faculdade de Medicina da Universidade de Stanford.
