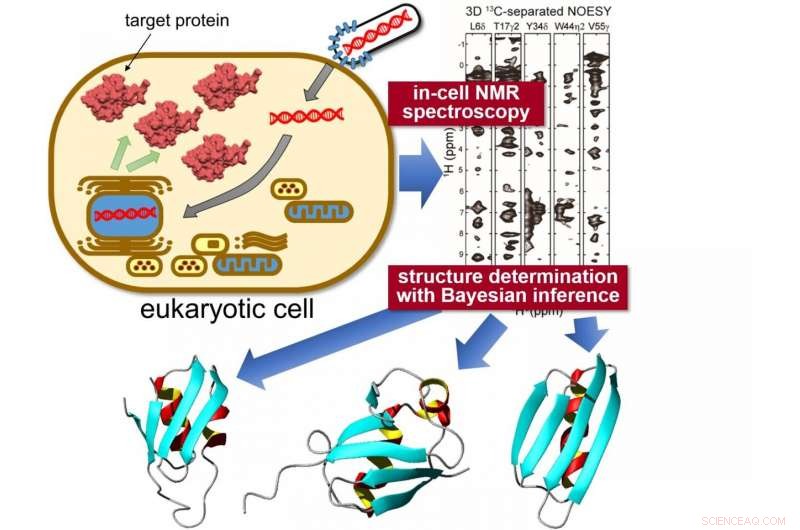
As proteínas alvo são expressas dentro sf9 células, então medido usando espectroscopia de NMR "na célula". Análise estatística única com a ajuda de Inferência bayesiana é aplicado para calcular a estrutura 3D precisa das proteínas com uma precisão sem precedentes. Crédito:Tokyo Metropolitan University
Pesquisadores da Tokyo Metropolitan University determinaram com sucesso a alta resolução, estrutura tridimensional de proteínas dentro de células eucarióticas vivas. Eles combinaram espectroscopia de ressonância magnética nuclear (NMR) "in-cell", um sistema de biorreator e algoritmos computacionais de ponta para determinar estruturas de proteínas em ambientes intracelulares lotados pela primeira vez. A técnica promete uma visão sobre o comportamento intracelular de proteínas causadoras de doenças e novas aplicações de triagem de drogas, permitindo a visualização in situ de como as proteínas respondem a estímulos bioquímicos.
As células eucarióticas são os blocos de construção de uma vasta gama de organismos, incluindo todos os fungos, plantas e animais. Sua estrutura interna é extremamente complexa e variada, com uma hierarquia estrutural intrincada e uma vasta gama de biomacromoléculas distribuídas em torno de uma rede citoesquelética. Isso tornou difícil ver o que cada proteína dentro das células faz em seu ambiente natural, apesar dos óbvios benefícios biomédicos de saber, por exemplo. como uma proteína específica reage quando as células são submetidas a estímulos químicos, como drogas farmacêuticas.
Para enfrentar este desafio, uma equipe da Universidade Metropolitana de Tóquio liderada pelo professor assistente Teppei Ikeya e pelo professor Yutaka Ito aplicou medições de espectroscopia de ressonância magnética nuclear (NMR) a proteínas específicas expressas no interior sf9 células de inseto cultivadas, uma linhagem de células originalmente derivada de um tipo de larva de mariposa amplamente utilizada para a produção de proteínas. O trabalho pioneiro de RMN da equipe já havia conseguido elucidar estruturas de proteínas de alta resolução dentro de bactérias (não eucariotos). O problema de simplesmente aplicar as mesmas técnicas às proteínas em sf9 células era a concentração significativamente mais baixa de proteínas alvo e vida curta das células, tornando difícil coletar espectros de NMR multidimensionais de alta qualidade para espectroscopia de efeito nuclear Overhauser (NOESY), que daria informações precisas sobre como diferentes átomos estão espaçados dentro de moléculas individuais. Assim, eles combinaram um esquema de medição de NMR rápido baseado em amostragem esparsa com métodos computacionais de última geração que empregam técnicas estatísticas como inferência Bayesiana, métodos adaptados para elucidar estruturas de proteínas de forma eficiente com base em uma quantidade limitada de informações estruturais de espectros de NMR na célula com sensibilidade inerentemente baixa. Um sistema de biorreator também foi equipado dentro do aparelho de NMR que manteve as células em um estado saudável durante as medições.
Com esses novos dados, a equipe foi capaz de elucidar a estrutura 3-D de três proteínas modelo com alta resolução sem precedentes, com uma precisão de 0,5 Angstroms (0,05 nanômetros) para a posição dos átomos da cadeia principal da proteína. Em particular, eles identificaram uma conformação significativamente diferente em uma região localizada de uma das proteínas em comparação com sua estrutura de referência em solução diluída. A diferença conformacional entre as proteínas "nas células" e "nos tubos de ensaio" foi provavelmente causada por interações não específicas com outras moléculas dentro das células. Está se tornando claro que essas interações contribuem para as funções biológicas das proteínas:espera-se que a capacidade de localizar e quantificar as mudanças estruturais das proteínas em um ambiente intracelular tenha um impacto significativo na pesquisa biomédica, tornando possível ver como diferentes condições, por ex. doenças neurodegenerativas afetam as conformações de proteínas in situ, e avaliar quantitativamente como os tratamentos afetam as anomalias estruturais.