Pesquisadores desvendam a estrutura cristalina de uma enzima chave do SARS-CoV-2, abrindo caminho para novos antivirais
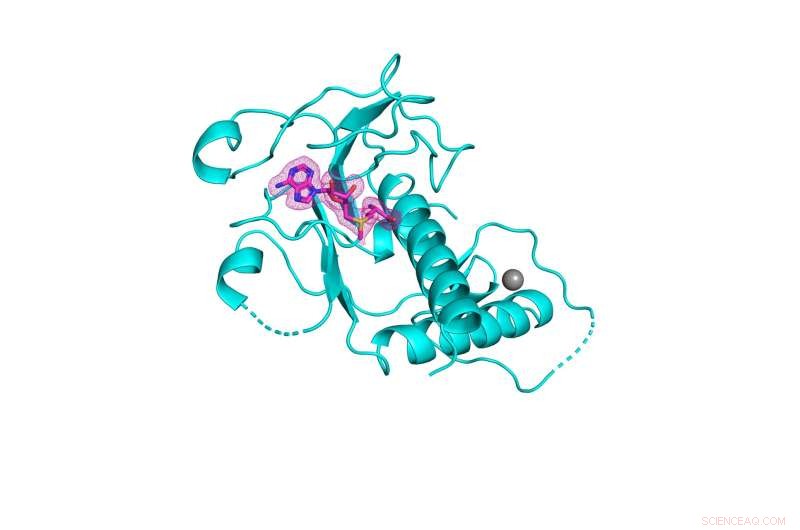
Estrutura 3-D do domínio SARS-CoV-2 nsp14 metiltransferase (mostrado em ciano) ligado ao seu cofator natural S-adenosilmetionina (mostrado em malha rosa). Crédito:Kottur, et al; Natureza Biologia Estrutural e Molecular
Uma equipe de pesquisadores do Monte Sinai produziu uma estrutura cristalina de alta resolução de uma enzima essencial para a sobrevivência do SARS-CoV-2, o vírus que causa o COVID-19. A descoberta pode levar ao design de novos antivirais extremamente necessários para combater os coronavírus atuais e futuros.
A enzima, conhecida como nsp14, possui uma região crucialmente importante conhecida como domínio RNA metiltransferase, que iludiu tentativas anteriores da comunidade científica de caracterizar sua estrutura cristalina tridimensional. Um artigo descrevendo o processo inovador foi publicado na edição online de 8 de setembro da
Nature Structural &Molecular Biology .
"Ser capaz de visualizar a forma do domínio metiltransferase de nsp14 em alta resolução nos dá insights sobre como projetar pequenas moléculas que se encaixam em seu sítio ativo e, assim, inibem sua química essencial", diz o autor sênior Aneel Aggarwal, Ph.D. , Professor de Ciências Farmacológicas da Escola de Medicina Icahn no Monte Sinai. “Com essas informações estruturais e em colaboração com químicos medicinais e virologistas, agora podemos projetar inibidores de pequenas moléculas para adicionar à família de antivirais que andam de mãos dadas com vacinas para combater o SARS-CoV-2”.
Os antivirais prescritos que visam as principais enzimas do SARS-CoV-2 incluem nirmatrelvir para a principal protease (M
Pro
) e molnupiravir e remdesivir para a enzima RNA polimerase (nsp12). A pesquisa para desenvolver novos antivirais visando diferentes atividades enzimáticas vem se acelerando em laboratórios ao redor do mundo, e a descoberta do Monte Sinai contribuiu significativamente para esse esforço.
"Parte do que impulsiona nosso trabalho", diz o Dr. Aggarwal, "é o conhecimento adquirido com o tratamento do HIV - que você normalmente precisa de um coquetel de inibidores para obter o máximo impacto contra o vírus".
A equipe do Mount Sinai realmente desenvolveu três estruturas cristalinas de nsp14, cada uma com cofatores diferentes, a partir das quais identificaram o melhor andaime para o design de antivirais para inibir a atividade da RNA metiltransferase que a enzima permite e o vírus precisa para sobreviver. De acordo com seu esquema, o antiviral substituiria o cofator natural S-adenosilmetionina, impedindo assim a ocorrência da química da metiltransferase. As estruturas cristalinas que a equipe elucidou foram disponibilizadas ao público e agora servirão como guias para bioquímicos e virologistas de todo o mundo para projetar esses compostos.
Tornar a descoberta possível foi a capacidade dos pesquisadores de superar um obstáculo que impedia outros no passado de criar cristais tridimensionais do domínio nsp14 metiltransferase. "Nós empregamos uma abordagem conhecida como cristalização assistida por fusão", explica o principal autor Jithesh Kottur, Ph.D., pós-doutorado em Icahn Mount Sinai, e um cristalógrafo e bioquímico. "Envolve a fusão da enzima com outra pequena proteína que a ajuda a cristalizar."
Dr. Aggarwal, um biólogo estrutural reconhecido internacionalmente, ressalta a importância do trabalho de investigação em andamento por pesquisadores em seu campo contra um vírus que levou a milhões de mortes em todo o mundo. “O vírus evolui tão rápido que pode desenvolver resistência aos antivirais agora disponíveis, por isso precisamos continuar desenvolvendo novos”, observa. “Devido à alta conservação de sequência do nsp14 nos coronavírus e suas variantes (o que significa que não sofre muitas mutações), nosso estudo ajudará no design de antivirais de amplo espectro para surtos de coronavírus presentes e futuros”.
+ Explorar mais Novo estudo descobre a estrutura tridimensional da máquina de replicação do genoma
