Os vírus reconectam a maquinaria celular do hospedeiro para maximizar a produção viral
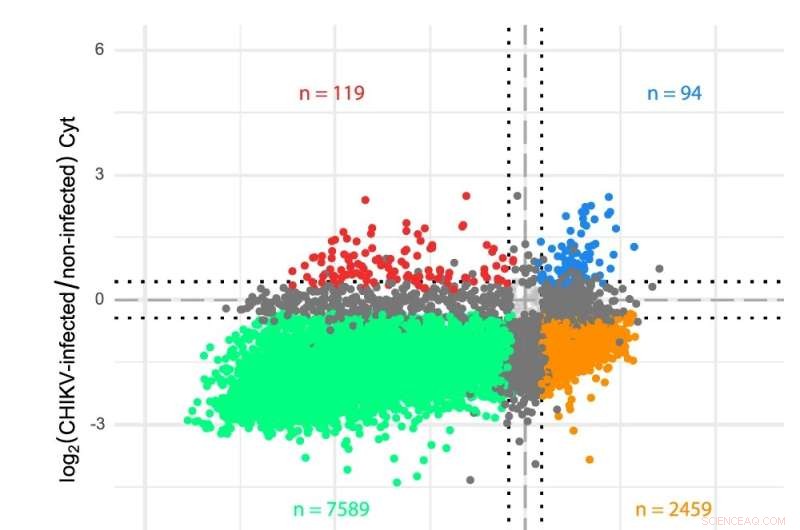
um Esquema de genoma de RNA de CHIKV. b Análise do transcriptoma dos compartimentos do citosol (Cyt) e ER em células infectadas com CHIKV. Os genes foram plotados de acordo com suas alterações log2 vezes em células infectadas com CHIKV versus células não infectadas para ambos os compartimentos de tradução. As leituras de sequenciamento foram analisadas usando uma anotação de transcriptoma personalizada e a expressão diferencial foi determinada via limma-voom (consulte Métodos). As cores indicam genes que são significativamente (FDR < 0,05) regulados negativamente em ambos os compartimentos (verde), regulados positivamente em ambos os compartimentos (azul), regulados para cima ou para baixo em apenas um compartimento (cinza escuro) ou mostram comportamento oposto em os dois compartimentos (laranja e vermelho). As linhas pontilhadas indicam alterações log2 vezes de ±0,433 (ou seja, uma alteração de 35% em qualquer direção) usadas como limite mínimo para declarar os níveis de mRNA alterados. As células HEK 293T foram infectadas em um MOI de 4 e as amostras coletadas doze horas após a infecção. Três réplicas independentes foram realizadas para cada condição. Crédito:Comunicação da Natureza DOI:10.1038/s41467-022-31835-x
O Grupo de Pesquisa em Virologia Molecular da Universidade Pompeu Fabra (UPF), em colaboração com o grupo Epitranscriptômica e Dinâmica de RNA do Centro de Regulação Genômica (CRG), descobriu um novo mecanismo pelo qual os vírus modificam a maquinaria celular para ler melhor as instruções no genoma do vírus invasor e, assim, produzir grandes quantidades de progênie viral. O estudo foi publicado em
Nature Communications e foi liderado por Juana Díez.
Os genes contêm as informações necessárias para a formação de proteínas, moléculas complexas essenciais à vida, formadas a partir de aminoácidos. A leitura dessas informações ocorre em duas etapas principais, sendo a primeira a transcrição, na qual a informação do gene (DNA) é transferida para uma molécula chamada RNA mensageiro (mRNA). O mRNA consiste em um "texto" formado por tripletos de nucleotídeos (as letras GCT, CAT, etc.). Cada tripleto corresponde a um aminoácido. A segunda fase é a tradução, na qual uma molécula chamada RNA de transferência (tRNA) reconhece cada tripleto e atua como tradutora trazendo o aminoácido correspondente. As proteínas são construídas através deste processo.
Existem 61 códons e 20 aminoácidos, e muitos tripletos codificam o mesmo aminoácido. Cada organismo utiliza preferencialmente um desses trigêmeos (trigêmeo ótimo) porque possui uma concentração mais alta do tRNA que reconhece esse trigêmeo. Assim, quando o "texto" do mRNA é enriquecido em tripletos ótimos, as proteínas serão geradas de forma rápida e eficiente enquanto que quando são enriquecidas em tripletos não ótimos, a eficiência da expressão diminuirá porque os tRNAs relacionados são escassos.
Os vírus são muito simples e, para se multiplicar e expressar suas proteínas, eles precisam sequestrar a maquinaria celular do hospedeiro. Os vírus geram seu próprio mRNA nas células que infectam, que lêem e geram proteínas virais para produzir mais vírus. Mas os mRNAs de muitos vírus, incluindo SARS-CoV-2 e vírus transmitidos pelos mosquitos dengue, zika e chikungunya, são enriquecidos em trigêmeos não ideais e ainda expressam proteínas virais com grande eficácia. “Para resolver esse dilema, usamos o vírus chikungunya como modelo porque seu genoma se multiplica em níveis extremamente altos”, explicam Jennifer Jungfleisch e René Böetcher, coautores do estudo.
“Nossas descobertas mostram pela primeira vez que os vírus modificam o tRNA do hospedeiro para adaptar o mecanismo de tradução do hospedeiro ao texto do mRNA viral”, diz Marc Talló, também coautor do artigo. “Em outras palavras, a infecção viral induz uma mudança de linguagem na célula, de modo que ela expresse as proteínas virais de forma muito eficiente. na célula infectada", acrescenta.
“Embora o estudo tenha se concentrado no vírus chikungunya, nossa proposta é que a modificação de tRNAs induzida pela infecção viral seja um mecanismo geral seguido por muitos vírus”, explica Juana Díez, professora titular do Departamento de Medicina e Ciências da Vida da UPF.
"Além disso, nossos resultados fornecem uma base para considerar a regulação do tRNA como um novo e promissor alvo terapêutico para o desenvolvimento de antivirais de amplo espectro que são eficazes contra vários vírus", conclui Díez. O estudo também envolveu o grupo de pesquisa coordenado por Eva María Novoa no CRG, e os outros autores são Gemma Pérez-Vilaró e Andres Merits (Instituto de Tecnologia da Universidade de Tartu).
+ Explorar mais Os vírus se adaptam à 'linguagem das células humanas' para sequestrar a síntese de proteínas
