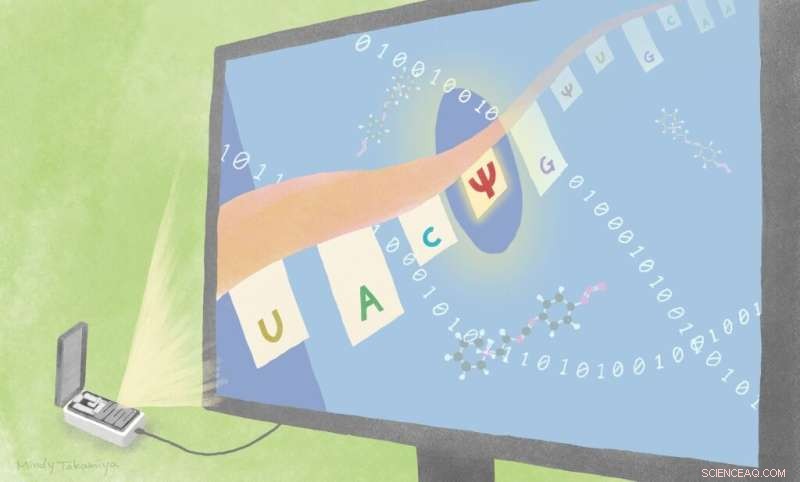
Os cientistas encontraram duas novas maneiras de identificar substituições de uracila para pseudouridina, combinando a tecnologia de sequenciamento existente com diferentes abordagens. Um é com cálculo baseado em algoritmo e um é com uma sonda química CMC. Crédito:Mindy Takamiya/Kyoto University iCeMS
Duas novas abordagens podem ajudar os cientistas a usar a tecnologia de sequenciamento existente para distinguir melhor as alterações de RNA que afetam a forma como o código genético é lido.
Os cientistas da Universidade de Kyoto estão cada vez mais próximos de encontrar maneiras de identificar alterações nas sequências de RNA que afetam a formação de proteínas e podem causar doenças. Sua abordagem, publicada na revista
Genomics , utiliza algoritmos de probabilidade juntamente com um dispositivo de sequenciamento de bolso já disponível.
"As modificações que são encontradas em todos os tipos de RNA biológico influenciam a regulação do gene, que finalmente decide como as diferentes células funcionam em nosso corpo", explica Ganesh Pandian Namasivayam, do Instituto de Ciência Integrada de Materiais Celulares (WPI-iCeMS) da Universidade de Kyoto. "Anormalidades nessas modificações podem levar a doenças graves, como diabetes, distúrbios do neurodesenvolvimento e câncer. Saber como e onde estão essas modificações de RNA é de primordial importância do ponto de vista clínico", acrescenta Soundhar Ramaswamy, o primeiro autor do estudo.
Já existem formas de identificar modificações de RNA, mas são insuficientes. Abordagens biofísicas como cromatografia e espectrometria de massa só podem processar pequenas quantidades de RNA de cada vez. Métodos de sequenciamento de alto rendimento, que podem processar grandes quantidades de RNA, envolvem preparação de amostra trabalhosa, não podem mapear simultaneamente várias modificações e são propensos a erros.
Namasivayam, Hiroshi Sugiyama e colegas da Universidade de Kyoto testaram e encontraram duas abordagens que podem distinguir com relativa sucesso uma modificação de RNA bem conhecida e abundante envolvendo a substituição da base nucleotídica uracila por outra chamada pseudouridina.
Semelhante ao DNA, o RNA é formado por uma fita de combinações variadas de quatro bases nucleotídicas diferentes:uracila, citosina, adenina e guanina. Como essas bases são organizadas determina o código que sinaliza qual proteína deve ser produzida. Quando a pseudouridina substitui a uracila no esqueleto do RNA, pode levar ao aumento da produção de proteínas ou à alteração do código de um que sinaliza a interrupção da tradução da informação para um que sinaliza a formação de aminoácidos.
A abordagem da equipe envolve o uso de uma plataforma de sequenciamento direto de RNA já disponível desenvolvida pela Oxford Nanopore Technologies. Nessa plataforma, as fitas de RNA passam por minúsculos poros de uma membrana. As interrupções são causadas na corrente que se move através da membrana, dependendo da ordem das diferentes bases de RNA. Isso permite que os cientistas "leiam" a sequência. Mas os cientistas que usam essa abordagem geralmente acham difícil distinguir diferentes tipos de modificações umas das outras.
Shubham Mishra, um dos primeiros autores deste estudo, desenvolveu algoritmos para identificar uma alta probabilidade de existência de uma substituição de pseudouridina em comparação com a possibilidade de que fosse um tipo diferente de mudança de base.
Uma de suas estratégias compara corridas curtas de RNA de cinco bases de nucleotídeos nas quais uracila, pseudouridina ou citosina estão cercadas de ambos os lados pelas mesmas bases. As leituras passam então por algoritmos que calculam a probabilidade da base do meio ser uma das três. Eles usaram sua estratégia, chamada Indo-Compare (Indo-C), em sequências de RNA projetadas e depois em levedura e RNA humano e descobriram que era bom para distinguir as substituições de pseudouridina das outras.
Eles também foram capazes de identificar substituições de pseudouridina misturando uma sonda química com amostras de RNA, que então se liga seletivamente a elas. Isso alterou as leituras de sequência de uma maneira que identifica a modificação.
"Acreditamos que nosso trabalho tornará os métodos baseados em sequenciamento de nanoporos menos trabalhosos para detectar modificações de RNA e mais capazes de caracterizar os impactos dessas modificações no desenvolvimento e na doença", diz Namasivayam.
A equipe em seguida visa otimizar o uso de ambas as abordagens em conjunto para identificar com mais precisão as modificações de RNA e DNA. Isso envolverá a fabricação de novas sondas químicas que correspondem a mudanças específicas. Eles também planejam desenvolver algoritmos avançados de aprendizado de máquina que complementam as abordagens de sequenciamento direto de RNA baseadas em sondas químicas.
+ Explorar mais Sequenciando várias modificações de base de RNA simultaneamente:uma nova era da biologia do RNA
