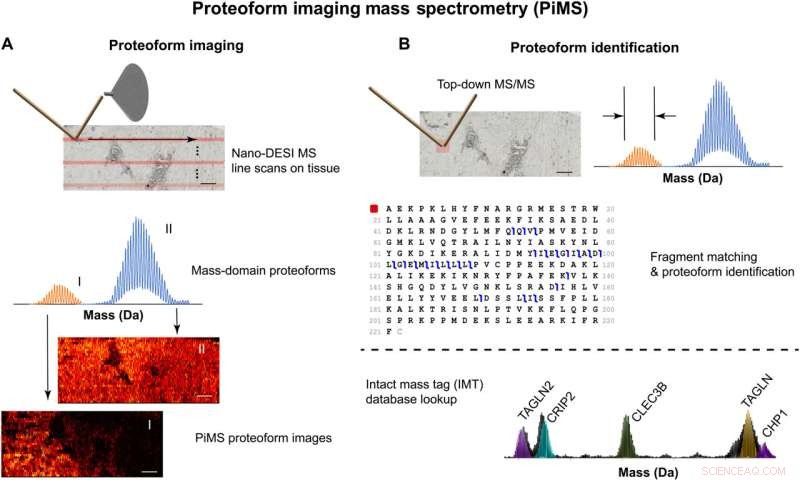
Ilustração do fluxo de trabalho do PiMS para geração de imagens e identificação de proteoformas. (A) Abordagem de varredura (topo), detecção de proteoformas no domínio de massa (meio) e reconstrução de imagem (abaixo). (B) Duas abordagens para identificar proteoformas usando fragmentação direta de íons proteoformes e leitura espectral por íon individual MS/MS (topo) ou pesquisa de banco de dados de valores de massa precisos (IMT, inferior). Barras de escala, 1 mm. Crédito:Avanços Científicos (2022). DOI:10.1126/sciadv.abp9929
Investigadores liderados por Neil Kelleher, Ph.D., professor de Medicina na Divisão de Hematologia e Oncologia e de Bioquímica e Genética Molecular, desenvolveram uma nova técnica de imagem que aumenta a detecção de proteoformas intactas em quatro vezes quando comparado aos atuais métodos de imagem de proteínas .
A técnica de imagem, detalhada em um artigo recente publicado em
Science Advances , fornece imagens de alta resolução e alto rendimento de proteoformas ou todas as versões modificadas de proteínas. É importante ressaltar que a técnica é "livre de rótulos", não requer anticorpos e pode identificar proteoformas inteiras diretamente de qualquer tecido não fixado. Atualmente, a técnica pode detectar cerca de 1.000 proteoformas e localizar proteoformas com resolução espacial de 40 a 70 mícrons.
Várias técnicas são comumente usadas para imagens de proteínas em tecidos humanos, mas muito poucas são capazes de imagens de proteoformas. Aqueles que podem visualizar proteoformas inteiras o fazem separando a proteoforma do tecido e ionizando-os para espectrometria de massa. No entanto, essas técnicas oferecem baixa especificidade molecular.
Para resolver esse problema, a equipe de Kelleher desenvolveu espectrometria de massa de imagem de proteoform (PiMS). A técnica funciona extraindo proteoformas do tecido com nanogotículas, "pesando" as proteoformas extraídas para identificá-las e, em seguida, usando esses dados para construir imagens de proteoformas do tecido digitalizado.
"A verdadeira inovação do PiMS é que ele combina uma técnica robusta existente para extrair e ionizar proteoformas, nanoDESI, com uma tecnologia inovadora para espectrometria de massa de íons individuais que foi co-inventada pela Thermo Fisher Scientific e pela Northwestern Proteomics. Em comparação com as técnicas de detecção regulares, A espectrometria de massa de íons individuais oferece até 500 vezes mais sensibilidade e 20 vezes mais poder de resolução. Isso aumenta significativamente o poder da técnica, e o PiMS detecta proteoformas maiores e mais raras e estende muito os limites de cobertura do proteoma", disse Kelleher, que também é diretor do Proteomics Center of Excellence da Northwestern, do Robert H. Lurie Comprehensive Cancer Center's Proteomics Core Facility e do Instituto de Processos de Química da Vida da Northwestern.
Para demonstrar as capacidades do PiMS, a equipe de Kelleher usou a técnica para criar imagens de proteoformas de unidades funcionais do rim humano. Essas imagens revelaram localizações espaciais distintas de proteoformas de diferentes regiões anatômicas e unidades funcionais de tecidos como o córtex renal versus a medula.
O aumento da cobertura de proteoma do PiMS também abre as portas para aplicações mais amplas no mapeamento de tecidos moleculares, identificando novos biomarcadores e melhorando o diagnóstico de doenças, de acordo com Kelleher.
"Recentemente, houve um grande impulso em genômica e proteômica para a biologia unicelular:capturar melhor a heterogeneidade das doenças usando abordagens espaciais ou unicelulares que preservam os diversos sinais em vez de abordagens em massa que misturam todos os tipos de células e A abordagem espacial em particular adiciona uma precisão muito maior para imagens de proteínas e atualmente estamos pressionando para identificar milhares de proteoformas com resolução de célula única", disse Kelleher.
+ Explorar mais Projeto proteoform humano para mapear proteínas no corpo humano
