Etiquetas moleculares revelam como os lisossomos danificados são selecionados e marcados para eliminação
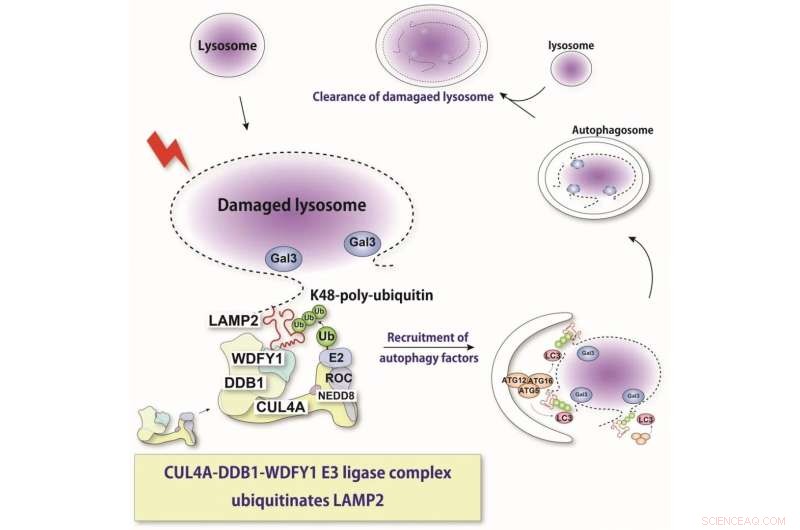
O lisossomo é uma organela intracelular ácida crucial para a degradação de vários componentes celulares. Após o dano da membrana lisossomal, as hidrolases lisossômicas são liberadas no citosol e induzem respostas ao estresse ou morte celular. Aqui, mostramos que um complexo proteico recém-identificado reconhece lisossomos danificados e ubiquitina um substrato, LAMP2. Este reconhecimento induz autofagia seletiva. Crédito:Keisuke Tabata
A autofagia é um processo de autodegradação que as células usam para remover componentes desnecessários ou danificados. Existem várias formas de autofagia, incluindo macroautofagia, que é um sistema de degradação em massa usado para direcionar materiais no citosol da célula para organelas chamadas lisossomos para degradação enzimática. No entanto, mesmo os próprios lisossomos às vezes precisam ser degradados. Recentemente, pesquisadores da Universidade de Osaka examinaram os detalhes moleculares específicos de como os lisossomos danificados são selecionados e marcados para eliminação.
Em um artigo recente publicado em
Cell Reports , a equipe descreveu um processo chamado lisofagia, a forma específica de autofagia seletiva responsável por limpar os lisossomos danificados. Estudos anteriores mostraram que substâncias como toxinas, lipídios e cristais de colesterol ou urato podem romper os lisossomos. Além de tornar a organela disfuncional, esse dano também pode induzir estresse oxidativo e inflamação que podem levar ao desenvolvimento de doenças. Portanto, a célula usa a lisofagia para resolver isso. No entanto, os mecanismos que controlam como as células podem reconhecer os lisossomos danificados e direcioná-los para degradação não são totalmente compreendidos.
"Sabemos de investigações anteriores que os lisossomos podem ser marcados por uma enzima específica, SCFFBXO27 através de um processo chamado poliubiquitinação", diz um dos principais autores, Hirofumi Teranishi. "A expressão de SCFFBXO27 só foi observada no cérebro e nos tecidos musculares, então levantamos a hipótese de que outra enzima mais onipresente deve existir para a lisofagia em outros tipos de células".
A equipe usou esferas de poliestireno revestidas com um reagente que poderia induzir danos endossomais e depois ser ubiquitinado. Eles então isolaram as esferas por meio de centrifugação e usaram um método chamado espectrometria de massa para identificar as proteínas associadas a elas, reduzindo a lista para 123 proteínas.
“Com a ajuda de técnicas moleculares onde poderíamos derrubar a expressão dessas várias proteínas, descobrimos que proteínas chamadas CUL4A, DDB1 e WDFY1 compõem um complexo que responde ao dano lisossomal”, explica Maho Hamasaki, autor sênior do estudo.
A caracterização posterior indicou que este complexo atua preferencialmente durante a lisofagia e facilita a adição das moléculas de ubiquitina. A proteína WDFY1 é necessária para reconhecer especificamente os lisossomos danificados.
"Nós então nos perguntamos qual parte do lisossomo é reconhecida por esse complexo proteico", diz Teranishi. "Várias proteínas lisossômicas foram examinadas, até que descobrimos que LAMP2 é a que é ubiquitinada pelo complexo CUL4A".
A equipe também descobriu que a presença de LAMP2 e sua interação com WDFY1 são essenciais para iniciar o processo de lisofagia. No geral, essas descobertas fornecem informações críticas sobre os mecanismos moleculares que são centrais para a lisofagia. Isso também pode ajudar no combate a doenças em que esse processo está desregulado. No futuro, os pesquisadores planejam determinar detalhes mais precisos sobre como o complexo CUL4A reconhece o LAMP2.
+ Explorar mais Via PITT:Cientistas descobrem como as células reparam o 'sistema de reciclagem' que promove a longevidade
