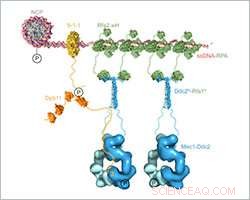
Modelo estrutural composto em escala da montagem e ativação Mec1-Ddc2-RPA em sítios de dano ao DNA. Crédito:Friedrich Miescher Institute for Biomedical Research
Ishan Deshpande e seus colegas do grupo de Susan Gasser e o Protein Structure Facility no FMI elucidaram o mecanismo de como Mec1-Ddc2, um supressor de tumor envolvido na resposta ao dano ao DNA se reúne nos locais de dano ao DNA. Eles se concentram na interação Ddc2-RPA, que traz o complexo da quinase Mec1-Ddc2 para o DNA de fita simples em locais de dano ao DNA. Com seus dados estruturais, os cientistas do FMI fornecem o elo que faltava entre duas estruturas publicadas anteriormente, permitindo-lhes montar um modelo estrutural composto em escala de Mec1-Ddc2 em ssDNA-RPA em sítios de dano de DNA.
A cinase ATR atraiu o interesse por mais de 20 anos. Esta quinase é uma das duas quinases centrais de detecção de danos ao DNA em células de mamíferos, e também é um supressor de tumor. Dois compostos que inibem a quinase ATR estão em ensaios clínicos para o tratamento de leucemia e tumores sólidos. Ainda, como essa enzima é regulada em um nível molecular permanece indescritível.
Ishan Deshpande, um estudante de doutorado na instalação de estrutura de proteínas e o grupo de Susan Gasser no Instituto Friedrich Miescher de Pesquisa Biomédica (FMI), já elucidou o mecanismo de como o homólogo de levedura do ATR, Mec1, se reúne em locais de dano ao DNA para ativação específica do local.
Em fermento, como em humanos, Mec1 forma um complexo com uma subunidade reguladora chamada Ddc2 (ATRIP em humanos). Após o dano ao DNA, grandes extensões de DNA de fita simples (ssDNA) são expostas, que são rapidamente cobertos pela proteína de replicação A (RPA). Mec1-Ddc2 reconhece esses alongamentos de ssDNA-RPA e se acumula nesses locais para iniciar os mecanismos de reparo.
Deshpande e colegas resolveram agora a estrutura 3-D do terminal N Ddc2 em associação com parte do RPA, fornecendo, assim, dados estruturais na ligação entre RPA e Mec1, que até agora tinha sido evasivo. Obtivemos uma estrutura de cocristal de alta resolução do terminal N Ddc2 juntamente com uma subunidade de RPA, Deshpande explicou. Com esses dados, poderíamos mostrar que Ddc2 forma homodímeros e liga RPA por seu N-terminal. Interessantemente, nossa estrutura Ddc2-RPA é o elo que faltava entre duas estruturas publicadas anteriormente, e isso nos permite montar um modelo estrutural composto em escala de Mec1-Ddc2 em ssDNA-RPA em sítios de dano de DNA.
O terminal N Ddc2 não só facilita a interação de Mec1 com RPA, mas também funciona como um espaçador para a quinase Mec1. Deshpande explica:O terminal N alongado Ddc2 permite que o grande módulo da quinase Mec1 se mova sem encontrar o DNA danificado nem a maquinaria de reparo trabalhando na lesão. Além disso, o espaçador Ddc2 pode permitir que Mec1 fosforile múltiplos substratos espacialmente distintos, enquanto permanece ligado ao local do dano ao DNA. Você pode pensar no terminal N Ddc2 como o pescoço de uma girafa que permite que a girafa alcance a grama no chão, bem como as folhas de uma árvore alta.
A atividade Mec1-Ddc2 adequada é importante para todos os tipos de reparo de danos ao DNA. Contudo, os dados funcionais de Deshpande e colegas indicam que o terminal N Ddc2 é particularmente importante para a sobrevivência da célula após dano ao DNA por luz ultravioleta. Nosso modelo sugere que o mero recrutamento de Mec1-Ddc2 para ssDNA pode contribuir para sua homodimerização e, portanto, ativação do complexo Mec1-Ddc2, explica Deshpande.
Além disso, Susan Gasser aponta "que esta tem sido uma colaboração maravilhosa entre o meu laboratório e as instalações de estrutura do FMI, permitindo-nos fazer uma ponte entre a estrutura e a função. "