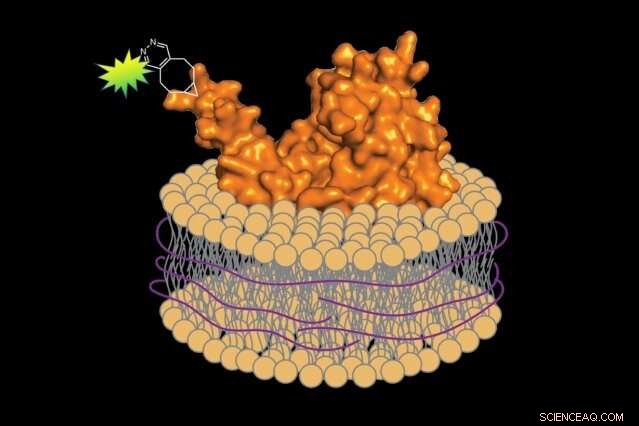
Usando um método sem detergente, os biólogos podem preparar proteínas marcadas com fluorescência junto com um pequeno segmento da membrana celular associada, preservando o ambiente nativo da proteína. Crédito:Jean-Marie Swiecicki
Todas as células têm uma membrana lipídica que envolve seus componentes internos - formando uma barreira protetora para controlar o que entra e o que fica fora. As proteínas embutidas nessas membranas são essenciais para a vida; eles ajudam a facilitar o transporte de nutrientes, conversão e armazenamento de energia, e comunicação celular. Eles também são importantes nas doenças humanas, e representam cerca de 60 por cento dos alvos de medicamentos aprovados. A fim de estudar essas proteínas de membrana fora da complexidade da célula, os pesquisadores devem usar detergente para remover a membrana e extraí-la. Contudo, determinar o melhor detergente para cada proteína pode envolver muitas tentativas e erros. E, remover uma proteína de seu ambiente natural corre o risco de desestabilizar a estrutura dobrada e interromper a função.
Em um estudo publicado em 9 de dezembro em Biologia Química Celular , cientistas do MIT desenvolveram uma maneira rápida e generalizável de extrair, purificar, e rotular proteínas de membrana para geração de imagens sem nenhum detergente - trazendo uma parte da membrana circundante para proteger a proteína e simular seu ambiente natural. Sua abordagem combina técnicas químicas e bioquímicas bem estabelecidas de uma nova maneira, isolar com eficiência a proteína para que ela possa ser marcada com fluorescência e examinada ao microscópio.
"Eu sempre brinco que não é muito natural estudar proteínas em sabonetes, "diz a autora sênior Barbara Imperiali, professor de biologia e química. "Nós criamos um fluxo de trabalho que permite que as proteínas da membrana sejam visualizadas enquanto mantém suas identidades e interações nativas. Esperamos que agora menos pessoas evitem estudar as proteínas da membrana, dada a sua importância em muitos processos fisiológicos. "
Como membro do laboratório Imperiali, O ex-pós-doutorando e autor principal Jean-Marie Swiecicki investigou as proteínas de membrana do patógeno de origem alimentar Campylobacter jejuni. Neste estudo, Swiecicki focou em PglC e PglA, duas proteínas de membrana que desempenham um papel em permitir que as bactérias infectem células humanas. Seus experimentos exigiam a marcação de PglC e PglA com etiquetas fluorescentes para rastreá-los. Contudo, ele não estava satisfeito com os métodos existentes para fazer isso.
Em alguns casos, os marcadores fluorescentes que devem ser incorporados à proteína para visualizá-la são muito grandes para serem colocados em posições definidas. Em outros casos, essas marcas não brilham o suficiente, ou interferir com a estrutura e função da proteína.
Para evitar esses problemas, Swiecicki decidiu usar um método conhecido como "mutagênese não natural de aminoácidos". Aminoácidos são as unidades que compõem a proteína, e a mutagênese não natural de aminoácidos envolve a adição de um novo aminoácido contendo um grupo químico projetado dentro da sequência da proteína. Este grupo químico pode então ser rotulado com uma etiqueta brilhante.
Swiecicki inseriu o código genético para as proteínas da membrana de C. jejuni em uma bactéria diferente, Escherichia coli. Dentro de E. coli, ele poderia incorporar o aminoácido não natural, que pode ser quimicamente modificado para adicionar o marcador fluorescente.
Quando chegou a hora de remover as proteínas da membrana, ele substituiu o detergente por uma substância diferente:um polímero de ácido estireno-maleico (SMA). Ao contrário do detergente, SMA envolve a proteína extraída e um pequeno segmento da membrana associada em uma capa protetora, preservando seu ambiente nativo. Imperiali explica, "É como um lenço protegendo seu pescoço do frio."
Swiecicki poderia então monitorar as proteínas brilhantes sob um microscópio para verificar se sua técnica era seletiva o suficiente para isolar proteínas de membrana individuais. Todo o processo, ele diz, leva apenas alguns dias, e geralmente é muito mais rápido e confiável do que os métodos de extração à base de detergente, o que pode levar meses e requer a experiência de bioquímicos altamente treinados para otimizar.
"Eu não diria que é uma fórmula mágica que vai funcionar para cada proteína, "ele diz." Mas é uma ferramenta altamente eficiente que poderia tornar mais fácil estudar muitos tipos diferentes de proteínas de membrana. " ele diz, pode até ajudar a facilitar triagens de drogas de alto rendimento.
"Como alguém que trabalha com complexos de proteínas de membrana, Posso atestar a grande necessidade de melhores métodos para estudá-los, "diz Suzanne Walker, um professor de microbiologia da Harvard Medical School que não estava envolvido no estudo. Ela espera estender a abordagem descrita no artigo para os complexos de proteínas que ela investiga em seu próprio laboratório. "Apreciei os extensos detalhes incluídos no texto sobre como aplicar a estratégia com sucesso, " Ela adiciona.
As próximas etapas serão testar a técnica em proteínas de mamíferos, e isolar várias proteínas de uma vez no shell SMA para observar suas interações. E, claro, cada nova técnica merece um nome. "Ainda estamos trabalhando em uma sigla cativante, "Imperiali diz." Alguma ideia?
Esta história foi republicada por cortesia do MIT News (web.mit.edu/newsoffice/), um site popular que cobre notícias sobre pesquisas do MIT, inovação e ensino.