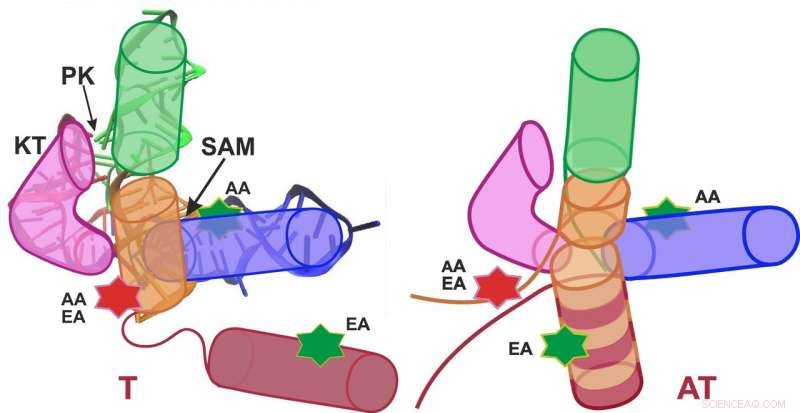
Representação esquemática do riboswitch SAM-I na conformação do terminador (desligar; esquerda) e na conformação do anti-terminador (ligar; direita). Crédito:Gráficos:APH / KIT
Muitas bactérias têm elementos de controle molecular por meio dos quais podem ligar e desligar genes. Esses riboswitches também abrem novas opções no desenvolvimento de antibióticos ou na detecção e decomposição de toxinas ambientais. Pesquisadores do Karlsruhe Institute of Technology (KIT), Universidade de Heidelberg, e Freie Universität Berlin agora usaram microscopia ótica de luz de moléculas individuais para estudar fundamentalmente a maneira como funcionam os riboswitches. Isso é relatado em Nature Chemical Biology .
Os riboswitches estão localizados no ácido ribonucléico mensageiro (mRNA) que transporta a informação genética para o local da biossíntese de proteínas. Um riboswitch consiste em um sensor que mede a concentração de uma pequena molécula metabólica e um efetor que controla a expressão do gene e, portanto, síntese de uma proteína. Como riboswitches existem em muitos patógenos bacterianos, eles representam alvos importantes no desenvolvimento de novos antibióticos. Outras aplicações são possíveis em biologia sintética. Por exemplo, bactérias podem ser geneticamente modificadas com riboswitches para detectar e decompor toxinas ambientais de baixo peso molecular, como herbicidas. Contudo, é necessária uma compreensão fundamental dos processos subjacentes à função dos riboswitches. O trabalho apresentado em Nature Chemical Biology é uma contribuição essencial a este respeito.
Os grupos de pesquisa do professor Gerd Ulrich Nienhaus do KIT e do professor Andres Jäschke da Universidade de Heidelberg estudaram o S-adenosil-L-metionina (SAM) -I riboswitch. "O anexo da molécula de SAM a este riboswitch causa a conformação, esse é o arranjo espacial dos átomos, mudar da estrutura anti-terminador (AT) para a estrutura terminadora (T), "Nienhaus explica." Como resultado, a expressão do gene está desligada. "
Primeiro, os cientistas em Heidelberg sintetizaram riboswitches SAM-I e os marcaram especificamente com dois corantes fluorescentes, cada um em pontos diferentes. Os pesquisadores do KIT então estudaram essas moléculas de RNA em alta resolução espacial e temporal usando microscópios de luz altamente sensíveis que medem a emissão fluorescente de moléculas de corante único. Por meio de experimentos de transferência de energia de ressonância de Förster (FRET), a dinâmica da conformação foi determinada diretamente. Para este propósito, a radiação laser é usada para fazer um corante verde emitir luz. Se um corante vermelho estiver localizado nas proximidades, pode assumir a energia de excitação do corante verde e emitir luz.
A probabilidade de transferência de energia depende fortemente da distância entre os corantes. As mudanças estruturais de uma molécula à qual os corantes estão especificamente ligados podem ser observadas diretamente por meio da emissão do corante vermelho. A emissão de luz é extremamente fraca, requer métodos complexos de análise de dados com base na modelagem de Markov oculta. A professora Bettina Keller, do Instituto de Química e Bioquímica da Freie Universität Berlin, desenvolveu os métodos especialmente para esse tipo de experimento, a fim de distinguir os sinais de emissão de luz dependentes do tempo do ruído.
Em sua análise, os pesquisadores distinguiram duas conformações (T e AT) do riboswitch SAM-I, e um total de quatro conformações (T1, T2, EM 1, e AT2). Surpreendentemente, o riboswitch não alternou totalmente entre as estruturas T e AT na presença e ausência de SAM, como era esperado, mas flutuou permanentemente entre todos os estados - apenas as ponderações foram alteradas. Um resultado importante para a função biológica foi que as flutuações estruturais observadas com um SAM anexado foram muito mais rápidas do que sem SAM. Como a sequência do riboswitch no RNA mensageiro está localizada diretamente na frente do gene a ser controlado, a molécula de RNA deve formar uma estrutura T (desligar) o mais rápido possível após a síntese na presença de SAM, a fim de evitar a transcrição subsequente do gene a ser controlado. A aceleração das flutuações da estrutura pelo anexo SAM garante, assim, a formação suficientemente rápida de uma estrutura em T. "Consequentemente, dinâmica do riboswitch SAM-I desempenha um papel importante para sua função, "Nienhaus diz." Esses insights detalhados sobre o funcionamento de uma biomolécula são os resultados de uma abordagem interdisciplinar da física, biotecnologia, e química teórica. "