
Projetos de computador (verde limão) são comparados com estruturas experimentais (roxo) em nível atômico, revelando a precisão atômica na estrutura geral (esquerda) e nas regiões de loop (direita). Crédito:Weizmann Institute of Science
A ideia de proteínas que podem ser projetadas em computadores para funções específicas tem sido um conceito de ponta que teimosamente permaneceu "no futuro". Novas pesquisas no Instituto de Ciência Weizmann podem trazer esse futuro um pouco mais perto. Voltando ao desenho da natureza - evolução - os cientistas criaram novas proteínas, com base em "partes naturais existentes, "que desempenham a função pretendida com louvor. Essas descobertas foram relatadas no Proceedings of the National Academy of Sciences ( PNAS )
A pesquisa foi conduzida pelo Dr. Sarel Fleishman e membros de seu grupo no Departamento de Ciências Biomoleculares, incluindo os estudantes de pesquisa Dror Baran, Maria Gabriele Pszolla, e Gideon Lapidoth. No laboratório do Dr. Fleishman, proteínas são projetadas com programas baseados em computador que lhes permitem gerar novas estruturas - por exemplo, anticorpos ou enzimas - que não existem na natureza. Se a equipe quiser uma proteína que realizará uma ação específica - digamos, se ligam a outra proteína ou realizam uma reação química - eles podem calcular, do começo ao fim, a sequência genética que alinhará os aminoácidos na ordem adequada e fará com que a proteína se enrole na forma tridimensional correta. Essas proteínas poderiam, em teoria, inaugurar uma nova era de medicamentos e catalisadores personalizados, mas os desafios do planejamento de grandes moléculas biológicas são imensos.
A equipe fez algumas perguntas simples:"O que uma enzima ou anticorpo natural tem que as proteínas artificiais não têm? Por que duas estruturas com composição semelhante são tão diferentes no que diz respeito à maneira como atuam dentro de um sistema biológico?"
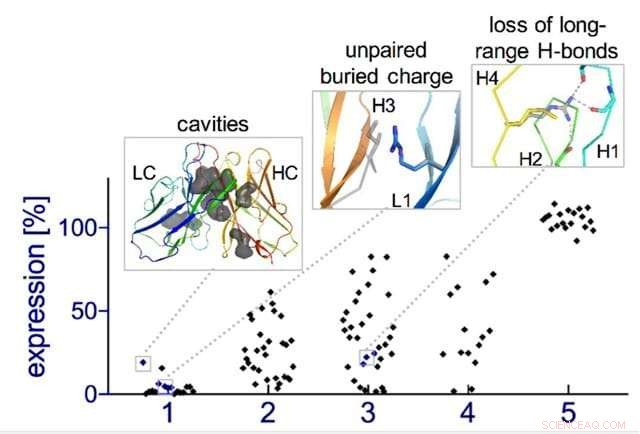
Aprendendo as regras do projeto de anticorpos:As falhas nos anticorpos direcionados à insulina (inserções) são tratadas por meio de cinco rodadas sucessivas de projeto-construção-teste de algoritmo “evolução, ”Elevando a fração de anticorpos projetados expressos de forma estável pelas células de cerca de 0% a 100%. Crédito:Weizmann Institute of Science
O grupo concentrou sua atenção em algumas partes dos anticorpos naturais ou enzimas que começam do zero e não entram nos projetos dos computadores; em particular, estruturas chamadas "loops, "que são inerentemente instáveis e" não ideais "e, Portanto, desafiador quando se trata de previsão computacional. Esses loops "não ideais" muitas vezes podem ser encontrados no centro das regiões ativas - aqueles que reconhecem um alvo ou se ligam ou clivam outra molécula.
Para incorporar esses elementos, os pesquisadores decidiram projetar um anticorpo funcional a partir de peças existentes, em vez de construir um do zero. Eles quebraram as estruturas encontradas nos anticorpos naturais em segmentos, incluindo os loops e outros recursos de suporte. Com efeito, os pesquisadores consertaram peças prontas, semelhante à forma como a evolução funciona.
A evolução natural é, claro, um processo muito lento:uma nova família de anticorpos pode levar dezenas de milhões de anos para se formar. Então, os pesquisadores voltaram ao processo de planejamento computadorizado, desta vez armados com seu novo insight. Os novos projetos foram testados experimentalmente em laboratório, algumas dezenas de novos anticorpos por vez. Inicialmente, os projetos tiveram um desempenho ruim, mas por meio de cinco ciclos de design-construção-teste, a equipe descobriu algumas regras gerais para o desenvolvimento de anticorpos. Em essência, eles criaram uma espécie de evolução simbiótica - os programas de design estavam evoluindo junto com os testes experimentais, cada um empurrando o outro para frente. Para demonstrar a viabilidade deste conceito, a equipe criou anticorpos artificiais que visam a insulina, caracterizando essas moléculas até a resolução de átomos individuais.
Em experiências futuras, os cientistas planejam projetar anticorpos artificiais modelados nos de camelos e lhamas. Por que esses animais em particular? Se um anticorpo humano, ou um de qualquer número de animais comuns, tem 200 aminoácidos, anticorpos em camelos e lhamas são feitos de apenas 100 - e ainda assim são estáveis e eficazes. Isso poderia tornar o projeto e a produção de anticorpos artificiais para condições humanas mais eficientes, e pode ter relevância para o desenho de novos diagnósticos e terapêuticas.