Descobrindo os principais atores no silenciamento de genes:insights sobre o crescimento das plantas e doenças humanas
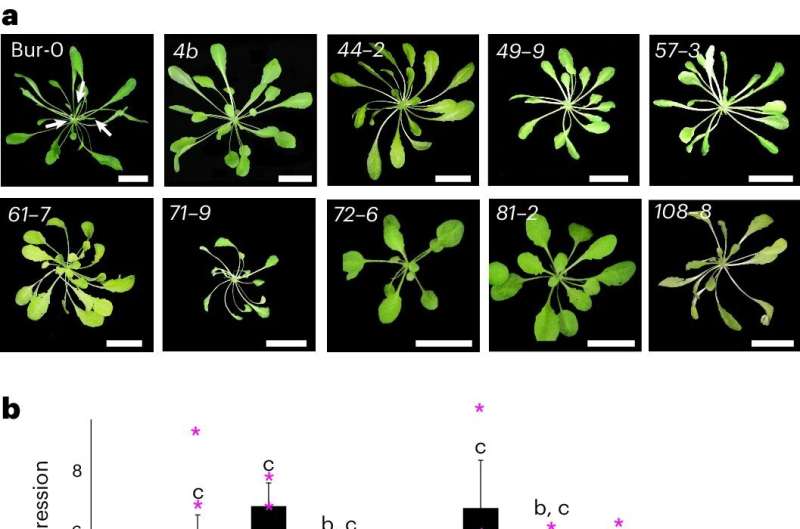 Uma triagem de supressor genético identifica RdDM como a principal via para o silenciamento epigenético induzido por expansão repetida. um , Fenótipos (denotados por seus identificadores de tela originais) dos supressores isolados em comparação com Bur-0. As folhas irregularmente prejudicadas são marcadas por setas brancas no tipo selvagem Bur-0. Barras de escala, 2 cm. b , Relativo IIL1 níveis de expressão em supressores genéticos identificados através da triagem genética. Os números representam os identificadores de tela originais e os genes correspondentes identificados após a clonagem são mostrados abaixo. Níveis médios de expressão baseados em três réplicas biológicas para cada linhagem (exceto Bur-0 e fug1 , onde n = 5 e 4, respectivamente) são mostrados. Asteriscos (*) indicam pontos de dados individuais. P os valores são baseados em uma análise de variância unidirecional com o teste post hoc de Tukey, e as linhas com letras diferentes são significativamente diferentes umas das outras (P < 0,05). Barras de erro representam sem. c , Um exemplo de análise SHOREmap com 44-2 identifica uma mutação em Pol V . Alelos de alta frequência (> 0,85) são coloridos em vermelho e cruzes vermelhas mostram os supostos alelos causais. Crédito:Plantas da Natureza (2024). DOI:10.1038/s41477-024-01672-5
Uma triagem de supressor genético identifica RdDM como a principal via para o silenciamento epigenético induzido por expansão repetida. um , Fenótipos (denotados por seus identificadores de tela originais) dos supressores isolados em comparação com Bur-0. As folhas irregularmente prejudicadas são marcadas por setas brancas no tipo selvagem Bur-0. Barras de escala, 2 cm. b , Relativo IIL1 níveis de expressão em supressores genéticos identificados através da triagem genética. Os números representam os identificadores de tela originais e os genes correspondentes identificados após a clonagem são mostrados abaixo. Níveis médios de expressão baseados em três réplicas biológicas para cada linhagem (exceto Bur-0 e fug1 , onde n = 5 e 4, respectivamente) são mostrados. Asteriscos (*) indicam pontos de dados individuais. P os valores são baseados em uma análise de variância unidirecional com o teste post hoc de Tukey, e as linhas com letras diferentes são significativamente diferentes umas das outras (P < 0,05). Barras de erro representam sem. c , Um exemplo de análise SHOREmap com 44-2 identifica uma mutação em Pol V . Alelos de alta frequência (> 0,85) são coloridos em vermelho e cruzes vermelhas mostram os supostos alelos causais. Crédito:Plantas da Natureza (2024). DOI:10.1038/s41477-024-01672-5
Biólogos da Monash University lançaram luz sobre os intricados mecanismos moleculares responsáveis pelo silenciamento genético induzido por repetições expandidas em um estudo internacional publicado hoje na revista Nature Plants .
