Estudo identifica nova via de topogênese para dobramento e montagem de proteínas de membrana multi-abrangentes
 Crédito:Célula Molecular (2024). DOI:10.1016/j.molcel.2024.04.010
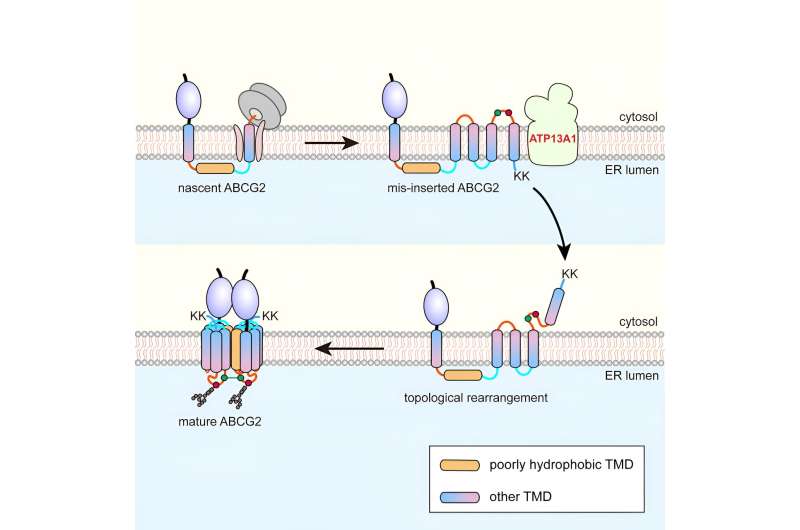
Crédito:Célula Molecular (2024). DOI:10.1016/j.molcel.2024.04.010 Pesquisadores liderados pelo professor Zhang Zairong, do Instituto de Química Orgânica de Xangai da Academia Chinesa de Ciências, identificaram uma via de topogênese pós-traducional para o dobramento e montagem de proteínas de membrana multi-abrangentes (MSPs).
Das aproximadamente 5.000 proteínas de membrana sintetizadas na membrana do retículo endoplasmático (ER) das células humanas, mais da metade são MSPs que desempenham papéis críticos na fisiologia celular e do organismo, funcionando como canais iônicos, transportadores, receptores e enzimas intramembrana.
Uma proporção significativa dessas funções depende de aminoácidos polares e carregados, levando à formação de DTMs pouco hidrofóbicas (pTMDs). No entanto, as pTMDs enfrentam desafios para serem reconhecidas e integradas na bicamada fosfolipídica pelo translocon Sec61, que prefere DTMs hidrofóbicas.
No proteoma humano, aproximadamente 30% das proteínas de membrana e mais de 50% das MSPs contêm pelo menos uma pTMD. Como esses pTMDs são efetivamente identificados e empacotados com precisão em estruturas MSP maduras tem sido uma questão científica importante.
Usando o transportador de cassete de ligação ao trifosfato de adenosina G2 (ABCG2) como modelo, os pesquisadores descobriram que durante a translocação co-traducional, o pTMD2 do ABCG2 passa através do poro central do translocon para o lúmen do RE, em vez de ser integrado em a bicamada fosfolipídica através da porta lateral do translocon.
Isto resulta na inserção de DTMs a jusante na membrana do RE com orientações reversas, formando assim um intermediário único. Após a tradução dos resíduos de lisina gêmeos carregados positivamente no terminal C, ocorre um processo de rearranjo topológico quase global.
A purificação por afinidade mostrou que o ATP13A1 pode detectar o sinal de carga positiva C-terminal do ABCG2. A substituição de resíduos de lisina por aminoácidos carregados negativamente ou neutros atenua significativamente as interações entre os mutantes ATP13A1 e ABCG2.
Além disso, o nocaute do ATP13A1 resultou no aparente acúmulo de conformações ABCG2 mal dobradas, principalmente aquelas com TMD6 desorientado dentro da membrana do RE. Assim, o ATP13A1 desempenha um papel crucial na topogênese das MSPs, onde sua atividade ATPase promove o deslocamento do TMD6 desorientado da bicamada lipídica para o citosol.
Posteriormente, o TMD6 citosólico é reintegrado na membrana do RE, conduzindo assim o rearranjo topológico pós-traducional de outras DTMs a montante.
Após o rearranjo bem-sucedido das DTMs 4-6, o intermediário pode oligomerizar em uma estrutura quaternária. É provável que este processo facilite a integração do pTMD2 na estrutura final do lúmen aquoso do RE e na estrutura madura, que é firmemente envolvida pelas DTMs circundantes.
Em resumo, o estudo, agora publicado na Molecular Cell , explica como um pTMD "difícil" é co-traducionalmente ignorado para inserção e pós-traducionalmente enterrado na estrutura final correta no estágio de dobramento tardio, evitando assim a exposição lipídica excessiva.
Notavelmente, devido à exposição do pTMD2 ao lúmen do RE durante a topogênese ABCG2, a modificação da glicosilação N441 causada pela mutação genética ABCG2-S441N pode bloquear significativamente a montagem do pTMD no estágio final da topogênese. Como ABCG2 é um transportador de ácido úrico, este estudo explica como esta mutação está intimamente associada a doenças humanas como gota e hiperuricemia.
