Avanços na pesquisa do genoma do arroz oferecem insights e aplicações promissoras para a agricultura
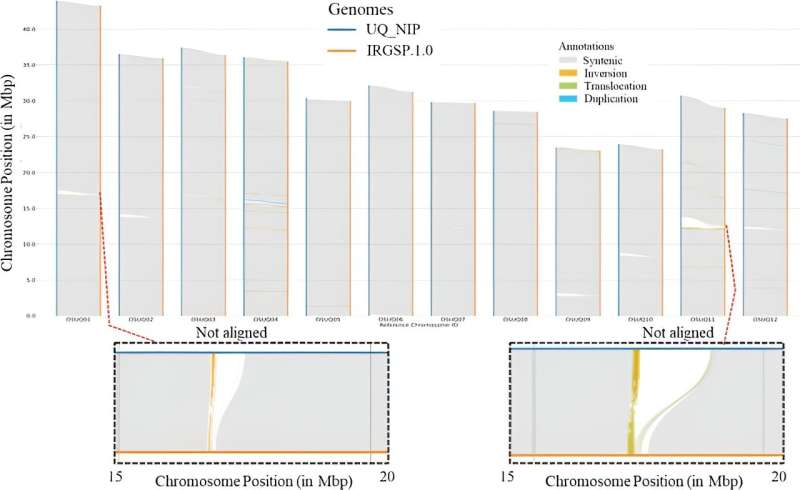 Colinearidade de sequência e variantes estruturais, incluindo inversões, translocações, duplicações e regiões não alinhadas, foram analisadas entre a montagem do genoma UQ Nipponbare e a montagem do genoma IRGSP-1.0-Nipponbare. Crédito:Plantas Tropicais (2024). DOI:10.48130/tp-0024-0007
Colinearidade de sequência e variantes estruturais, incluindo inversões, translocações, duplicações e regiões não alinhadas, foram analisadas entre a montagem do genoma UQ Nipponbare e a montagem do genoma IRGSP-1.0-Nipponbare. Crédito:Plantas Tropicais (2024). DOI:10.48130/tp-0024-0007 Uma equipe de pesquisa conseguiu uma melhoria na sequência do genoma resolvido por haplótipos do cultivar de arroz japonica Nipponbare. Este aprimoramento revela a identificação e anotação de mais de 3.000 novos genes, potencialmente oferecendo avanços significativos no melhoramento de culturas e estratégias de melhoramento.
A cultivar de arroz japonica Nipponbare tem sido fundamental como referência na genômica do arroz desde seu primeiro sequenciamento, há mais de duas décadas, marcando um avanço significativo na genômica de plantas. Apesar das melhorias contínuas na tecnologia de sequenciamento, a montagem do genoma Nipponbare ainda contém lacunas não resolvidas, principalmente devido à sequência repetitiva de DNA.
Os esforços contínuos e os avanços tecnológicos melhoraram a montagem do genoma em outras espécies de arroz e estenderam-se ao sequenciamento dos telômeros. No entanto, conseguir uma montagem totalmente resolvida por haplótipos continua a ser um problema não resolvido na investigação genómica do arroz, apresentando uma área crítica para estudos futuros.
Um estudo publicado em Tropical Plants em 3 de abril de 2024, gera um genoma de arroz resolvido com haplótipos melhorado para uma melhoria abrangente de telômero a telômero (T2T).
Neste estudo, leituras PacBio HiFi e leituras Hi-C foram utilizadas para gerar uma montagem contig com Hifiasm, resultando em uma montagem em fases de haplótipos. Este processo de montagem produziu contigs distintos para nove dos cromossomos. Em contraste, a montagem dos três cromossomos restantes envolveu dois contigs separados para cada um.
Os contigs montados foram então organizados hierarquicamente em 12 pseudo-cromossomos usando a ferramenta de andaime YaHS, culminando em uma montagem T2T que era maior e mais completa do que a referência anterior do IRGSP-1.0. Esta montagem refinada revelou a presença de 3.050 novos genes, com mais de 95% apoiados por evidências transcritas, indicando uma melhoria significativa na anotação e compreensão estrutural do genoma.
As descobertas destacam o imenso potencial das novas tecnologias de sequenciamento para expandir e refinar os dados genômicos, melhorando substancialmente a informação genética dos genomas estabelecidos. O genoma estendido e mais detalhado, cobrindo 99,3% dos genes universais de cópia única com um N50 de 30,7 Mb, fornece uma estrutura robusta para futuros estudos genéticos do arroz e programas de melhoramento.
A análise comparativa também iluminou variantes estruturais e regiões adicionais não alinhadas, enriquecendo a compreensão da arquitetura e funcionalidade genômica.
De acordo com o pesquisador principal do estudo, Robert J. Henry, “este genoma em fases será um recurso útil para a pesquisa do arroz”. Olhando para o futuro, esta equipa está focada na aplicação das suas técnicas avançadas de sequenciação e montagem a outras variedades de arroz e espécies estreitamente relacionadas.
Em resumo, este trabalho não só sublinha a rápida evolução da tecnologia genómica do arroz, mas também enfatiza a necessidade crítica de avanços contínuos para mapear com precisão genomas complexos, facilitando assim avanços significativos na genómica agrícola.
