A matemática permite que os cientistas entendam a organização dentro de um núcleo celular
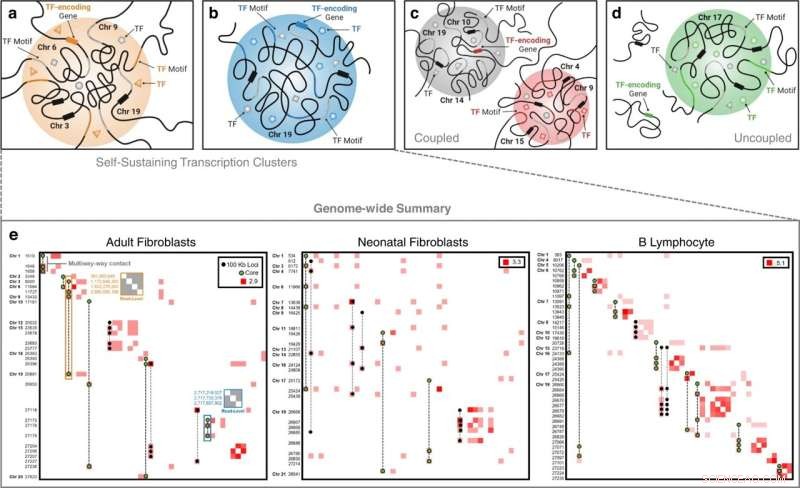
Classes de agrupamentos de transcrição. Em um cluster de transcrição auto-sustentável, um TF e o gene que codifica esse TF estão ambos presentes. Os exemplos inter e intracromossômicos em (a) e (b), respectivamente, ilustram esse fenômeno onde em a vemos o FT de interesse (triângulo laranja) circulando no cluster, seu motivo de ligação presente na cromatina (porção laranja) ), e seu gene correspondente expresso (retângulo laranja no cromossomo 6). As formas cinzas representam TFs adicionais com motivos de ligação (porção cinza da cromatina) no cluster. Retângulos pretos nos cromossomos 3, 9 e 19 representam genes adicionais presentes no cluster. c Uma classe independente de análogos de agrupamentos de transcrição onde observamos uma ligação de TF (quadrado vermelho) em um agrupamento de transcrição (agrupamento vermelho) e seu gene correspondente expresso em um agrupamento de transcrição separado (agrupamento cinza), mas não no mesmo agrupamento. d Uma classe independente de análogos de agrupamentos de transcrição onde observamos uma ligação de TF (círculo verde) em um agrupamento de transcrição (agrupamento verde) e seu gene correspondente expresso, mas não dentro de um agrupamento de transcrição. e Agrupamentos de transcrição auto-sustentáveis específicos do tipo de célula de todo o genoma extraídos de dados de contato multidirecional e decompostos em matrizes de contato Hi-C com resolução de 100 kb. As frequências de contato são transformadas em log para melhor visualização. Frequências ao longo da diagonal indicam interação entre dois ou mais loci multidirecional únicos que se enquadram no mesmo compartimento de 100 kb. Os rótulos de eixo são coordenadas bin não contíguas de 100 kb em ordem cromossômica. Os contatos multidirecionais que compõem os clusters de transcrição autossustentáveis são sobrepostos. Contatos multidirecionais com loci de cor verde representam clusters de transcrição 'core' - clusters de transcrição contendo um regulador mestre e seu análogo de gene. Um exemplo de mapa de contato de nível de leitura para o cluster de transcrição autossustentável FOXO3 intercromossômico é indicado pela caixa laranja destacada na matriz de contato de fibroblastos adultos e um mapa de contato de nível de leitura para o cluster de transcrição autossustentável ZNF320 intracromossômico é indicado pela caixa azul realçada. Os valores ao longo do eixo esquerdo dessas matrizes de contato de nível de leitura são posições de pares de bases dos loci de contato no genoma. Crédito:Comunicação da Natureza (2022). DOI:10.1038/s41467-022-32980-z
A terceira lei do escritor de ficção científica Arthur C. Clarke diz que "qualquer tecnologia suficientemente avançada é indistinguível da magia".
Indika Rajapakse, Ph.D., é uma crente. O engenheiro e matemático agora se vê biólogo. E ele acredita que a beleza de combinar essas três disciplinas é crucial para desvendar como as células funcionam.
Seu último desenvolvimento é uma nova técnica matemática para começar a entender como o núcleo de uma célula é organizado. A técnica, que Rajapakse e colaboradores testaram em vários tipos de células, revelou o que os pesquisadores chamaram de clusters de transcrição autossustentáveis, um subconjunto de proteínas que desempenham um papel fundamental na manutenção da identidade celular.
Eles esperam que esse entendimento exponha vulnerabilidades que podem ser direcionadas para reprogramar uma célula para interromper o câncer ou outras doenças.
"Cada vez mais biólogos de câncer pensam que a organização do genoma desempenha um grande papel na compreensão da divisão celular incontrolável e se podemos reprogramar uma célula cancerosa. Isso significa que precisamos entender mais detalhes sobre o que está acontecendo no núcleo", disse Rajapakse, professor associado de computação medicina e bioinformática, matemática e engenharia biomédica na Universidade de Michigan. Ele também é membro do U-M Rogel Cancer Center.
Rajapakse é o autor sênior do artigo, publicado na
Nature Communications . O projeto foi liderado por um trio de alunos de pós-graduação com uma equipe interdisciplinar de pesquisadores.
A equipe aprimorou uma tecnologia mais antiga para examinar a cromatina, chamada Hi-C, que mapeia quais partes do genoma estão próximas. Ele pode identificar translocações cromossômicas, como as que ocorrem em alguns tipos de câncer. Sua limitação, no entanto, é que ele vê apenas essas regiões genômicas adjacentes.
A nova tecnologia, chamada Pore-C, usa muito mais dados para visualizar como todas as peças dentro do núcleo de uma célula interagem. Os pesquisadores usaram uma técnica matemática chamada hipergrafos. Pense:diagrama de Venn tridimensional. Ele permite que os pesquisadores vejam não apenas pares de regiões genômicas que interagem, mas a totalidade das relações complexas e sobrepostas em todo o genoma dentro das células.
"Podemos entender essa relação multidimensional sem ambiguidade. Ela nos dá uma maneira mais detalhada de entender os princípios organizacionais dentro do núcleo. Se você entender isso, também poderá entender onde esses princípios organizacionais se desviam, como no câncer", disse Rajapakse. "É como juntar três mundos - tecnologia, matemática e biologia - para estudar mais detalhes dentro do núcleo."
Os pesquisadores testaram sua abordagem em fibroblastos neonatais, fibroblastos adultos biopsiados e linfócitos B. Eles identificaram organizações de agrupamentos de transcrição específicos para cada tipo de célula. Eles também encontraram o que chamaram de clusters de transcrição auto-sustentáveis, que servem como assinaturas transcricionais chave para um tipo de célula.
Rajapakse descreve isso como o primeiro passo em um quadro maior.
"Meu objetivo é construir esse tipo de imagem ao longo do ciclo celular para entender como uma célula passa por diferentes estágios. O câncer é uma divisão celular incontrolável", disse Rajapakse. “Se entendermos como uma célula normal muda ao longo do tempo, podemos começar a examinar sistemas controlados e não controlados e encontrar maneiras de reprogramar esse sistema”.
+ Explorar mais Desvendando os mistérios da estrutura do genoma no núcleo da célula humana usando uma simulação computacional 3D
