Nova ferramenta baseada em RNA pode iluminar circuitos cerebrais, editar células específicas
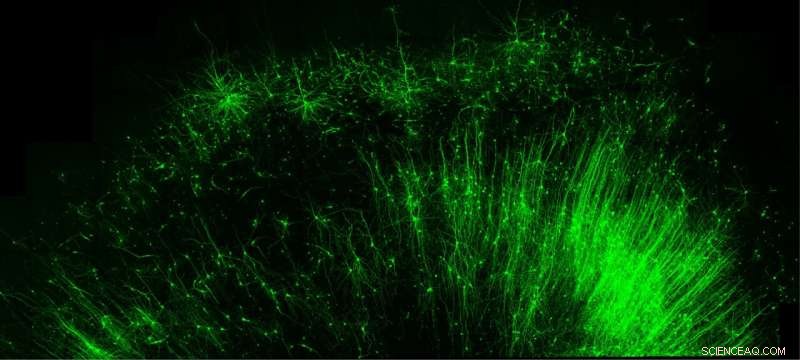
Marcar e iluminar apenas as células inibitórias de “freio” (verde) no tecido cerebral humano é apenas uma das muitas coisas que a nova ferramenta da Duke University, CellREADR, pode fazer. Crédito:Derek Southwell, Duke University
Pesquisadores da Duke University desenvolveram uma ferramenta de edição baseada em RNA que tem como alvo células individuais, em vez de genes. É capaz de atingir com precisão qualquer tipo de célula e adicionar seletivamente qualquer proteína de interesse.
Os pesquisadores disseram que a ferramenta pode permitir a modificação de células e funções celulares muito específicas para gerenciar doenças.
Usando uma sonda baseada em RNA, uma equipe liderada pelo neurobiólogo Z. Josh Huang, Ph.D. e pesquisador de pós-doutorado Yongjun Qian, Ph.D. demonstraram que podem introduzir etiquetas fluorescentes nas células para rotular tipos específicos de tecido cerebral; um botão liga/desliga sensível à luz para silenciar ou ativar neurônios de sua escolha; e até mesmo uma enzima de autodestruição para expurgar com precisão algumas células, mas não outras. A obra aparece em 5 de outubro em
Nature .
Seu sistema seletivo de monitoramento e controle de células depende da enzima ADAR, que é encontrada nas células de todos os animais. Embora estes sejam os primeiros dias para o CellREADR (acesso à célula através da detecção de RNA pelo Endogenous ADAR), as aplicações possíveis parecem ser infinitas, disse Huang, assim como seu potencial para funcionar em todo o reino animal.
"Estamos empolgados porque isso fornece uma tecnologia simplificada, escalável e generalizável para monitorar e manipular todos os tipos de células em qualquer animal", disse Huang. "Nós poderíamos realmente modificar tipos específicos de função celular para gerenciar doenças, independentemente de sua predisposição genética inicial", disse ele. "Isso não é possível com as terapias ou medicamentos atuais."
CellREADR é uma string personalizável de RNA composta de três seções principais:um sensor, um sinal de parada e um conjunto de plantas.
Primeiro, a equipe de pesquisa decide que tipo de célula específico deseja investigar e identifica um RNA alvo que é produzido exclusivamente por esse tipo de célula. A notável especificidade tecidual da ferramenta se baseia no fato de que cada tipo de célula fabrica RNA de assinatura não visto em outros tipos de células.
Uma sequência sensora é então projetada como a fita complementar do RNA alvo. Assim como os degraus do DNA são compostos de moléculas complementares que são inerentemente atraídas umas às outras, o RNA tem o mesmo potencial magnético para se ligar a outro pedaço de RNA se tiver moléculas correspondentes.
Depois que um sensor entra em uma célula e encontra sua sequência de RNA alvo, ambos os pedaços se juntam para criar um pedaço de RNA de fita dupla. Esse novo mashup de RNA aciona a enzima ADAR para inspecionar a nova criação e, em seguida, alterar um único nucleotídeo de seu código.
A enzima ADAR é um mecanismo de defesa celular projetado para editar o RNA de fita dupla quando ocorre, e acredita-se que seja encontrado em todas as células animais.
Sabendo disso, Qian projetou o sinal de parada do CellREADR usando as mesmas edições específicas do nucleotídeo ADAR no RNA de fita dupla. The stop sign, which prevents the protein blueprints from being built, is only removed once CellREADR's sensor docks to its target RNA sequence, making it highly specific for a given cell type.
Once ADAR removes the stop sign, the blueprints can be read by cellular machinery that builds the new protein inside the target cell.
In their paper, Huang and his team put CellREADR through its paces. "I remember two years ago when Yongjun built the first iteration of CellREADR and tested it in a mouse brain," Huang said. "To my amazement, it worked spectacularly on his first try."
The team's careful planning and design paid off as they were then able to demonstrate CellREADR accurately labelled specific brain cell populations in living mice, as well as effectively added activity monitors and control switches where directed. It also worked well in rats, and in human brain tissue collected from epilepsy surgeries.
"With CellREADR, we can pick and choose populations to study and really begin to investigate the full range of cell types present in the human brain," said co-author Derek Southwell, M.D., Ph.D., a neurosurgeon and assistant professor in the department of neurosurgery at Duke.
Southwell hopes CellREADR will improve his and others' understanding of the wiring diagram for human brain circuits and the cells within them, and in doing so, help advance new therapies for neurological disorders, such as a promising new method to treat drug-resistant epilepsy he is piloting.
Huang and Qian are especially hopeful about CellREADR's potential as a "programmable RNA medicine" to possibly cure diseases—since that's what drew them both to science in the first place. They have applied for a patent on the technology.
"When I majored in pharmacology as an undergraduate, I was very naïve," Qian said. "I thought you could do a lot of things, like cure cancer, but actually it's very difficult. However, now I think, yes maybe we can do it."
+ Explorar mais New technology identifies molecular properties of cells and maps their location within tissues
