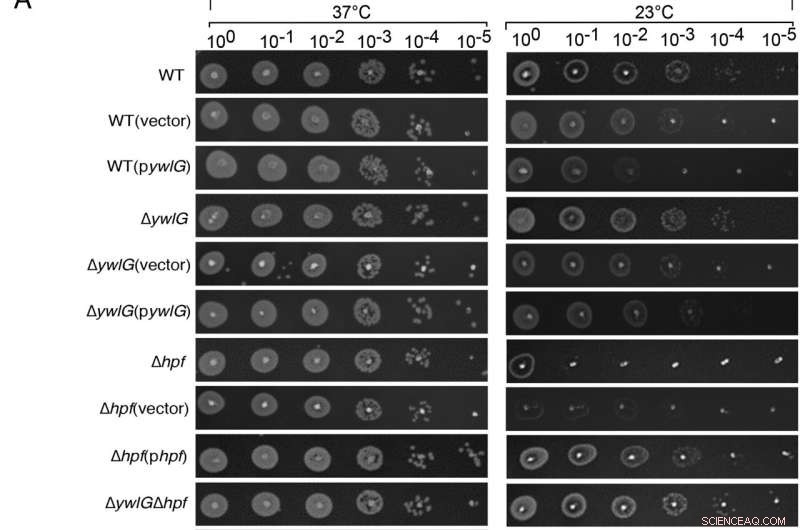
A inativação de ywlG suprime a sensibilidade ao frio de um mutante Δhpf-nulo de uma maneira dependente do meio. A complementação com ywlG e hpf codificados por plasmídeo confirmou a supressão mediada por YwlG e a resposta ao frio dependente de HPF. Ensaios de ponto de diluição em série foram realizados em (A) placas de ágar à base de caldo de lisogenia (LB) ou (B) à base de caldo de soja tríptica (TSB). Células de crescimento exponencial (OD600 =0,6) em TSB foram ajustados para OD600 =0,2 após duas lavagens em 1xPBS. Após diluições em série, 2 µL de cada diluição foram colocados nas placas de ágar. Os resultados foram registrados após 24 h e 48 h de incubação a 37 °C e 23 °C, respectivamente. As imagens são representativas de três experimentos independentes. Crédito:Proceedings of the National Academy of Sciences (2022). DOI:10.1073/pnas.2207257119
Pesquisadores da Northwestern Medicine descobriram novos mecanismos regulatórios que promovem a sobrevivência bacteriana, de acordo com as descobertas publicadas no
Proceedings of the National Academy of Sciences .
O estudo, liderado por M.-N. Frances Yap, Ph.D., professor associado de Microbiologia-Imunologia, também estabelece as bases para a identificação de novos alvos terapêuticos para tratar infecções bacterianas.
A hibernação de ribossomos é um mecanismo realizado por bactérias e outros organismos, incluindo eucariotos, para garantir a sobrevivência a longo prazo. Os ribossomos 100S hibernantes em bactérias consistem em dois complexos 70S unidos por uma proteína de fator promotor de hibernação (HPF).
Este complexo 100S serve a dois propósitos principais:prevenir a degradação da maquinaria de biossíntese de proteínas (ribossomas) e conservar energia na célula desligando a tradução de mRNA.
No estudo atual, a equipe de Yap utilizou espectrometria de massa para estudar as interações de proteínas em Staphylococcus aureus resistente à meticilina, uma bactéria comumente conhecida por causar infecções por estafilococos. A partir dessas análises, eles descobriram que o HPF interage com um parceiro de ligação anteriormente não identificado localizado fora do ribossomo, uma proteína chamada YwlG.
Além disso, ao resolver a estrutura atômica de YwlG, eles descobriram que essa proteína está envolvida na atividade da glutamato hidrogenase, que é uma via metabólica essencial necessária para a sobrevivência das bactérias.
"Em condições normais, uma fração da proteína YwlG está ligada ao HPF. Ao se ligar, o YwlG não pode estimular a atividade da glutamato hidrogenase e o HPF não pode se ligar ao ribossomo, então é um 'sequestro mútuo' das duas proteínas para garantir a quantidade certa de Complexos 100S e glutamato desidrogenase celular são produzidos", disse Yap.
Os achados apontam para HPF e YwlG como fatores que influenciam a colonização bacteriana e a gravidade da infecção. O objetivo final, de acordo com Yap, é identificar um composto que possa interromper a formação desses complexos e, por sua vez, inibir a sobrevivência a longo prazo das bactérias.
“Uma das principais vantagens é que a proteína semelhante ao HPF ou YwlG não existe em humanos, então você pode desenvolver um antibacteriano direcionado ao HPF ou YwlG que inibe o crescimento bacteriano, mas não prejudica os hospedeiros”, disse Yap.
+ Explorar mais Ribossomos em hibernação ajudam as bactérias a sobreviver
