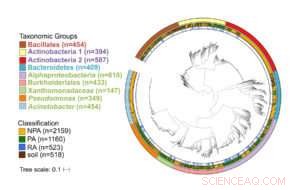
 p Árvore filogenética de mais de 3, 800 genomas bacterianos de alta qualidade e não redundantes. O anel externo denota o grupo taxonômico, anel central denota a fonte de isolamento, e o anel interno denota os genomas associados à raiz dentro dos genomas associados às plantas. Os nomes dos táxons são codificados por cores com base no filo:verde - Proteobactéria, vermelho - Firmicutes, azul –Bacteroidetes, roxo - Actinobactérias. Crédito:Asaf Levy
p Árvore filogenética de mais de 3, 800 genomas bacterianos de alta qualidade e não redundantes. O anel externo denota o grupo taxonômico, anel central denota a fonte de isolamento, e o anel interno denota os genomas associados à raiz dentro dos genomas associados às plantas. Os nomes dos táxons são codificados por cores com base no filo:verde - Proteobactéria, vermelho - Firmicutes, azul –Bacteroidetes, roxo - Actinobactérias. Crédito:Asaf Levy
p À medida que a população global aumenta, estimado em quase 10 bilhões em 2050, o mesmo acontece com a necessidade de aumentar o rendimento das safras e produzir material vegetal suficiente para alimentos e combustíveis alternativos sustentáveis. Para ajudar a melhorar as estratégias de cultivo e superar desafios, como tornar as plantas mais tolerantes às terras marginais, e estresses como seca e baixa disponibilidade de nutrientes, os pesquisadores estão se concentrando em compreender e promover relações benéficas entre planta e micróbio. p Publicado em 18 de dezembro, Edição de 2017 em
Nature Genetics , uma equipe liderada por pesquisadores do Joint Genome Institute (JGI) do Departamento de Energia dos EUA (DOE), um DOE Office of Science User Facility, e o Howard Hughes Medical Institute da Universidade da Carolina do Norte em Chapel Hill (UNC) exploraram um catálogo de genomas bacterianos para identificar e caracterizar genes candidatos que auxiliam as bactérias na adaptação a ambientes vegetais, especificamente genes envolvidos na colonização de raízes bacterianas.
p A maioria dos estudos de campo até o momento enfocou a estrutura da comunidade do microbioma da planta, ou seja, "quem está lá, "e menos na função, ou seja, "o que eles estão fazendo, como e quando estão fazendo isso. "Estudos anteriores que consideraram a função analisaram principalmente uma única interação hospedeiro-micróbio, como aquele entre uma planta Arabidopsis e um patógeno.
p "Se quisermos projetar o microbioma certo para apoiar o crescimento da planta, precisamos entender a função real do microbioma e não apenas genes marcadores de sequência, "disse o co-primeiro autor do estudo, Asaf Levy, um cientista pesquisador do JGI. "Aqui, usamos um grande esforço genômico e computacional para abordar a questão fundamental e importante:'Como o microbioma da planta interage com a planta?'"
p A maior parte da interação entre micróbios e plantas ocorre na interface entre as raízes e o solo. Pesquisadores da UNC, Oak Ridge National Lab, e o Instituto Max Planck isolou novas bactérias do ambiente de raiz de Brassicaceae (191), choupos (135), e milho (51). Os genomas desses 377 isolados bacterianos, mais um adicional de 107 células bacterianas únicas de raízes de A. thaliana, foram então sequenciados, montado, e anotado no JGI.
p Os autores então combinaram os novos genomas com milhares de genomas disponíveis publicamente que representam os principais grupos de bactérias associadas a plantas, e incluiu bactérias de vários ambientes vegetais e não vegetais, como o intestino humano, para comparação. O banco de dados resultante de 3837 genomas, 1160 dos quais são de plantas, foi usado em uma análise genômica comparativa.
p Os pesquisadores então identificaram genes que são enriquecidos nos genomas de organismos associados a plantas e raízes.
p "É muito importante para nós entendermos quais genes e funções os micróbios usam para colonizar as plantas, porque só então teremos a chance de desenvolver racionalmente 'probióticos de plantas' úteis para nos ajudar a cultivar mais safras de alimentos e energia com menos insumos químicos, como fertilizantes e pesticidas ou fungicidas, "disse o autor sênior do estudo, Jeff Dangl, um investigador do Howard Hughes Medical Institute e John N. Couch Professor de Biologia da University of North Carolina em Chapel Hill
p Um dos principais insights obtidos com o estudo foi que os genomas associados à planta e ao solo tendem a ser maiores do que os genomas de controle do mesmo clado. Verificou-se que isso se devia em parte ao enriquecimento de genes envolvidos no metabolismo e transporte do açúcar, provavelmente uma adaptação ao carbono vegetal derivado da fotossíntese, gerado pelas "fábricas de doces da natureza, "disse Asaf Levy. Até 20% do carbono fixado pelas plantas através da fotossíntese é exsudado pelas raízes como açúcares para atrair micróbios.
p Numerosos genes que parecem imitar as funções das plantas - por meio da codificação de "Domínios PA e RA que se assemelham a plantas" ou PREPARADOs - também foram identificados. "É bem conhecido que os patógenos vegetais usam proteínas que imitam os domínios vegetais necessários para a função imunológica, "disse Dangl." Imagine que o patógeno injeta diretamente na célula da planta uma proteína que imita parte de uma máquina específica do sistema imunológico. É como colocar uma engrenagem parcialmente defeituosa em uma roda - as rodas não podem mais girar. Reconhecemos que os domínios de proteína associados a plantas que identificamos podem funcionar da mesma maneira. "
p Genes em rápida evolução costumam ser uma marca registrada de uma corrida armamentista molecular entre organismos que compartilham um ambiente. Esses genes são freqüentemente usados no ataque ou defesa contra outros organismos. Duas novas famílias de proteínas de evolução rápida associadas a diferentes "estilos de vida" de bactérias associadas a plantas relacionadas foram identificadas no estudo. 1, encontrado em bactérias comensais, foi apelidado de "Jekyll"; o outro, encontrado em bactérias patogênicas, foi nomeado "Hyde". Com colaboradores da Virginia Tech e ETH (Suíça), Os cientistas do JGI descobriram que os últimos são muito eficientes em matar bactérias concorrentes, potencialmente para ajudar esses "Hydes" a assumirem o nicho das folhas.
p O catálogo completo de novos genomas e genes associados a plantas está disponível para a comunidade de pesquisa por meio de um portal da web dedicado:Características genômicas de adaptação bacteriana a plantas.
p "O banco de dados é um recurso precioso para a comunidade de pesquisa que estuda as interações planta-micróbio, pois é uma forma imparcial de identificar genes potencialmente interessantes envolvidos na interação com uma planta - incluindo muitos genes totalmente novos. Atualmente, estamos estudando experimentalmente a função de muitos dos esses genes para obter uma melhor compreensão funcional do microbioma da planta. " Levy disse.
