A proteína encerra intencionalmente a própria síntese ao desestabilizar a maquinaria de síntese - o ribossomo
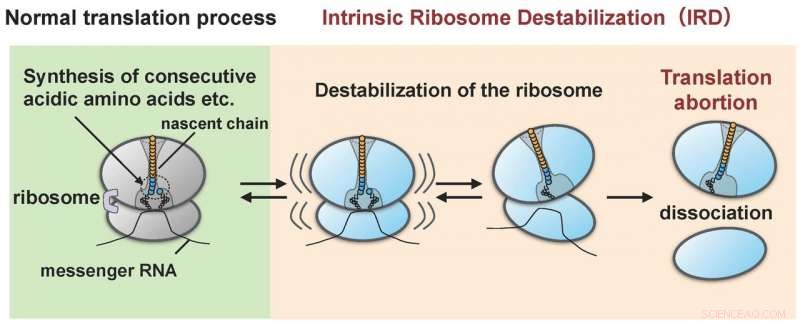 p O ribossomo torna-se desestabilizado pela proteína (cadeia nascente) que ele próprio sintetizou (IRD) e se divide em subunidades grandes e pequenas. Como resultado, a síntese de proteínas termina no meio do ciclo sem atingir o ponto final. Crédito:Hidekii Taguchi
p O ribossomo torna-se desestabilizado pela proteína (cadeia nascente) que ele próprio sintetizou (IRD) e se divide em subunidades grandes e pequenas. Como resultado, a síntese de proteínas termina no meio do ciclo sem atingir o ponto final. Crédito:Hidekii Taguchi
p Um grupo de pesquisa conjunto do Instituto de Tecnologia de Tóquio (Tokyo Tech) e da Universidade Kyoto Sangyo descobriu que uma proteína, durante sua síntese, pode desestabilizar a estrutura do ribossomo e encerrar sua própria síntese prematuramente, e descobriram que esse fenômeno é usado para adaptar a célula ao seu ambiente. p
Fundo
p As proteínas são moléculas funcionais, que suportam a vida e são estruturas tridimensionais de cadeias polipeptídicas, cadeias de aminoácidos ligados. A configuração dos aminoácidos nesta cadeia (a sequência) está escrita na sequência de DNA. Cadeias polipeptídicas são formadas onde o ribossomo, a maquinaria de síntese de proteínas, reside em uma cópia da sequência de DNA (RNA mensageiro). Ele começa a polimerizar os aminoácidos um por um, selecionando entre 20 tipos de aminoácidos para adicionar ao link de acordo com o código genético. É concluído no ponto final onde a cadeia se separa do ribossomo.
p O processo de uma proteína sendo sintetizada em um ribossomo é chamado de "tradução, "e todas as proteínas de todos os organismos vivos, incluindo humanos, são produzidos por meio da tradução. Pensou-se que, no processo de ligação de aminoácidos no ribossomo, a cadeia polipeptídica recém-formada (cadeia nascente) foi sintetizada continuamente.
p Em estudos recentes feitos por este grupo de pesquisa e outros, tornou-se aparente que a flutuação na velocidade ocorre significativamente na tradução - que algumas cadeias nascentes, dependendo da sequência de aminoácidos, agir no ribossomo que o está formando para diminuir o tempo de translação.
p
Visão geral da realização da pesquisa
p O grupo de pesquisa liderado por Hideki Taguchi da Tokyo Tech e Koreaki Ito da Kyoto Sangyo University descobriu que, quando um sistema de tradução livre de células reconstituído de E. coli foi feito para traduzir proteínas com sequências de cerca de dez aminoácidos ácidos (ácidos aspártico e glutâmico) ou sequências em que o aminoácido ácido e um aminoácido chamado prolina estão ligados alternadamente, a tradução para no meio do caminho quando tal sequência foi traduzida. Este evento abortivo ocorre quando os ribossomos que sintetizam a sequência de aminoácidos tornam-se desestabilizados como resultado da ação da cadeia nascente (os pesquisadores designam este "IRD":"desestabilização intrínseca do ribossomo"). Isso resulta na divisão do ribossomo em subunidades grandes e pequenas (Figura 1). O ribossomo, com seu papel de sintetizar vários milhares ou dezenas de milhares de tipos de proteínas dentro da célula, pensava-se que comandava a ligação de quaisquer combinações de sequências de aminoácidos. Contudo, a descoberta do IRD, em que o ribossomo é desestabilizado durante a tradução por cadeias nascentes que ele mesmo está sintetizando, indica que a tradução é algo que progride com o risco potencial de ser abortado. Proteínas que incluem sequências de aminoácidos que desencadeiam o IRD não são capazes de completar sua síntese.
p À primeira vista, este fenômeno de IRD pode parecer um defeito do ribossomo, mas o que isso indica para os organismos vivos?
p Os pesquisadores descobriram que os organismos vivos também possuem um mecanismo para neutralizar o IRD. Essa descoberta os levou a usar um mutante de E. coli sem esse mecanismo, com ribossomos, portanto, ligeiramente desestabilizados e propensos a IRD, e analisar todas as proteínas dentro de uma célula (o proteoma). Como resultado, a equipe descobriu que, no mutante, as quantidades celulares de uma série de proteínas variaram quando comparadas à cepa selvagem. Em particular, MgtA, uma proteína de membrana que transporta íons de magnésio para as células, foi encontrado para ser expresso mais de dez vezes mais. Interessantemente, o gene denominado mgtL e regulador da expressão de MgtA possuía uma sequência IRD. O resultado da análise mostrou que E. coli regula a expressão de MgtA, usando um mecanismo especial que caracteriza o fenômeno IRD devido à sequência de mgtL, em resposta a mudanças nas concentrações de magnésio na célula.
p Os íons de magnésio são necessários para muitos processos vitais dentro da célula, e é necessário para tradução, em particular, para estabilizar o ribossomo. Assim, os resultados desta pesquisa sugerem que E. coli adquiriu um mecanismo para manter a concentração de íon magnésio celular usando a capacidade IRD da mgtL para expressar uma abundância de MgtA quando seu ambiente de crescimento torna-se pobre em magnésio. Em outras palavras, um organismo vivo tem um mecanismo para monitorar mudanças no ambiente intracelular, aproveitando o fenômeno de IRD (Figura 2).
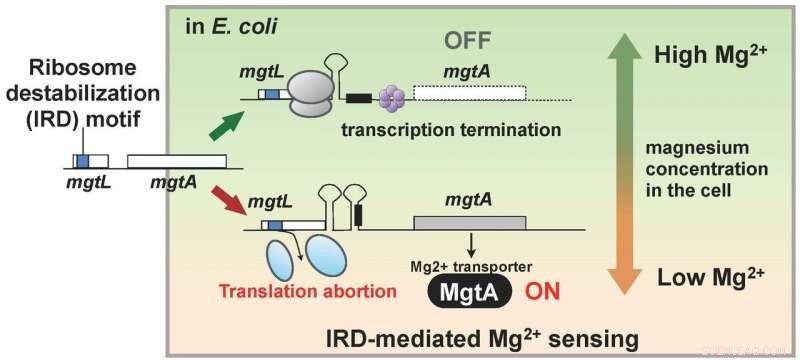 p O gene, mgtL, controlar a expressão da proteína de membrana MgtA que transporta magnésio para as células tem uma sequência desestabilizadora de ribossomo, e a tradução é abortada quando a concentração de magnésio dentro da célula é baixa. Há um mecanismo que ativa a síntese de MgtA quando a tradução de mgtL é abortada, e acredita-se que a expressão abundante resultante de MgtA aumenta a concentração de íons de magnésio dentro da célula. Crédito:Hideki Taguchi
p O gene, mgtL, controlar a expressão da proteína de membrana MgtA que transporta magnésio para as células tem uma sequência desestabilizadora de ribossomo, e a tradução é abortada quando a concentração de magnésio dentro da célula é baixa. Há um mecanismo que ativa a síntese de MgtA quando a tradução de mgtL é abortada, e acredita-se que a expressão abundante resultante de MgtA aumenta a concentração de íons de magnésio dentro da célula. Crédito:Hideki Taguchi
p
Desenvolvimentos futuros
p Este estudo revelou que, além de conter informações que determinam as estruturas tridimensionais das proteínas, as sequências de aminoácidos ainda possuem a capacidade de influenciar a estabilidade da maquinaria que a sintetiza e de abortar a tradução no meio do ciclo, eventualmente. Isso aumenta nossa compreensão dos fundamentos dos processos vitais, que são apoiados pela expressão regulada de informação genética. Cedo, a interrupção prematura da tradução não havia sido considerada um processo vital nas ciências da vida até o momento. Taguchi e os membros da equipe acreditam que sua realização pode ser expandida, em conjunto com a atual prosperidade das ciências da vida, para várias aplicações, como a produção de novas proteínas úteis ou o desenvolvimento de biofármacos.

