
Crédito:DeepMind
Os jogadores aplaudem, diga palavras como uau, bata as facas de plástico na mesa e aproveite os melhores finais de semana com a inteligência artificial como protagonista, graças à IA desencadeada nos jogos.
WIRED UK editor de ciências de, Matt Reynolds, analisou o impacto do DeepMind nos marcos da IA:"Ele superou os campeões de Go, superou jogadores profissionais de StarCraft e voltou sua atenção para o xadrez e o shogi. "
Deixe os jogos continuarem, mas as coisas sérias devem brilhar seriamente. Em resumo, podemos admirar que o desencadeamento da IA para fins de descoberta científica tornou-se especialmente vivo e saudável graças à pesquisa da DeepMind.
Observadores de tecnologia comentaram esta semana em trabalhos de pesquisa que mostram os pontos fortes da IA. "À medida que a IA amadurece como um campo (e fica sem videogames para conquistar), provavelmente mais de suas realizações se parecerão com estas:melhorias sólidas em domínios de pesquisa importantes."
Um artigo de pesquisa publicado em Natureza fez notícia instantânea como um artigo sobre como a IA pode ser usada para prever o enovelamento de proteínas. VentureBeat refere-se a desafios formidáveis que se aproximam cada vez mais das metas.
O artigo é "Predição da estrutura de proteínas melhorada usando potenciais de aprendizagem profunda, "O artigo foi publicado online em 15 de dezembro em Natureza .
O sistema DeepMind é denominado AlphaFold. Reynolds fez uma observação interessante sobre AlphaFold e o conjunto de dados:"O campo de dobramento de proteínas também está bem configurado para treinar agentes artificialmente inteligentes. Ele tem um grande conjunto de dados - o Protein Data Bank, um repositório da estrutura 3-D e composição genética de 150, 000 proteínas, que foi usado para treinar o sistema de previsão de estrutura de proteínas da DeepMind, chamado AlphaFold. "
O artigo explorou as conquistas da DeepMind até agora no uso de IA para prever o dobramento de proteínas. O grande problema do dobramento de proteínas é que, como Kelsey Piper em Vox comentou, é uma “questão crucial para o desenvolvimento de novos medicamentos”.
Três da equipe DeepMind, Andrew Senior, John Jumper e Demis Hassabis, postou uma entrada no blog em 15 de janeiro explicando as estruturas das proteínas, o desafio do dobramento de proteínas e o que eles queriam alcançar.
"Conforme demonstrado pelo paradoxo de Levinthal, levaria mais tempo do que a idade do universo conhecido para enumerar aleatoriamente todas as configurações possíveis de uma proteína típica antes de atingir a verdadeira estrutura 3-D - embora as próprias proteínas se dobrem espontaneamente, em milissegundos. Prever como essas cadeias se dobrarão na intrincada estrutura 3-D de uma proteína é o que é conhecido como 'problema de dobramento de proteínas' - um desafio no qual os cientistas trabalharam por décadas. "
A equipe mostrou que poderia treinar uma rede neural para fazer previsões precisas "das distâncias entre pares de resíduos, que transmitem mais informações sobre a estrutura do que previsões de contato. "
Eles falaram sobre otimização por meio de "um algoritmo de descida gradiente simples para gerar estruturas sem procedimentos complexos de amostragem".
O grande problema do negrito de proteína é que, disse Piper, é uma “questão crucial para o desenvolvimento de novos medicamentos”. Como assim? E, por que as "formas" são tão importantes? "A forma que as proteínas assumem prevê com quais outras substâncias elas irão interagir, Portanto, compreender o enovelamento de proteínas é crucial para a descoberta de medicamentos e pode até mesmo ser usado para desenvolver novos processos de fabricação, "Piper disse.
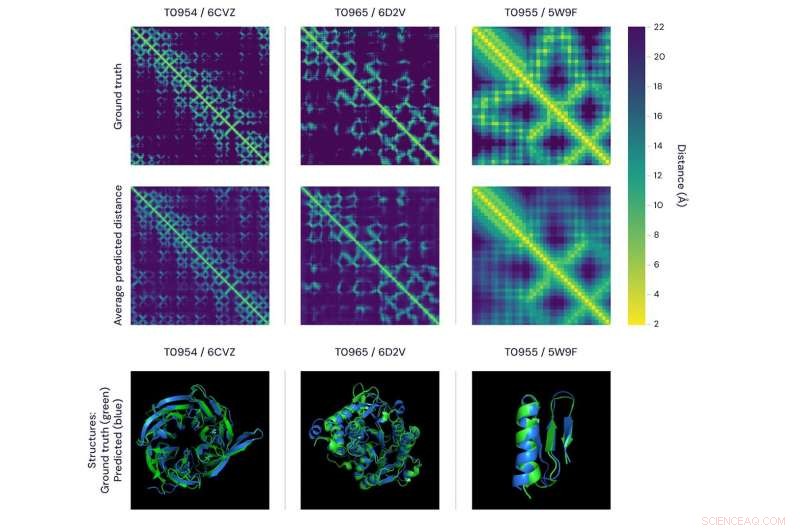
Duas maneiras de visualizar a precisão das previsões do alphafold. a figura superior apresenta as matrizes de distância para três proteínas. O brilho de cada pixel representa a distância entre os aminoácidos na sequência que compreende a proteína - quanto mais brilhante o pixel, quanto mais próximo o par. Na linha superior estão os reais, distâncias determinadas experimentalmente e, na linha inferior, a média das distribuições de distância previstas de AlphaFold. Mais importante, estes combinam bem em escalas global e local. os painéis inferiores representam a mesma comparação usando modelos 3D, apresentando previsões do AlphaFold (azul) versus dados verdadeiros (verde) para as mesmas três proteínas. Crédito:DeepMind
O blog DeepMind disse que "pensa-se que a forma de uma proteína dita sua função. Uma vez que a forma de uma proteína é compreendida, seu papel dentro da célula pode ser adivinhado, e os cientistas podem desenvolver drogas que funcionam com a forma única da proteína. "
AlphaFold pode ajudar os pesquisadores a compreender melhor a função - e o mau funcionamento - das proteínas.
Uma citação digna de nota em VentureBeat do chefe do grupo de bioinformática UCL David Jones, que aconselhou a equipe DeepMind em partes do projeto:
"Técnicas experimentais para determinar estruturas de proteínas são demoradas e caras, portanto, há uma enorme demanda por melhores algoritmos de computador para calcular as estruturas das proteínas diretamente das sequências de genes que as codificam, e o trabalho da DeepMind na aplicação de IA a este problema de longa data na biologia molecular é um avanço definitivo. "
Que tipos de técnicas experimentais são demoradas e caras? O blog mencionou microscopia crioeletrônica, ressonância magnética nuclear e cristalografia de raios-X, cada um dependendo de tentativa e erro, "o que pode levar anos de trabalho, e custam dezenas ou centenas de milhares de dólares por estrutura de proteína. "
Kyle Wiggers em VentureBeat :"Com AlphaFold, A equipe de pesquisa da DeepMind se concentrou no problema de modelar formas de destino a partir do zero, sem recorrer a proteínas resolvidas como modelos. "
Como a Natureza define o dobramento de proteínas:"O dobramento de proteínas é o processo pelo qual as proteínas alcançam sua estrutura terciária funcional (nativa) madura, e muitas vezes começa co-tradução. O dobramento de proteínas requer chaperones e muitas vezes envolve o estabelecimento gradual de estruturas secundárias e supersecundárias regulares, a saber α-hélices e folhas β, que dobra rapidamente, estabilizado por ligações de hidrogênio e pontes dissulfeto, e depois a estrutura terciária. "
Em julho, antes da publicação do artigo DeepMind, Natureza já havia notado que "a corrida para quebrar um dos maiores desafios da biologia - prever as estruturas 3-D das proteínas a partir de suas sequências de aminoácidos - está se intensificando, graças às novas abordagens de inteligência artificial (IA). "
A postagem do blog DeepMind disse que disponibilizaram o código (GitHub), e ofereceu o link, para quem deseja aprender mais ou replicar seus resultados.
Em sua postagem no blog, eles declararam por que seu sistema é importante:"Nosso sistema, AlphaFold ... baseia-se em décadas de pesquisas anteriores usando grandes conjuntos de dados genômicos para prever a estrutura da proteína. Os modelos 3-D de proteínas que AlphaFold gera são muito mais precisos do que qualquer um que veio antes. "
Seguindo em frente, o blog afirmou que uma ferramenta como AlphaFold pode ajudar os pesquisadores de doenças raras a prever a forma de uma proteína de interesse de forma rápida e econômica e pode, eventualmente, ajudá-los a contribuir para a descoberta de medicamentos eficientes, enquanto reduz os custos associados à experimentação.
A pesquisa de doenças raras não é a única área que pode se beneficiar do que realizaram. O blog falou sobre poluentes como plástico e óleo; os avanços em enzimas biodegradáveis habilitados pelo design de proteínas podem ajudar a decompor os resíduos de maneiras mais amigáveis ao meio ambiente.
"Na verdade, pesquisadores já começaram a criar bactérias para secretar proteínas que tornarão os resíduos biodegradáveis, e mais fácil de processar, "de acordo com o blog.
© 2020 Science X Network