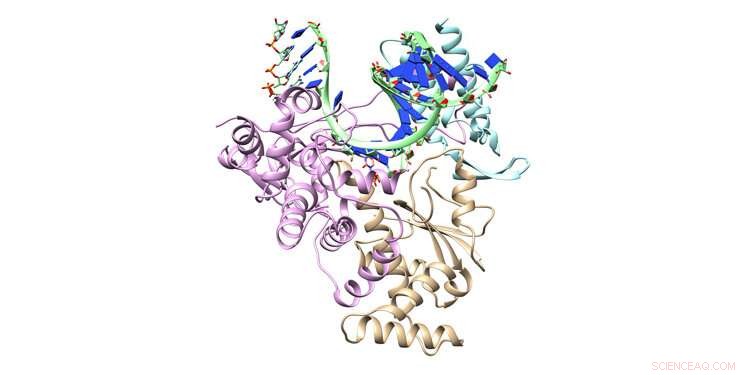
 p Remdesivir, medicamento antiviral, forma a principal linha de defesa terapêutica aprovada pela FDA contra o vírus COVID-19. Pesquisadores da University of North Texas estão usando o supercomputador Frontera para modelar como o remdesivir bloqueia a reprodução do coronavírus, na esperança de desenvolver melhorias no medicamento. Aqui são mostradas as estruturas cristalinas do modelo de complexo ternário de RNA polimerase dependente de RNA com RNA de fita dupla e trifosfato de remdesivir de entrada. Crédito:Cisneros Research Group, UNT
p Remdesivir, medicamento antiviral, forma a principal linha de defesa terapêutica aprovada pela FDA contra o vírus COVID-19. Pesquisadores da University of North Texas estão usando o supercomputador Frontera para modelar como o remdesivir bloqueia a reprodução do coronavírus, na esperança de desenvolver melhorias no medicamento. Aqui são mostradas as estruturas cristalinas do modelo de complexo ternário de RNA polimerase dependente de RNA com RNA de fita dupla e trifosfato de remdesivir de entrada. Crédito:Cisneros Research Group, UNT
p Proteínas-chave usadas pelo coronavírus para sua reprodução sendo modeladas no supercomputador Frontera financiado pela NSF pelo grupo de pesquisa Andres Cisneros da Universidade do Norte do Texas. Os objetivos da pesquisa incluem encontrar maneiras de melhorar o remdesivir terapêutico COVID-19. Alocação da Frontera financiada pela NSF concedida a Cisneros por meio do COVID-19 High Performance Computing Consortium. p Em maio de 2020, a U.S. Food and Drug Administration autorizou o remdesivir de medicamento antiviral para tratamento de emergência de COVID-19, uma de apenas quatro terapêuticas atualmente com esse status. Remdesivir interrompe a maquinaria química que o coronavírus usa para se copiar, ligação a uma enzima que faz a montagem. Embora o remdesivir tenha se mostrado promissor em ajudar os pacientes a se recuperarem do COVID-19, os cientistas estão investigando maneiras de melhorar sua eficácia.
p Uma equipe de cientistas liderada por G. Andres Cisneros, da University of North Texas, está modelando as principais partes do coronavírus que ele usa para se copiar. As simulações estão sendo feitas nos supercomputadores Stampede2 e Frontera do Texas Advanced Computing Center (TACC).
p "Tivemos a sorte de receber uma alocação no Frontera para poder trabalhar na investigação do mecanismo de drogas que visam duas proteínas específicas no COVID-19, "Disse Cisneros. Seu trabalho investiga como o remdesivir e outras drogas disponíveis inibem as proteínas NSP-12 e a principal protease, ambas as enzimas que o coronavírus necessita para a replicação. "Ao observar como essas drogas funcionam, talvez esta informação possa ser usada para melhorá-los. "
p A proteína NSP-12 reúne os nucleotídeos que compõem o RNA viral, abreviado como A, VOCÊ, G, e C, construção de conjuntos completos de material genético para novas cópias de coronavírus. A NSP-12 é, na verdade, parte de uma estrutura maior chamada RNA polimerase dependente de RNA (RDRP), que copia o RNA completo. Remdesivir liga-se ao RDRP, obstruindo o maquinário.
p "Estamos investigando como esse processo acontece, "disse Cisneros." Ao fazer isso, talvez haja uma maneira de nós e outros cientistas termos ideias sobre se e como o remdesivir pode ser melhorado. "
p A outra proteína que Cisneros está estudando é chamada de protease principal. Ele separa uma poliproteína produzida pelo SARS-CoV-2 traduzida do RNA viral em proteínas funcionais que colocam 'carne' em seus ossos virais. Pare a protease, e você impede a formação do vírus. Isso o torna um grande alvo de drogas.
p Cisneros explicou que usa a matemática e a física básicas das equações de Newton e da mecânica quântica para calcular as propriedades das proteínas, incluindo tudo o que é relevante para o seu funcionamento, como o RNA e a água. Uma abordagem chamada dinâmica molecular clássica usa as equações de Newton para simular como as proteínas se movem e interagem dinamicamente no tempo. "Estamos falando de sistemas que simulamos que têm centenas de milhares de átomos, "Disse Cisneros.
p Ele também simula as reações químicas dentro das proteínas para investigar como as drogas interrompem a RDRP ou a protease. Um método híbrido chamado QM / MM (mecânica quântica / mecânica molecular) economiza tempo computacional e dinheiro ao se concentrar mais intensamente nas interações no sítio ativo, usando a dinâmica molecular mais aproximada para todo o resto.
p O grupo Cisneros desenvolveu e mantém um programa chamado LICHEM que permite usar a abordagem QM / MM. "Uma das características do LICHEM é que ele nos permite usar abordagens para a parte da mecânica clássica que incluem uma melhor descrição da física que está acontecendo entre as moléculas no ambiente clássico, especificamente, o potencial AMOEBA ", disse Cisneros. AMOEBA é desenvolvido por Pengyu Ren da UT Austin; Jay Ponder da Universidade de Washington; e Jean-Philip Piquemal da Universidade Sorbonne em Paris com contribuições do grupo Cisneros para líquidos iônicos.
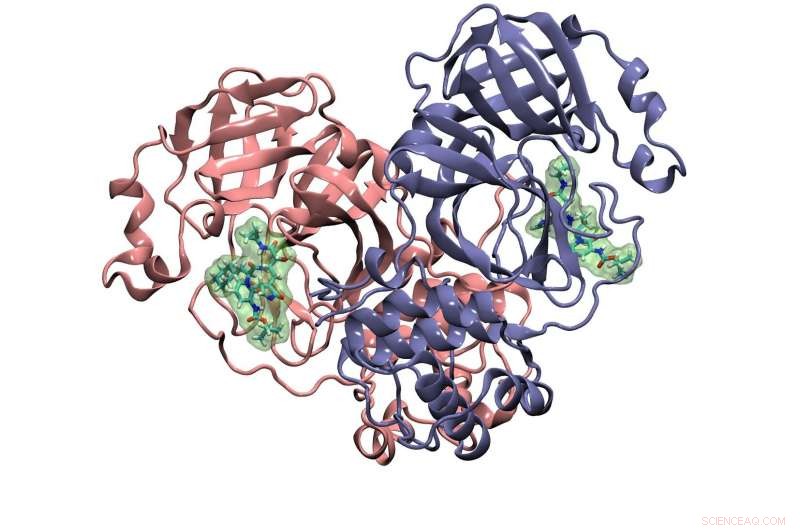 p Outro alvo de pesquisa do grupo Cisneros modelado no Frontera é uma proteína chamada protease principal. Ele cliva uma poliproteína produzida pelo vírus que constrói as proteínas funcionais das cópias de si mesmo que gera. Estrutura de cristal da protease principal do coronavírus com inibidor ligado mostrado aqui. Crédito:Cisneros Research Group, UNT
p Outro alvo de pesquisa do grupo Cisneros modelado no Frontera é uma proteína chamada protease principal. Ele cliva uma poliproteína produzida pelo vírus que constrói as proteínas funcionais das cópias de si mesmo que gera. Estrutura de cristal da protease principal do coronavírus com inibidor ligado mostrado aqui. Crédito:Cisneros Research Group, UNT
p "Frontera, com não apenas poder de computação, mas a intercomunicação entre os nós, nos permite executar esses cálculos QM / MM com muito mais, não só velocidade, mas também rendimento, "Cisneros disse. Frontera os liberou para rodar vários sistemas ao mesmo tempo." No meu grupo, Eu tenho cinco cientistas diferentes, alunos de graduação e pós-doutorado, que estão trabalhando em ambos os sistemas, mas em diferentes peças do quebra-cabeça. Todos eles podem acessar esses recursos. É definitivamente muito útil, e apreciamos muito a alocação. "
p O que impulsionou Cisneros foram as notícias em abril de 2020 sobre o relato da estrutura cristalina do SARS-CoV-2 RDRP. "Entrei em contato com meu grupo e disse a eles que, com essas informações, há algo que podemos fazer para ajudar com a pandemia, " ele disse.
p Dois dias depois da notícia, Cisneros propôs com sucesso sua pesquisa sobre alvos de drogas contra coronavírus ao COVID-19 High Performance Computing Consortium. Dezenas de instalações de supercomputação nacionais e internacionais, indústria, e organizações incluindo a TACC ofereceram seus recursos ao consórcio em apoio aos esforços dos cientistas para combater o coronavírus.
p A alocação foi inicialmente concedida apenas no Stampede2 da TACC, o carro-chefe da supercomputação da National Science Foundation (NSF), que está classificado como o 21º mais rápido do mundo e o número 2 em sistemas acadêmicos de acordo com o Top500. “Então, fomos contatados pelo TACC e gratos por termos obtido acesso ao Frontera. Agora temos acesso aos dois sistemas, o que é realmente ótimo, "Disse Cisneros.
p O supercomputador Frontera é o supercomputador acadêmico nº 1 mais rápido e o 8º mais rápido em todo o mundo. Frontera e Stampede2 são financiados pela NSF.
p "Estamos muito felizes com este sistema. Conseguimos transferir parte do conhecimento que tínhamos de Stampede2 para Frontera, "Cisneros disse. Um de seus alunos recém-formados, Erik Vazquez Montelongo, configurou todos os cálculos para LICHEM em Frontera com base no que ele aprendeu em Stampede2. "Isso realmente foi uma dádiva. O Frontera, para nossos cálculos, está funcionando muito bem. Estamos muito felizes com isso."
p Um dos pós-doutorandos do The Cisneros Group, Sehr Nazeem-Kahn, gerou o modelo para RDRP, o remdesivir e outros candidatos a medicamentos, tudo no site ativo. Com isso em mãos, eles começaram a fazer simulações.
p "Ficamos muito felizes em ver que o modelo dela estava realmente muito próximo da estrutura experimental. Isso é muito útil para nós, porque valida o modelo que foi construído pelo grupo e mostra que estamos no caminho certo, " ele adicionou.
p Atualmente, Dr. Naseem-Khan está executando simulações de dinâmica molecular deste modelo com remdesivir no Frontera. "Também estamos começando com nossos cálculos QM / MM para RDRP. No caso da protease principal, havia estruturas que também precisavam ser modeladas e posteriormente confirmadas. Isso também foi muito satisfatório, "Disse Cisneros.
p Com esses dados de estrutura, eles estão olhando para seis moléculas inibidoras diferentes. "Um daqueles, já estamos iniciando os cálculos QM / MM em Frontera, e outro no Stampede2, "Disse Cisneros. Se tudo correr bem, ele espera obter resultados nos próximos cinco a seis meses. "São cálculos muito caros, "acrescentou." Além disso, a execução da análise leva tempo. Se fôssemos usar apenas os recursos de casa, levaria vários anos. "

