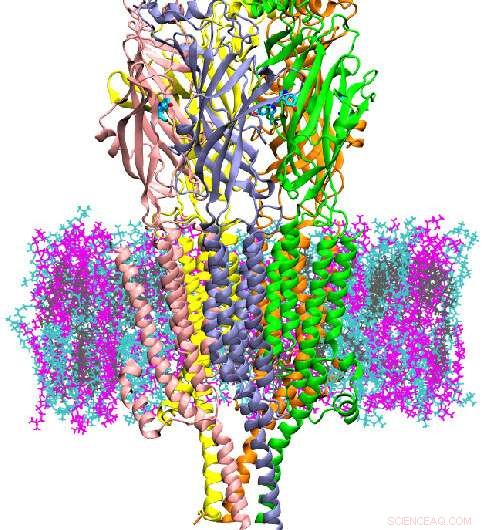
Crédito:A. James Clark School of Engineering, Universidade de Maryland
Cientistas da Universidade de Maryland (UMD) e do Instituto Nacional de Padrões e Tecnologia (NIST) descobriram uma maneira de detectar o mecanismo molecular pelo qual 5HT3A, um receptor de serotonina localizado na sinapse do neurônio, está ativado. Ter um modelo molecular dessa ativação permitirá o teste de inibidores de produtos farmacêuticos usando modelos de computador em vez de experimentos tradicionais, potencialmente reduzindo o custo e o tempo de triagem de novos medicamentos.
O estudo liderado por Jeffery Klauda, um professor de Engenharia Química e Biomolecular (ChBE) na Universidade de Maryland (UMD), foi publicado no Proceedings of the National Academy of Sciences ( PNAS ) hoje. Nicholas Guros, um assistente de pesquisa de pós-graduação da ChBE, serviu como primeiro autor do estudo e foi co-aconselhado por Arvind Balijepalli, um engenheiro mecânico no NIST.
5HT3A é um tipo de proteína conhecida como canal iônico, implicado na depressão, ansiedade e direcionados para reduzir a náusea induzida pela quimioterapia. Ele regula o fluxo de íons na fenda pós-sináptica dos neurônios. Os autores realizaram simulações de computador de 5HT3A por até 20 microssegundos - muito mais do que as simulações anteriores realizadas para
"Se você pensar na dinâmica molecular (MD) como um microscópio molecular, isso significa que somos capazes de observar o mecanismo de ativação por x10 mais do que no passado, "disse Guros." Novas capacidades computacionais podem fornecer o poder de observar a ativação molecular de proteínas complexas em escalas de tempo significativamente maiores. Quanto maior for a escala de tempo, a melhor validação que podemos ter dos resultados experimentais para demonstrar que os modelos de DM são um método robusto para estudar proteínas e testar produtos farmacêuticos. "
A ativação e dessensibilização de proteínas como 5HT3A ocorrem ao longo de milissegundos, então, embora mais perto da verdadeira escala de tempo, os cientistas ainda estão um pouco atrasados na observação de todo o ciclo de transporte da proteína. Este estudo, Contudo, foi capaz de fornecer informações sobre os efeitos da ligação da serotonina no desenvolvimento de um estado pré-ativo de 5HT3A e como a diversidade de lipídios da membrana influencia a estrutura e função das proteínas.
Muito espaço de simulação para explorar com esses modelos permanece, e isso só foi possível nos últimos anos, com avanços em poder computacional. Especificamente, este estudo foi realizado usando uma alocação baseada em subsídio no supercomputador Anton2 mantido pelo Pittsburgh Supercomuting Center (PSC) e patrocinado pela pesquisa DE Shaw. Os recursos tradicionais de computação de alto desempenho não seriam capazes de atingir as escalas de tempo deste estudo.
À medida que a velocidade e o poder da computação aumentam no futuro, o grupo espera estudar os efeitos dos produtos farmacêuticos, como granissetron ou ondansetron, em um esforço para modelar com precisão seus efeitos na proteína e investigar outras terapias direcionadas.