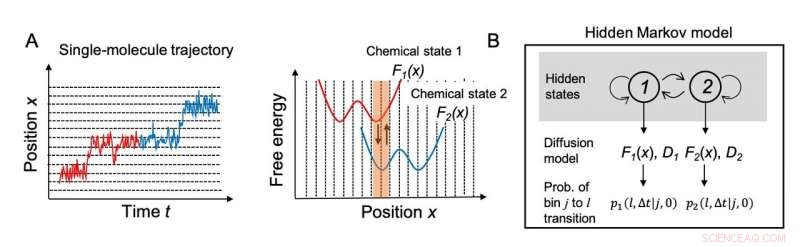
Fig. 1. (A) Trajetória do movimento de uma única molécula e perfis de energia livre dependentes do estado químico correspondentes. (B) Esquema de um modelo oculto de Markov no qual os estados químicos são considerados estados "ocultos". Crédito:NINS / IMS
Um novo método de modelagem matemática foi desenvolvido para estimar modelos de operação de motores biomoleculares a partir de dados de imagem de uma única molécula de movimento com a estrutura de inferência Bayesiana. O mecanismo de operação de um motor linear molecular "quitinase, "que se move unilateralmente em uma cadeia de quitina com a degradação da cadeia passada, foi elucidado por modelagem matemática de dados de imagem experimental com o método.
Os motores biomoleculares nas células geram movimento unidirecional, consumindo energia química adquirida por, por exemplo, hidrólise de ATP. Elucidação do princípio de operação de tais motores moleculares, que são nanomáquinas feitas pela natureza compostas de proteínas, atraiu muita atenção. Imagem de molécula única, que pode capturar diretamente o movimento dos motores moleculares, é uma técnica promissora para a compreensão do princípio de operação de motores moleculares. Contudo, ainda não está claro como o consumo de energia química, ou seja, mudança nos estados químicos dessas proteínas motoras, dá origem ao movimento unidirecional de todos os motores. Pesquisadores do Instituto de Ciência Molecular e da Universidade de Shizuoka descobriram a mudança das formas dos perfis de energia livre ao longo do movimento de um motor molecular, desencadeada por mudanças de estado químico do motor.
Os pesquisadores primeiro tentaram estabelecer um modelo computacional para descrever o movimento dos motores moleculares. O movimento de um motor pode ser considerado um movimento difusivo em perfis de energia livre que mudam de acordo com os estados químicos das moléculas que consistem no motor. Mais especificamente, como mostrado na Fig. 1A, o motor se move primeiro na superfície de energia livre do estado químico 1 (vermelho) das moléculas do motor, e então se move na superfície de energia livre do estado químico 2 (azul). Contudo, essa mudança de estado químico geralmente não é observada em imagens de uma única molécula. Os pesquisadores trataram a transição entre os estados químicos usando um modelo oculto de Markov, no qual os estados químicos são considerados estados "ocultos" (Fig. 1B).
Usando este modelo oculto de Markov, é possível calcular a "probabilidade, "que avalia a probabilidade de mostrar quão bem o modelo explica a trajetória do movimento real de uma única molécula. Também é possível incorporar o conhecimento dos perfis de energia livre como probabilidades anteriores. Os pesquisadores desenvolveram um método para estimar o estado químico perfis de energia livre dependentes, coeficientes de difusão em cada perfil, e constantes de taxa de transições entre esses estados dentro da estrutura de inferência Bayesiana por amostragem de Monte Carlo usando probabilidades posteriores expressas como um produto da probabilidade e as probabilidades anteriores.
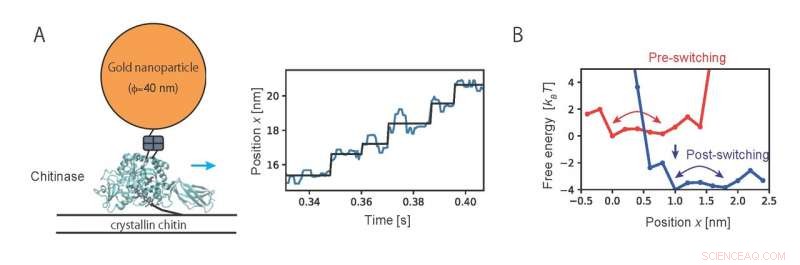
Fig. 2. (A) Movimento unidirecional observado por imagem de uma única molécula de quitinase. (B) Perfis de energia livre dependentes do estado químico estimados a partir dos dados de imagem. Crédito:NINS / IMS
Então, o método desenvolvido no presente estudo foi aplicado para analisar o movimento da quitinase, um motor molecular linear, observado por imagem de molécula única. A análise dos dados da trajetória do movimento unidirecional da quitinase com a degradação de uma cadeia de quitina revelou os perfis de energia livre característicos que governam o movimento (Fig. 2). Os resultados da análise mostraram que uma quitinase fica em um trilho da cadeia de quitina ao longo de uma barreira de energia livre relativamente baixa por movimento browniano. Então, o movimento unidirecional é obtido pela mudança de estados químicos por meio da reação de hidrólise da cadeia de quitina e da dissociação dos produtos da reação. O presente estudo fornece uma base física para o mecanismo de catraca browniana de "ponte queimada" que os pesquisadores relataram anteriormente.
"Aplicaremos nosso método desenvolvido neste estudo a vários motores moleculares e esperamos esclarecer as semelhanças e diferenças nos mecanismos dos motores moleculares. Acreditamos que novas descobertas serão obtidas por nosso método no futuro e nos darão uma pista para os princípios gerais de operação de motores moleculares. Estudos usando nosso método abrirão o caminho para o projeto de novos motores moleculares artificiais, "disse Okazaki.