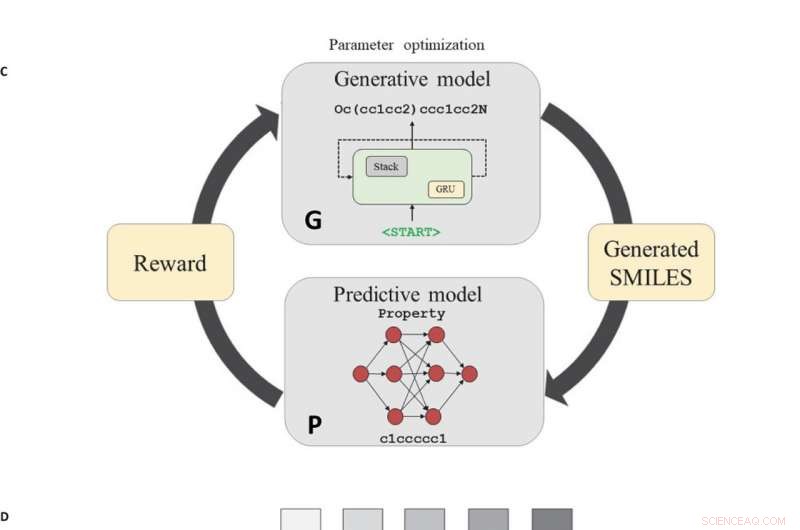
O fluxo de trabalho do algoritmo RL profundo para gerar novas sequências SMILES de compostos com as propriedades desejadas. (A) Etapa de treinamento do gerador Stack-RNN. (B) Etapa do gerador do Stack-RNN gerador. Durante o treinamento, o token de entrada é um caractere na string SMILES atualmente processada do conjunto de treinamento. O modelo gera o vetor de probabilidade p Θ (uma t | s t - 1 ) do próximo caractere com um prefixo. O vetor de parâmetros Θ é otimizado pela minimização da função de perda de entropia cruzada. No regime do gerador, o token de entrada é um caractere gerado anteriormente. Próximo, caractere em é amostrado aleatoriamente a partir da distribuição p Θ (em | s t - 1 ) (C) Pipeline geral do sistema RL para a geração de novos compostos. (D) Esquema do modelo preditivo. Este modelo recebe uma string SMILES como entrada e fornece um número real, que é um valor estimado de propriedade, como uma saída. Os parâmetros do modelo são treinados pela minimização da função de perda de 12 quadrados. Crédito: Avanços da Ciência (2018). DOI:10.1126 / sciadv.aap7885
Uma abordagem de inteligência artificial criada na Universidade da Carolina do Norte em Chapel Hill Eshelman School of Pharmacy pode aprender a projetar novas moléculas de medicamentos do zero e tem o potencial de acelerar drasticamente o design de novos candidatos a medicamentos.
O sistema é chamado de Aprendizagem por Reforço para Evolução Estrutural, conhecido como ReLeaSE, e é um algoritmo e programa de computador que compreende duas redes neurais que podem ser consideradas um professor e um aluno. O professor conhece a sintaxe e as regras linguísticas por trás do vocabulário das estruturas químicas de cerca de 1,7 milhão de moléculas biologicamente ativas conhecidas. Ao trabalhar com o professor, o aluno aprende com o tempo e torna-se melhor em propor moléculas que provavelmente serão úteis como novos medicamentos.
Alexander Tropsha, Olexandr Isayev e Mariya Popova, todos da UNC Eshelman School of Pharmacy, são os criadores do ReLeaSE. A Universidade solicitou uma patente para a tecnologia, e a equipe publicou um estudo de prova de conceito na revista Avanços da Ciência Semana Anterior.
"Se compararmos esse processo com o aprendizado de um idioma, então, depois que o aluno aprende o alfabeto molecular e as regras da língua, eles podem criar novas palavras, 'ou moléculas, "disse Tropsha." Se a nova molécula for realista e tiver o efeito desejado, o professor aprova. Se não, o professor desaprova, forçando o aluno a evitar moléculas ruins e criar boas. "
ReLeaSE é uma inovação poderosa para a triagem virtual, o método computacional amplamente utilizado pela indústria farmacêutica para identificar candidatos a fármacos viáveis. A triagem virtual permite que os cientistas avaliem grandes bibliotecas químicas existentes, mas o método só funciona para produtos químicos conhecidos. ReLeASE tem a capacidade única de criar e avaliar novas moléculas.
"Um cientista que usa a triagem virtual é como um cliente que faz um pedido em um restaurante. O que pode ser pedido geralmente é limitado pelo menu, ", disse Isayev." Queremos dar aos cientistas uma mercearia e um chef pessoal que possa criar qualquer prato que eles quiserem. "
A equipe usou ReLeaSE para gerar moléculas com propriedades que eles especificaram, tais como bioatividade desejada e perfis de segurança. A equipe usou o método ReLeaSE para projetar moléculas com propriedades físicas personalizadas, como ponto de fusão e solubilidade em água, e desenhar novos compostos com atividade inibidora contra uma enzima associada à leucemia.
"A capacidade do algoritmo de projetar novos, e, portanto, imediatamente patenteável, entidades químicas com atividades biológicas específicas e perfis de segurança ideais devem ser altamente atraentes para uma indústria que está constantemente em busca de novas abordagens para encurtar o tempo que leva para trazer um novo candidato a medicamento para ensaios clínicos, "disse Tropsha.