Pesquisadores descobrem detalhes de como as bactérias constroem barreiras protetoras e podem informar novos antibióticos
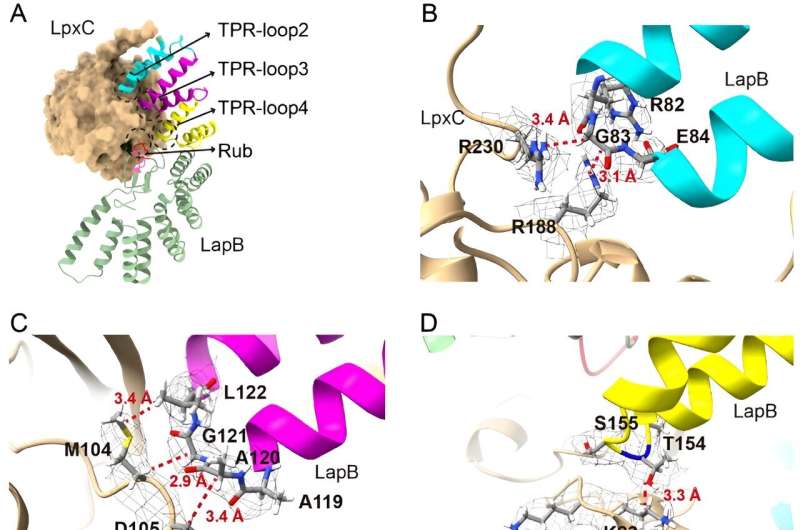 Interações entre LapBcito e LpxC. A, interações gerais entre um LapBcito e uma molécula LpxC. BE, interações detalhadas em cada site. Os resíduos envolvidos nas interações são mostrados como bastões com o mapa crioEM correspondente (mostrado em malha cinza) ao seu redor. As interações potenciais são mostradas como linhas tracejadas com distância rotulada. Crédito:Anais da Academia Nacional de Ciências (2024). DOI:10.1073/pnas.2321510121
Interações entre LapBcito e LpxC. A, interações gerais entre um LapBcito e uma molécula LpxC. BE, interações detalhadas em cada site. Os resíduos envolvidos nas interações são mostrados como bastões com o mapa crioEM correspondente (mostrado em malha cinza) ao seu redor. As interações potenciais são mostradas como linhas tracejadas com distância rotulada. Crédito:Anais da Academia Nacional de Ciências (2024). DOI:10.1073/pnas.2321510121 Os pesquisadores de Yale descobriram novos detalhes sobre como bactérias como a E. coli constroem suas barreiras protetoras, o que informará o desenvolvimento de novos antibióticos.
Bactérias resistentes a antibióticos são um problema crescente no combate a infecções. As bactérias que possuem uma camada protetora adicional em suas paredes celulares - um tipo conhecido como "Gram-negativo" em referência ao método de coloração usado para identificá-las - são especialmente difíceis de combater.
Os investigadores de Yale fizeram progressos na compreensão de como as bactérias geram esta camada protetora através de um novo estudo que revela nuances adicionais – e alvos adicionais para o desenvolvimento de novos antibióticos.
Suas descobertas foram publicadas em 18 de abril no Proceedings of the National Academy of Sciences .
Um componente crítico desta camada protetora é uma molécula chamada lipopolissacarídeo (LPS). As bactérias precisam de uma certa quantidade de LPS; muito ou pouco mata a célula. Pesquisas anteriores do laboratório de Wei Mi, professor assistente de farmacologia na Escola de Medicina de Yale, revelaram como os sensores moleculares em E. coli atingem o equilíbrio certo na produção de LPS.
No novo estudo, os investigadores aprofundaram-se na parte deste processo que evita a acumulação excessiva de LPS, com o objetivo de esclarecer como uma proteína chamada LapB se liga e estimula a degradação de LpxC, uma enzima que inicia a produção de LPS. Os pesquisadores usaram microscopia eletrônica criogênica para visualizar a estrutura do complexo criado quando essas duas moléculas se ligam.
“Olhar para a estrutura fornece a visualização mais direta de como esta parte do processo acontece”, disse Mi, autor sênior do novo estudo. “Assim que vimos a estrutura, fizemos alterações nas moléculas para ver como isso afetava a ligação, o que nos permitiu identificar quais componentes são necessários para que LapB reconheça LpxC”.
Mas os investigadores também descobriram, para sua surpresa, que o LapB tinha um segundo papel. Não só é responsável pela degradação do LpxC, mas também inibe a ação da enzima antes que a degradação aconteça.
“Basicamente, o LapB desliga o LpxC antes que ele seja destruído”, disse Mi. “Não entendemos por que as bactérias fazem isso, pois parece redundante, mas é isso que estamos investigando agora”.
Os pesquisadores especulam que esse duplo papel pode estar relacionado à flexibilidade. A degradação é um processo lento mas irreversível, enquanto a inibição é rápida e reversível. Ter ambas as capacidades pode permitir que as bactérias respondam às mudanças ambientais com mais agilidade.
“Tudo isso é relevante para o desenvolvimento de antibióticos”, disse Mi. "Esses detalhes nos ajudarão a encontrar novas abordagens e a entender por que outras não funcionam."
