Os cientistas revelam como o SID-1 reconhece o dsRNA e inicia a interferência sistêmica do RNA
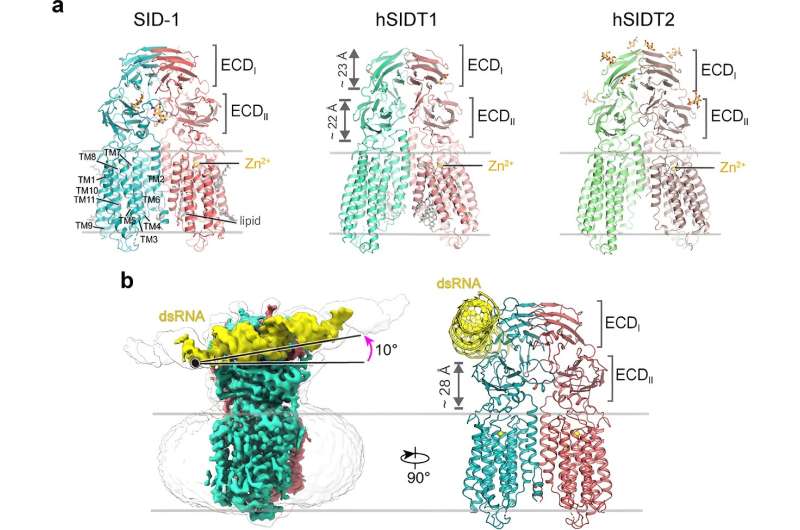 a. Estruturas Cryo-EM de homólogos de SID-1; b. o complexo SID-1/dsRNA. Crédito:Instituto de Física
a. Estruturas Cryo-EM de homólogos de SID-1; b. o complexo SID-1/dsRNA. Crédito:Instituto de Física A interferência de RNA (RNAi) é um processo biológico fascinante em vermes, plantas, fungos e metazoários que tem sido uma ferramenta valiosa para estudar a função genética e como terapêutica.
Em Caenorhabditis elegans, a proteína transmembrana multipass, proteína 1 com defeito de interferência de RNA sistêmico (SID-1), desempenha um papel indispensável na captação e entrega de RNA de fita dupla (dsRNA) entre células e tecidos, levando ao RNAi sistêmico.
Além disso, dois homólogos humanos de SID-1, o membro 1 da família transmembrana SID1 (SIDT1) e SIDT2, foram implicados no transporte de RNA. No entanto, os mecanismos moleculares subjacentes de como o SID-1 distingue especificamente o dsRNA do RNA de cadeia simples (ssRNA) e do DNA e facilita o transporte subsequente de dsRNA entre as células permanecem desconhecidos.
As respostas a essas perguntas são importantes para a compreensão do RNAi sistêmico e para auxiliar nas aplicações relacionadas ao RNA.
Zhang Jiangtao, do grupo do Prof. Jiang Daohua do Instituto de Física da Academia Chinesa de Ciências, demonstrou como o SID-1 reconhece especificamente o dsRNA e forneceu informações importantes sobre a internalização do dsRNA pelo SID-1 combinando crio-EM, experimentos in vitro e in vivo. O trabalho está publicado na revista Nature Structural &Molecular Biology .
Por mais de duas décadas, pensou-se que o SID-1 funcionava como um canal de dsRNA. Aqui, os pesquisadores resolveram estruturas crio-EM de alta resolução do SID-1 e dos homólogos humanos do SID-1 SIDT1 e SIDT2, revelando a arquitetura conservada de C. elegans e homólogos humanos do SID-1.
Os homólogos de SID-1 estão organizados de forma homodimérica. Surpreendentemente, o dímero SID-1 não apresenta um poro óbvio dentro do domínio transmembranar, sugerindo que o SID-1 pode não funcionar como um canal de dsRNA. Os ensaios de ligação ao MST confirmaram que o SID-1 pode ligar-se de forma potente e específica ao dsRNA, mas não ao dsDNA.
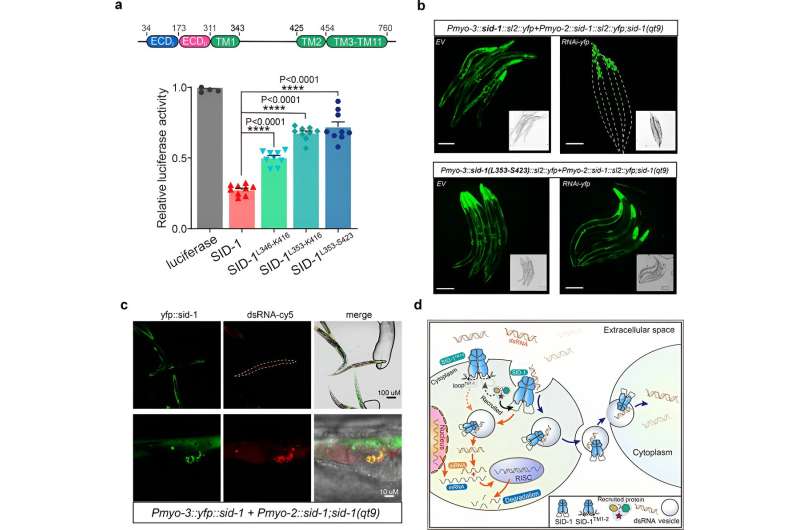 uma. Captação de dsRNA em células S2 de SID-1
TM1-2
mutantes; b. Defeitos no RNAi sistêmico do SID-1
TM1-2
; c. A colocalização de SID-1 e dsRNA; d. O modelo proposto de internalização de dsRNA pelo SID-1. Crédito:Instituto de Física
uma. Captação de dsRNA em células S2 de SID-1
TM1-2
mutantes; b. Defeitos no RNAi sistêmico do SID-1
TM1-2
; c. A colocalização de SID-1 e dsRNA; d. O modelo proposto de internalização de dsRNA pelo SID-1. Crédito:Instituto de Física
Posteriormente, os pesquisadores obtiveram a estrutura crio-EM do complexo SID-1-dsRNA, demonstrando o modo detalhado de ligação do dsRNA e os determinantes moleculares de como o SID-1 distingue o dsRNA do ssRNA e do DNA.
Curiosamente, tais determinantes não estão presentes no SIDT1 ou SIDT2 humano. Os resultados estruturais foram apoiados por estudos de mutagénese utilizando ensaios de ligação a MST, captação de dsRNA em células S2 e ensaios sistémicos de RNAi in vivo.
Finalmente, os pesquisadores mostram que a remoção das longas hélices transmembranares de alça intracelular 1 e 2 não afetou a dimerização do SID-1, a localização celular ou a ligação do dsRNA, mas prejudicou significativamente a captação de dsRNA nas células S2 e RNAi sistêmico em C. elegans.
Além disso, a co-localização revelou que o SID-1 e o dsRNA co-localizam em organelas subcelulares semelhantes a vesículas. Com base nestes resultados, os investigadores propõem que o SID-1 funciona como um receptor de dsRNA e facilita a subsequente internalização de dsRNA, recrutando proteínas relacionadas com o endocítico através da alça longa.

