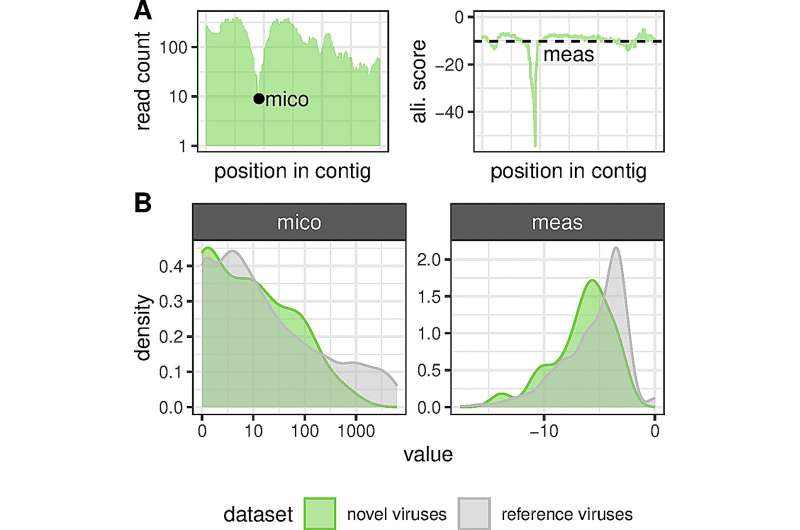
 Avaliação da qualidade da montagem. (A) Exemplo de brinquedo que visualiza como as métricas de qualidade de montagem meas (esquerda) e mico (direita) são calculadas. As pontuações de alinhamento usadas para medidas foram calculadas usando Bowtie2 e têm um valor máximo de zero correspondente a leituras de alinhamento completo sem incompatibilidades. (B) Distribuição dos valores de medidas e mico obtidos para as sequências nidovirais descobertas e montadas neste estudo (verde) e para 2.350 sequências de vírus RNA de referência (cinza). Ambos os eixos x estão em log10 escala. Crédito:PLOS Patógenos (2024). DOI:10.1371/journal.ppat.1012163
Avaliação da qualidade da montagem. (A) Exemplo de brinquedo que visualiza como as métricas de qualidade de montagem meas (esquerda) e mico (direita) são calculadas. As pontuações de alinhamento usadas para medidas foram calculadas usando Bowtie2 e têm um valor máximo de zero correspondente a leituras de alinhamento completo sem incompatibilidades. (B) Distribuição dos valores de medidas e mico obtidos para as sequências nidovirais descobertas e montadas neste estudo (verde) e para 2.350 sequências de vírus RNA de referência (cinza). Ambos os eixos x estão em log10 escala. Crédito:PLOS Patógenos (2024). DOI:10.1371/journal.ppat.1012163 De repente eles aparecem e, como o coronavírus SARS-CoV-2, podem desencadear grandes epidemias:vírus que ninguém tinha no radar. Eles não são realmente novos, mas mudaram geneticamente. Em particular, a troca de material genético entre diferentes espécies de vírus pode levar ao surgimento repentino de agentes patogénicos ameaçadores com características significativamente alteradas.
Isto é sugerido por análises genéticas atuais realizadas por uma equipe internacional de pesquisadores. Virologistas do Centro Alemão de Pesquisa do Câncer (DKFZ) foram responsáveis pelo estudo em larga escala, publicado na revista PLOS Pathogens .
“Usando um novo método de análise assistido por computador, descobrimos 40 nidovírus até então desconhecidos em vários vertebrados, de peixes a roedores, incluindo 13 coronavírus”, relata o líder do grupo DKFZ, Stefan Seitz. Com a ajuda de computadores de alto desempenho, a equipe de pesquisa, que também inclui o grupo de trabalho de Chris Lauber do Centro Helmholtz para Pesquisa de Infecções em Hanover, analisou quase 300 mil conjuntos de dados. Segundo o virologista Seitz, o facto de podermos agora analisar tão grandes quantidades de dados de uma só vez abre perspectivas completamente novas.
A pesquisa de vírus ainda está relativamente incipiente. Apenas uma fração de todos os vírus que ocorrem na natureza é conhecida, especialmente aqueles que causam doenças em humanos, animais domésticos e culturas. O novo método promete, portanto, um salto quântico no conhecimento no que diz respeito ao reservatório natural do vírus. Stefan Seitz e seus colegas enviaram dados genéticos de vertebrados armazenados em bancos de dados científicos através de seus computadores de alto desempenho com novas questões. Eles procuraram animais infectados pelo vírus para obter e estudar material genético viral em larga escala. O foco principal foram os chamados nidovírus, que incluem a família dos coronavírus.
Os nidovírus, cujo material genético consiste em RNA (ácido ribonucléico), são comuns em vertebrados. Este grupo de vírus rico em espécies tem algumas características comuns que os distinguem de todos os outros vírus de RNA e documentam sua relação. Caso contrário, porém, os nidovírus são muito diferentes uns dos outros, isto é, em termos do tamanho do seu genoma.
Uma descoberta é particularmente interessante no que diz respeito ao surgimento de novos vírus:em animais hospedeiros que são simultaneamente infectados com diferentes vírus, pode ocorrer uma recombinação de genes virais durante a replicação do vírus.
“Aparentemente, os nidovírus que descobrimos em peixes trocam frequentemente material genético entre diferentes espécies de vírus, mesmo através das fronteiras familiares”, diz Seitz. E quando parentes distantes “cruzam”, isso pode levar ao surgimento de vírus com propriedades completamente novas. Segundo Seitz, tais saltos evolutivos podem afetar a agressividade e a periculosidade dos vírus, mas também a sua ligação a determinados animais hospedeiros.
“Uma troca genética, como descobrimos nos vírus dos peixes, provavelmente também ocorrerá nos vírus dos mamíferos”, explica Seitz. Os morcegos, que – como os musaranhos – são frequentemente infectados com um grande número de vírus diferentes, são considerados um verdadeiro caldeirão. O coronavírus SARS-CoV-2 provavelmente também se desenvolveu em morcegos e de lá saltou para os humanos.
Após a troca genética entre os nidovírus, a proteína spike com a qual os vírus se acoplam às células hospedeiras muda frequentemente. Chris Lauber, primeiro autor do estudo, conseguiu mostrar isso por meio de análises de árvores genealógicas. A modificação desta molécula âncora pode alterar significativamente as propriedades dos vírus em seu benefício – aumentando a sua infecciosidade ou permitindo-lhes mudar de hospedeiro.
Uma mudança de hospedeiro, especialmente de animais para humanos, pode facilitar enormemente a propagação do vírus, como a pandemia corona demonstrou enfaticamente. As "virações de jogo" virais podem aparecer repentinamente a qualquer momento, tornando-se uma enorme ameaça e - se a situação for complicada - desencadeando uma pandemia. O ponto de partida pode ser um único animal hospedeiro duplamente infectado.
O novo processo informático de alto desempenho poderá ajudar a prevenir a propagação de novos vírus. Permite uma pesquisa sistemática de variantes de vírus potencialmente perigosas para os seres humanos, explica Seitz.
O pesquisador do DKFZ vê outra importante aplicação possível no que diz respeito ao seu campo especial de pesquisa, a carcinogênese associada a vírus:"Eu poderia imaginar que poderíamos usar a nova Computação de Alto Desempenho (HPC) para examinar sistematicamente pacientes com câncer ou pessoas imunocomprometidas em busca de vírus. Nós sabemos que o cancro pode ser desencadeado por vírus, sendo o exemplo mais conhecido o vírus do papiloma humano. Mas, até agora, provavelmente só estamos a ver a ponta do icebergue. organismo humano e aumentar o risco de tumores malignos."
