Pesquisadores usam ferramentas de modelagem de aprendizado de máquina para melhorar a tecnologia de edição de nuclease de dedo de zinco
 As nucleases de dedo de zinco (ZFNs) têm grande potencial para pesquisa translacional e uso clínico. Os cientistas conseguiram a construção eficiente de ZFNs funcionais e a melhoria da eficiência da edição do genoma usando ferramentas de modelagem de biomoléculas. Crédito:Shota Katayama/Universidade de Hiroshima
As nucleases de dedo de zinco (ZFNs) têm grande potencial para pesquisa translacional e uso clínico. Os cientistas conseguiram a construção eficiente de ZFNs funcionais e a melhoria da eficiência da edição do genoma usando ferramentas de modelagem de biomoléculas. Crédito:Shota Katayama/Universidade de Hiroshima A edição do genoma está fazendo incursões na pesquisa biomédica e na medicina. Ao empregar ferramentas de modelagem de biomoléculas, uma equipe de pesquisa japonesa está acelerando o ritmo e reduzindo o custo da tecnologia de nuclease dedo de zinco (ZFN), uma ferramenta primária de edição genética.
Em um estudo publicado na Advanced Science , pesquisadores da Universidade de Hiroshima e do Instituto Nacional Japonês de Ciência e Tecnologia Industrial Avançada demonstram como os sistemas de montagem modular orientados por aprendizado de máquina podem melhorar a edição de genes.
“A edição do genoma é uma ferramenta promissora para o tratamento de doenças genéticas em vários campos diferentes”, disse Shota Katayama, professor associado do Centro de Inovação em Edição do Genoma da Universidade de Hiroshima. “Ao melhorar a eficiência das tecnologias de edição genética, podemos alcançar maior precisão nas modificações da informação genética nas células vivas”.
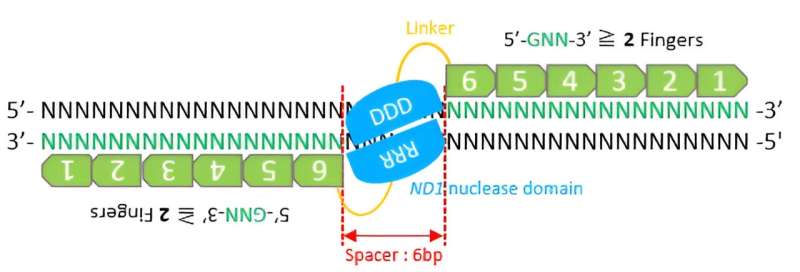
Juntamente com CRISPR/Cas9 e TALEN, a nuclease dedo de zinco é uma ferramenta importante no campo da edição do genoma. Projetadas para quebrar certas ligações dentro da cadeia polinucleotídica de uma molécula de DNA, essas proteínas quiméricas são compostas de dois domínios fundidos:domínios de ligação ao DNA e domínios de clivagem de DNA. O domínio de ligação à proteína dedo de zinco (ZF) reconhece a sequência de DNA alvo dentro do genoma completo, enquanto o domínio de clivagem envolve uma enzima especial de corte de DNA chamada endonucleases ND1.
As ZFNs apresentam algumas vantagens sobre CRISPR/Cas9 e TALEN:primeiro, ao contrário do CRISPR-Cas9, as patentes para ZFNs já expiraram, impedindo altos royalties de patentes para aplicações industriais. Em segundo lugar, as ZFNs são menores, permitindo que o DNA que codifica ZFN seja facilmente empacotado em um vetor viral com espaço de carga limitado para aplicações in vivo e clínicas.
Para cortar o DNA, duas ZFNs devem estar ligadas. Portanto, eles devem ser projetados em pares para funcionarem em qualquer novo local. No entanto, construir ZFNs funcionais e melhorar a eficiência da edição do genoma tem se mostrado um desafio.
"Fizemos grandes avanços nos métodos para derivar conjuntos de dedos de zinco para novos alvos genômicos, mas ainda há espaço para melhorar nossas abordagens de design e seleção", disse Katayama.
Métodos baseados em seleção podem ser usados para construir proteínas ZF montadas, mas esses métodos são trabalhosos e demorados. Uma abordagem alternativa para a construção de proteínas ZF montadas é a montagem de módulos ZF utilizando técnicas padrão de biologia molecular. Este método fornece aos pesquisadores um método muito mais fácil para construir proteínas ZF montadas.
No entanto, ZFNs montados modularmente têm um pequeno número de pares ZFN funcionais com uma taxa de falha de 94% para os pares ZFN testados.
Em seu estudo, os pesquisadores da Universidade de Hiroshima e do Instituto Nacional Japonês de Ciência e Tecnologia Industrial Avançada tiveram como objetivo criar uma nuclease de dedo de zinco mais eficiente e facilmente construível para edição genética usando recursos disponíveis publicamente em um sistema de montagem modular.
Uma consideração importante no projeto de ZFNs é o número de dedos de zinco necessários para uma clivagem eficiente e específica. A equipe levantou a hipótese de que a montagem modular dos módulos ZF seria útil para a construção de ZFNs com cinco ou seis dedos.
Em sua publicação, a equipe de pesquisa apresentou um método para aumentar a eficiência de construção de ZFNs funcionais e melhorar sua eficiência de edição de genoma usando três ferramentas de modelagem de biomoléculas:AlphaFold, Coot e Rosetta.
Dos 10 ZFNs testados, os pesquisadores obtiveram dois pares funcionais. Além disso, a engenharia de ZFNs usando AlphaFold, Coot e Rosetta aumentou a eficiência da edição do genoma em 5%, demonstrando a eficácia da engenharia de ZFNs baseada na modelagem estrutural.
