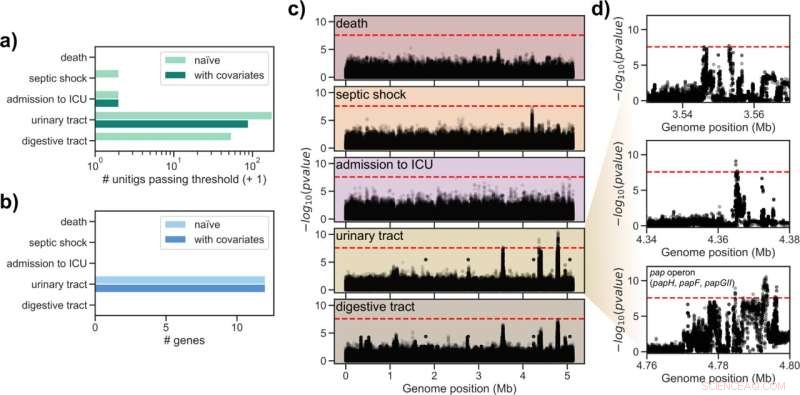
Resultados da análise de associação de todo o genoma no conjunto de dados combinado. a) Número de unidades que passam no limiar do valor p de correção de teste múltiplo para cada fenótipo alvo. b) Número de genes com unitigs significativamente associados mapeados para cada fenótipo alvo. c) Gráficos de Manhattan para mapeamento de unitigs testados para E. coli IAI39 para cada fenótipo alvo; linha tracejada vermelha indica o limiar do valor p usado para chamar associações significativas. d) Gráficos de Manhattan ampliados para o traço do trato urinário. Os genes anotados com um nome de gene em E. coli IAI39 e com unitigs associados são indicados no terceiro subpainel. Crédito:PLOS Genetics (2022). DOI:10.1371/journal.pgen.1010112
A bactéria Escherichia coli é encontrada no intestino humano e em outros lugares. Lá é inofensivo, mas em certas condições pode se tornar um patógeno. Pode causar infecções da bexiga ou até mesmo sepse. Uma equipe de pesquisadores liderada pelo professor da RESIST, Marco Galardini, da TWINCORE, juntamente com colegas da faculdade de medicina da Universidade de Paris, investigou agora se certos genes da bactéria estão associados à gravidade das doenças causadas. Eles já publicaram suas descobertas na revista
PLOS Genetics .
A Escherichia coli, muitas vezes abreviada como E. coli, faz parte da flora intestinal humana. Como um chamado comensal, normalmente não causa nenhum dano lá. No entanto, pode se tornar um patógeno no intestino e em outros órgãos. No trato urogenital, por exemplo, a E. coli causa infecções na bexiga e na corrente sanguínea pode causar sepse. O envenenamento do sangue é uma consequência temida de infecções bacterianas e pode até ser fatal em 10 a 30 por cento dos casos. Até agora, não era possível prever a gravidade de tal infecção com base na composição genética do patógeno.
Pesquisadores do TWINCORE em Hannover analisaram agora se certas variantes genéticas da E. coli estão associadas a um curso mais grave. "Realizamos o chamado estudo de associação de todo o genoma", diz Marco Galardini, chefe do grupo de pesquisa RESIST Systems Biology of Microbial Communities. “Para isso, sequenciamos amostras bacterianas de dois grandes estudos de pacientes e as correlacionamos com o curso da infecção”. Características como idade, sexo ou doenças prévias conhecidas também foram incluídas na análise.
A equipe de Galardini não conseguiu identificar genes que determinam a gravidade da doença. No entanto, eles fizeram outra descoberta interessante:"Um certo cassete de genes foi claramente associado a infecções que começaram no trato urinário", diz Galardini. A partir disso, uma estratégia para evitar doenças potencialmente fatais pode ser derivada. "No futuro, pode-se sequenciar os patógenos no caso de uma infecção da bexiga e então decidir se o tratamento medicamentoso deve ser ajustado por precaução", diz Galardini.
O fato de os pesquisadores não conseguirem provar uma conexão entre o genoma da bactéria e o curso da doença não significa necessariamente que não exista. "Pode ser que o número de amostras que estudamos tenha sido muito pequeno", diz Galardini. "Uma simulação mostrou que seria necessário dez vezes o número de amostras para detectar ou excluir o link com mais confiança."
É por isso que Galardini e seus parceiros de cooperação franceses estão preparando um estudo de acompanhamento mais amplo. Eles já apresentaram um pedido para o financiamento necessário.
