Estudos sobre enzimas vegetais mostram que as proteínas podem alterar seu arranjo estrutural com uma facilidade surpreendente
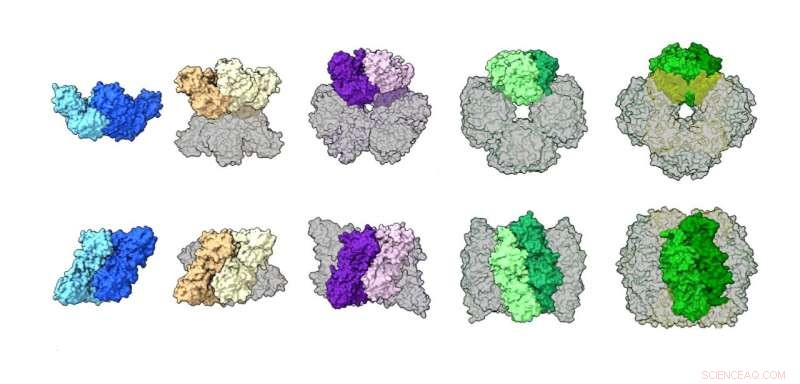
Uma comparação de conjuntos de rubisco de diferentes espécies, ilustrando uma ampla gama de estruturas, incluindo um dímero, tetrâmero, hexâmero, octâmero e hexadecâmero (proteína de 16 unidades). Crédito:Shih Lab/Berkeley Lab
Quando você pensa em proteínas – as enzimas, moléculas sinalizadoras e componentes estruturais em todos os seres vivos – você pode pensar em cadeias simples de aminoácidos, organizadas como contas em um cordão. Mas quase todas as proteínas consistem em múltiplas fitas dobradas e ligadas umas às outras, formando superestruturas 3D complicadas chamadas montagens moleculares. Um dos principais passos para entender a biologia é descobrir como uma proteína faz seu trabalho, o que requer conhecimento de suas estruturas até o nível atômico.
Ao longo do século passado, os cientistas desenvolveram e implantaram tecnologias incríveis, como cristalografia de raios-X e microscopia crioeletrônica para determinar a estrutura da proteína e, assim, responderam a inúmeras perguntas importantes. Mas um novo trabalho mostra que entender a estrutura das proteínas às vezes pode ser mais complicado do que pensamos.
Um grupo de pesquisadores do Lawrence Berkeley National Laboratory (Berkeley Lab) estudando a proteína mais abundante do mundo, uma enzima envolvida na fotossíntese chamada rubisco, mostrou como a evolução pode levar a uma surpreendente diversidade de conjuntos moleculares que realizam a mesma tarefa. As descobertas, publicadas hoje na revista
Science Advances , revelam a possibilidade de que muitas das proteínas que pensávamos saber realmente existem em outras formas desconhecidas.
Historicamente, se os cientistas resolvessem uma estrutura e determinassem que uma proteína era dimérica (composta de duas unidades), por exemplo, eles poderiam assumir que proteínas semelhantes também existiam na forma dimérica. Mas tamanho de amostra pequeno e viés de amostragem - fatores inevitáveis, uma vez que é muito difícil converter proteínas naturalmente líquidas em formas sólidas e cristalizadas que podem ser examinadas por cristalografia de raios-X -
estavam obscurecendo a realidade.

Albert Lui (à esquerda) e Patrick Shih demonstram como estudaram a estrutura e evolução da rubisco, a enzima que as plantas usam para aproveitar o CO2 para criar açúcares, em Koshland Hall na UC Berkeley. Crédito:Marilyn Sargent/Berkeley Lab
"É como se você saísse e visse alguém passeando com seu cachorro, se você nunca tivesse visto um cachorro antes e depois visse um cachorro salsicha, você pensaria, 'OK, é assim que todos os cachorros se parecem.' Mas o que você precisa fazer é ir ao parque de cães e ver toda a diversidade de cães que existe”, disse o principal autor Patrick Shih, cientista da faculdade na área de biociências e diretor de design de biossistemas vegetais no Joint BioEnergy Institute (JBEI). “Uma conclusão deste artigo que vai além da rubisco, para todas as proteínas, é a questão de saber se estamos ou não vendo a verdadeira gama de estruturas na natureza, ou se esses vieses fazem parecer que tudo parece um cachorro salsicha”.
Na esperança de explorar todos os diferentes arranjos de rubisco no parque metafórico para cães e aprender de onde eles vieram, o laboratório de Shih colaborou com especialistas em biologia estrutural da Área de Biociência usando a Fonte de Luz Avançada do Berkeley Lab. Juntos, a equipe estudou um tipo de rubisco (forma II) encontrado em bactérias e um subconjunto de micróbios fotossintéticos usando cristalografia tradicional - uma técnica capaz de resolução em nível atômico - combinada com outra técnica de resolução de estrutura, espalhamento de raios X de pequeno ângulo (SAXS), que tem resolução mais baixa, mas pode tirar instantâneos de proteínas em sua forma nativa quando estão em misturas líquidas. O SAXS tem a vantagem adicional de capacidade de alto rendimento, o que significa que pode processar dezenas de conjuntos de proteínas individuais em rápida sucessão.
Trabalhos anteriores mostraram que o tipo de rubisco mais bem estudado encontrado em plantas (forma I) sempre leva um conjunto de "núcleo octamérico" de oito grandes unidades de proteína dispostas com oito pequenas unidades, enquanto a forma II se acredita existir principalmente como um dímero com um alguns exemplos raros de hexâmeros de seis unidades. Depois de usar essas técnicas complementares para examinar amostras de rubisco de uma gama diversificada de espécies de micróbios, os autores observaram que a maioria das proteínas da forma II da rubisco são na verdade hexâmeros, com o dímero ocasional, e descobriram um tetramérico nunca antes visto (quatro unidades) conjunto.
A combinação desses dados estruturais com as respectivas sequências de genes codificadores de proteínas permitiu que a equipe realizasse a reconstrução de sequências ancestrais – um método de evolução molecular baseado em computador que pode estimar a aparência das proteínas ancestrais com base na sequência e aparência das proteínas modernas que evoluíram a partir delas.
Albert Liu verificando algumas placas de cultura de E. coli com os genes para fazer a forma II da rubisco. Crédito:Albert Liu
A reconstrução sugere que o gene da forma II da rubisco mudou ao longo de sua história evolutiva para produzir proteínas com uma variedade de estruturas que se transformam em novas formas ou revertem facilmente para estruturas mais antigas. Em contraste, durante o curso da evolução, pressões seletivas levaram a uma série de mudanças que travaram a forma I rubisco no lugar – um processo chamado entrincheiramento estrutural – e é por isso que a montagem octamérica é o único arranjo que vemos agora. De acordo com os autores, assumiu-se que a maioria dos conjuntos de proteínas foram entrincheirados ao longo do tempo por pressão seletiva para refinar sua função, como vemos com a forma I rubisco. Mas esta pesquisa sugere que a evolução também pode favorecer proteínas flexíveis.
"A grande descoberta deste artigo é que há muita plasticidade estrutural", disse Shih, que também é professor assistente na UC Berkeley. "As proteínas podem ser muito mais flexíveis, em todo o campo, do que acreditávamos."
Depois de concluir a reconstrução da sequência ancestral, a equipe realizou experimentos mutacionais para ver como a alteração da montagem da rubisco, neste caso, quebrando um hexâmero em um dímero, afetava a atividade da enzima. Inesperadamente, essa mutação induzida produziu uma forma de rubisco que é melhor na utilização de sua molécula alvo, CO
2 . Todos os rubiscos naturais frequentemente se ligam ao O
2 de tamanho semelhante molécula por acidente, diminuindo a produtividade da enzima. Há um grande interesse em modificar geneticamente a rubisco em espécies de plantas agrícolas para aumentar a afinidade da proteína pelo CO
2 , a fim de produzir culturas mais produtivas e eficientes em termos de recursos. No entanto, tem havido muito foco no sítio ativo da proteína - a região da proteína onde o CO
2 ou O
2 ligar.
"Esta é uma visão interessante para nós porque sugere que, para ter resultados mais frutíferos na engenharia da rubisco, não podemos apenas olhar para a resposta mais simples, a região da enzima que realmente interage com o CO
2 ", disse o primeiro autor Albert Liu, um estudante de pós-graduação no laboratório de Shih. "Talvez haja mutações fora desse sítio ativo que realmente participem dessa atividade e possam potencialmente alterar a função da proteína da maneira que queremos. Então isso é algo que realmente abre as portas para futuros caminhos de pesquisa."
O co-autor Paul Adams, Diretor Associado de Laboratório de Biociências e Vice-Presidente de Tecnologia da JBEI, acrescentou:"A combinação de técnicas empregadas e a natureza interdisciplinar da equipe foi uma verdadeira chave para o sucesso. O trabalho destaca o poder de combinar dados genômicos e métodos de biologia estrutural para estudar um dos problemas mais importantes da biologia e chegar a algumas conclusões inesperadas."
+ Explorar mais Estudo encontra 'elo perdido' na história evolutiva da proteína fixadora de carbono rubisco


