Resumo gráfico. Crédito:Célula (2022). DOI:10.1016/j.cell.2022.08.003
Estudos importantes na última década mostraram que o microbioma intestinal, a coleção de centenas de espécies bacterianas que vivem no sistema digestivo humano, influencia o desenvolvimento neural, a resposta a imunoterapias contra o câncer e outros aspectos da saúde. Mas essas comunidades são complexas e sem formas sistemáticas de estudar os constituintes, as células e moléculas exatas ligadas a certas doenças permanecem um mistério.
Pesquisadores da Universidade de Stanford construíram o microbioma sintético mais complexo e bem definido, criando uma comunidade de mais de 100 espécies bacterianas que foram transplantadas com sucesso em camundongos. A capacidade de adicionar, remover e editar espécies individuais permitirá que os cientistas entendam melhor as ligações entre o microbioma e a saúde e, eventualmente, desenvolvam terapias de microbioma de primeira classe.
Muitos estudos importantes do microbioma foram feitos usando transplantes fecais, que introduzem todo o microbioma natural de um organismo para outro. Embora os cientistas rotineiramente silenciem um gene ou removam uma proteína de uma célula específica ou mesmo de um camundongo inteiro, não existe esse conjunto de ferramentas para remover ou modificar uma espécie entre as centenas em uma determinada amostra fecal.
"Muito do que sabemos sobre biologia, não saberíamos se não fosse pela capacidade de manipular sistemas biológicos complexos por partes", disse Michael Fischbach, pesquisador do Instituto Sarafan ChEM-H e autor correspondente do estudo, publicado na
Célula em 6 de setembro.
Fischbach, que é professor associado de bioengenharia e de microbiologia e imunologia, e outros viram uma solução:construir um microbioma do zero, crescendo individualmente e depois misturando suas bactérias constituintes.
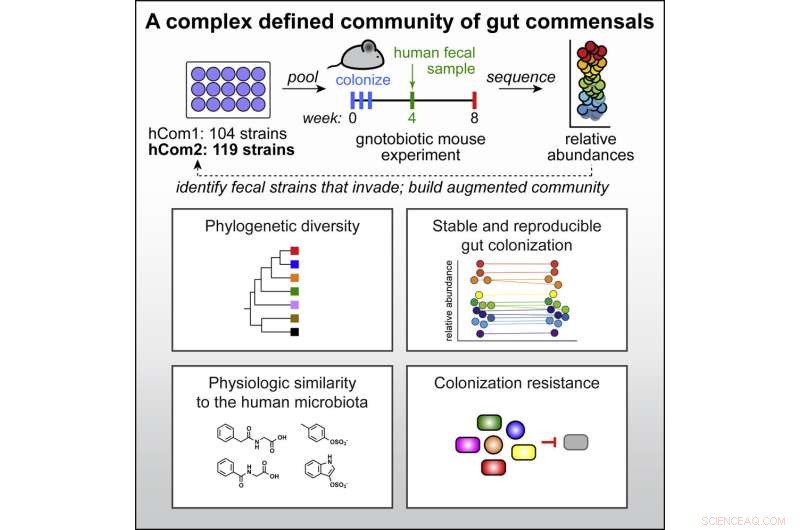
Construindo a arca Cada célula do microbioma ocupa um nicho funcional específico, realizando reações que quebram e acumulam moléculas. Para construir um microbioma, a equipe teve que garantir que a mistura final não fosse apenas estável, mantendo um equilíbrio sem que nenhuma espécie dominasse o resto, mas também funcional, realizando todas as ações de um microbioma natural e completo. A seleção de espécies para incluir em sua comunidade sintética também foi difícil, dada a variação natural entre os indivíduos; duas pessoas selecionadas aleatoriamente compartilham menos da metade de seus genes microbianos.
Os pesquisadores decidiram construir sua colônia a partir das bactérias mais prevalentes e recorreram ao Projeto Microbioma Humano (HMP), uma iniciativa dos Institutos Nacionais de Saúde para sequenciar os genomas microbianos completos de mais de 300 adultos.
“Estávamos procurando a Arca de Noé de espécies bacterianas no intestino humano, tentando encontrar aquelas que quase sempre estavam lá em qualquer indivíduo”, disse Fischbach.
Eles selecionaram mais de 100 cepas bacterianas que estavam presentes em pelo menos 20% dos indivíduos HMP. A adição de algumas espécies necessárias para alguns estudos subsequentes os levou a 104 espécies, que cresceram em estoques individuais e depois misturaram em uma cultura combinada para fazer o que eles chamam de comunidade humana, ou hCom1.
Embora convencido de que as cepas pudessem coexistir no laboratório, o verdadeiro teste era se sua nova colônia criaria raízes no intestino. Eles introduziram o hCom1 em camundongos cuidadosamente projetados para não apresentar bactérias. hCom1 foi notavelmente estável, com 98% das espécies constituintes colonizando o intestino desses camundongos livres de germes, e os níveis de abundância relativa de cada espécie permanecendo constantes ao longo de dois meses.
Invasão estrangeira Para tornar sua colônia mais completa, os pesquisadores queriam garantir que todas as funções vitais do microbioma fossem desempenhadas por uma ou mais espécies. They relied on a theory called colonization resistance, which explains that any bacterium, when introduced into an existing colony, will only survive if it can fill a niche not already occupied.
By introducing a complete microbiome, in the form of a human fecal sample, to their colony and tracking any new species that took up residence, they could build a more complete community.
Some were skeptical that this would work. "The bacterial species in hCom1 had lived together for only a few weeks," said Fischbach. "Here we were, introducing a community that had coexisted for a decade. Some people thought they would decimate our colony."
Remarkably, hCom1 held its own, and only about 10% of cells in the final community came from the fecal transplant.
They found over 20 new bacterial species that inserted themselves in at least two of their three fecal transplant studies. Adding those to their initial community and removing those that failed to take root in mouse guts gave them a new community of 119 strains, dubbed hCom2. This second iteration, still made from individually growing and then mixing the constituents, made mice even more resistant to fecal challenges than the first.
Final challenge To demonstrate the utility of their synthetic microbiome, the team took hCom2-colonized mice and challenged them with a sample of E. coli. These mice, like those that were colonized with a natural microbiome, resisted infection.
Prior studies have shown that a healthy natural microbiome leads to protection, but Fischbach and colleagues could take this a step further by iteratively eliminating or modifying certain strains to determine which ones specifically conferred protection. They found several key bacteria and plan to conduct further studies to narrow down to the most critical species.
Fischbach believes that hCom2, or future versions of it, will enable similar reductionist studies that reveal the bacterial agents involved in other areas, like immunotherapy responses.
"We built this consortium for the broader research community. We want to get this into as many hands as possible to have an impact on the field," said Fischbach.
He also envisions that this method of building a microbiome from the ground up will make engineered microbiome-based therapies possible in the future. As the director of the Stanford Microbiome Therapies Initiative (MITI), an initiative launched in 2019 by Sarafan ChEM-H and the Department of Bioengineering, he aims to construct engineered communities that could one day be transplanted into people to treat or prevent a variety of diseases.
+ Explorar mais Ecological principles at play in gut microbiome
