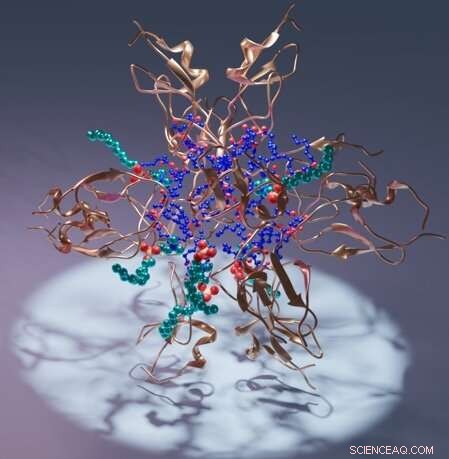
Renderização artística de um domínio de proteína quinase C C1 (cobre), seu ligante diacilglicerol (azul) e detergente (ciano). Crédito:Sachin Katti.
Em um grande avanço para o design racional de medicamentos, uma equipe da Texas A&M AgriLife descreveu várias estruturas de proteínas de um participante crucial nos processos celulares. O avanço pode trazer novas ideias para tratamentos de doenças como Alzheimer, AIDS, câncer e outras.
Especificamente, o trabalho descreve o domínio C1 da proteína quinase C, PKC, que ajuda a regular a atividade da proteína nos organismos. Nas estruturas, o domínio C1 envolve diferentes moléculas de intenso interesse terapêutico, fornecendo o primeiro guia confiável de resolução atômica para projetar candidatos a medicamentos.
Publicado em 16 de maio em
Nature Communications , a pesquisa foi dirigida por Tatyana Igumenova, Ph.D., professora associada do Departamento de Bioquímica e Biofísica do Texas A&M College of Agriculture and Life Sciences. O principal autor do projeto é Sachin Katti, Ph.D., um pós-doutorando trabalhando com Igumenova.
O estudo envolveu uma colaboração com Inna Krieger, Ph.D., professora assistente de pesquisa, e James Sacchettini, Ph.D., professor, ambos do Departamento de Bioquímica e Biofísica.
Uma das estruturas proteicas mais procuradas Uma célula saudável responde a sinais químicos de maneira precisa e complexa. Receber insumos químicos do ambiente da célula e encaminhá-los para os sistemas de controle central dentro do núcleo da célula é tarefa de proteínas especializadas, como a PKC.
Atividade inadequada de PKC aparece em muitas doenças humanas. Como resultado, há muito interesse em encontrar maneiras de ajustar a atividade da PKC com drogas. O projeto de tais drogas oferecerá novas abordagens para o tratamento da doença de Alzheimer, AIDS, câncer e muito mais.
"A proteína quinase C é uma das proteínas mais intensamente estudadas em biologia celular e farmacologia", disse Igumenova. "Um grande obstáculo tem sido a falta de informações estruturais precisas para orientar os esforços de design de medicamentos".
Uma complicação para o design de medicamentos é que a família PKC tem 11 membros. Diferentes membros da família PKC podem ter efeitos fisiológicos opostos, portanto, um candidato a medicamento bem-sucedido deve ser seletivo sobre qual PKC visa.
Para fazer isso, os candidatos a medicamentos devem ajustar um PKC alvo como uma chave para uma fechadura. Mas determinar a estrutura 3D de um PKC "on-switch" - o domínio C1 - ligado a ativadores de PKC não foi fácil.
Estruturas de proteínas são normalmente resolvidas usando cristalografia de raios-X. A técnica envolve o uso de raios X para determinar a posição dos átomos em um cristal. Para esse método, os pesquisadores precisam criar condições em que a proteína de interesse se cristalize. No entanto, esforços intensos em muitos laboratórios de pesquisa nas últimas três décadas não conseguiram produzir cristais de domínios C1 ligados a ligantes relevantes. Devido a essa falta de progresso, vários pesquisadores declararam a tarefa impossível, disse Igumenova.

Cristais de um domínio de proteína quinase C formados espontaneamente no tubo de amostra de RMN de Katti. Crédito:Sachin Katti.
Resolvendo um problema de 30 anos Aceitando o problema como desafiador, Katti e Igumenova decidiram estudar as moléculas em solução usando ressonância magnética nuclear, RMN, espectroscopia. Isso envolveu encontrar os componentes certos para imitar as membranas celulares, onde o domínio C1 encontraria ligantes.
"Então, um belo dia, Sachin descobriu cristais se formando em um antigo tubo de RMN", disse Igumenova. "Dou todo o crédito a Sachin, que basicamente disse:'Vou testá-los e ver se eles são realmente a proteína.' E ele estava certo. Isso nos deu confiança de que a cristalização é possível."
Por sua vez, Katti dá crédito aos insights obtidos da RMN e um pouco de sorte.
"Acho que essa é a beleza de fazer pesquisa em que você precisa usar várias abordagens", disse ele. "Você nunca sabe quando uma abordagem será útil para fazer algo com outras abordagens."
Percepções de cristalografia de RMN e raios-X As novas estruturas de proteínas, juntamente com os resultados de NMR da equipe, já renderam informações interessantes. Um mistério de longa data é como os domínios C1 podem acomodar ligantes que têm estruturas químicas muito diferentes, disse Igumenova.
"Nosso trabalho anterior de RMN indicou que as alças do domínio C1 que ligam os ligantes são muito dinâmicas", disse Igumenova. "Esse domínio C1 é como um PAC-man. Ele se liga à membrana e depois procura um ligante. Uma vez que encontra o ligante, ele se prende."
Além disso, a estrutura mostra que o sulco de ligação ao ligante tem uma superfície "amante de água" ou hidrofílica na parte inferior e uma superfície "repelente de água" ou hidrofóbica na parte superior.
"Se você pensar em uma molécula lipídica, o grupo da cabeça é hidrofílico e a cauda é hidrofóbica", disse Igumenova. "Então, quando os domínios C1 se ligam aos ligantes lipídicos, os padrões coincidem."
Os resultados da equipe incluem a estrutura de um domínio C1 ligado ao seu ligante natural, o diacilglicerol. Além disso, a equipe descreve várias outras estruturas de C1 que incluem diferentes compostos de interesse farmacológico.
O trabalho também fornece um método para testar diferentes candidatos a drogas, disse Katti.
"Se você quer estudar peixes, você quer estudá-los na água", disse Katti. "Agora sabemos como criar um ambiente semelhante a uma membrana onde esses compostos muito hidrofóbicos podem ser testados quanto à ligação C1".
Em seguida, Katti e Igumenova planejam explorar domínios C1 de outros membros da família PKC.
"É importante focarmos nos domínios C1 porque eles têm diferenças inerentes que podem ser exploradas para alcançar a seletividade", disse Igumenova. "O que estamos descobrindo agora é que nem todos os domínios C1 são criados iguais."
+ Explorar mais Descobertas revelam novo mecanismo de ativação para ALK

