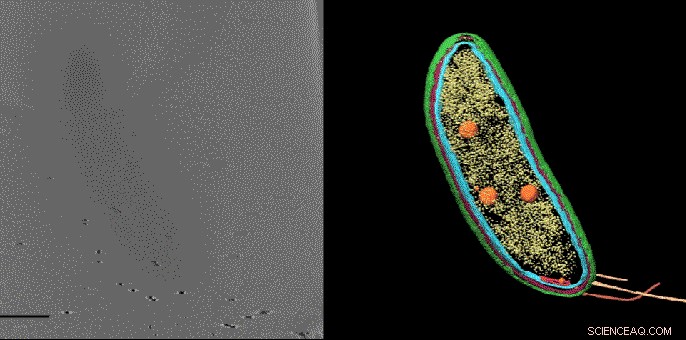
Cientistas da Universidade de Stanford pensaram que uma área aparentemente vazia em uma das extremidades da bactéria Caulobacter pode conter duas proteínas envolvidas na divisão celular. Ao rotular as proteínas com marcadores fluorescentes e, em seguida, imagens dessas mesmas amostras com tomografia crioeletrônica, eles foram capazes de confirmar esta localização e mostrar exatamente como as proteínas estavam organizadas. Crédito:Peter Dahlberg et al., PNAS , 8 de junho de 2020
Os cientistas casaram duas das técnicas de microscopia mais poderosas da atualidade para fazer imagens que apontam, pela primeira vez, as identidades e localizações precisas de proteínas individuais dentro do contexto detalhado de células bacterianas. Esta informação é crucial para aprender como as moléculas de proteína trabalham juntas para organizar a divisão celular e realizar outras tarefas importantes, como permitir que os micróbios farejem alimentos e o perigo.
O novo método já revelou novas informações sobre proteínas bacterianas e suas vizinhanças celulares próximas. Os pesquisadores dizem que também tem potencial para responder a questões fundamentais sobre a maquinaria molecular dos vírus, parasitas, e processos como fotossíntese.
"Este é um grande salto para a biologia, e eu acho que há muitos, muitos sistemas que se beneficiarão com este tipo de imagem, "disse a professora de Stanford Lucy Shapiro, cujo grupo de pesquisa participou do estudo.
O novo método híbrido, chamada de imagem correlacionada por anotação com moléculas únicas, ou CIASM (pronuncia-se "abismo"), foi desenvolvido por Peter Dahlberg, um pesquisador de pós-doutorado no laboratório do Professor W. E. Moerner na Universidade de Stanford.
É uma variação de uma técnica chamada microscopia de molécula única de baixa temperatura, inventado por Moerner três décadas atrás, que anexa rótulos brilhantes às moléculas para que possam ser individualmente identificadas. Este método é a base da microscopia de fluorescência de super-resolução, o tema do Prêmio Nobel de Química de 2014 de Moerner.
O que Dahlberg fez foi encontrar uma maneira de fazer esse tipo de imagem de fluorescência funcionar em temperaturas abaixo de zero, para que as mesmas amostras também pudessem ser examinadas com tomografia eletrônica criogênica (CET). CET usa fluxos de elétrons para fazer imagens 3-D de células congeladas por flash e seus componentes em resolução quase atômica. A combinação de CET com a imagem fluorescente permite que os cientistas vejam as moléculas marcadas no contexto da célula circundante, uma perspectiva crucial para a compreensão de seu papel na maquinaria celular.
"Podemos rotular moléculas específicas de interesse para que a luz que vemos venha apenas dessas moléculas, e então descobrimos onde eles estão dentro de cerca de 10 nanômetros, ou bilionésimos de um metro. Isso nos dá uma imagem muito mais precisa do que está acontecendo, "Dahlberg disse." Tiramos as fotos ultraprecisas fornecidas pela CET e adicionamos um pouco de cor. "
Ele adicionou, "É empolgante desenvolver novos métodos de imagem. Quando terminar, você tem que dar um passo para trás e olhar para todas as novas questões que você pode atacar. "
Com CIASM, a equipe de pesquisa foi capaz de identificar a localização de três tipos de proteínas em imagens CET de alta resolução de bactérias obtidas no Laboratório Nacional do Acelerador SLAC do Departamento de Energia. Os resultados foram relatados no Proceedings of the National Academy of Sciences hoje.
"Cada método tem suas vantagens e desvantagens, "Moerner disse, "e esta é uma situação agradável onde podemos combinar dois métodos para aprender mais."
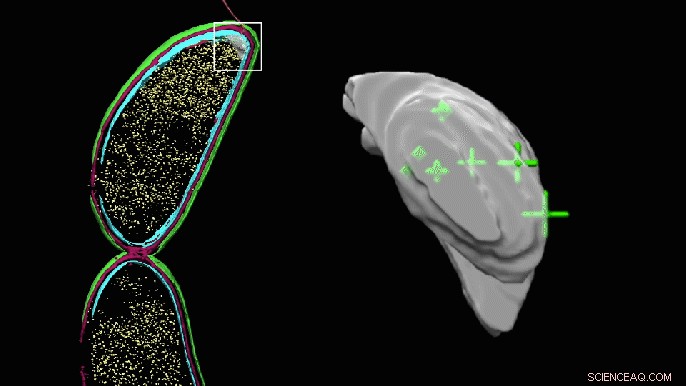
Uma imagem 3D giratória da bolsa aparentemente vazia em uma das extremidades de uma célula Caulobacter agora mostra a localização precisa das moléculas PopZ. O bolso parece irregular porque foi colorido manualmente para destacar a área onde os pesquisadores pensaram que as moléculas poderiam estar, mas não conseguiu identificar diretamente na tomografia crioeletrônica. Crédito:Peter Dahlberg et al., PNAS , 8 de junho de 2020
Encontrando ordem em uma sopa celular
Mesmo em células bacterianas relativamente simples, localização é tudo, disse Saumya Saurabh, um pesquisador de pós-doutorado no laboratório de Shapiro que desempenhou um papel importante na pesquisa.
"As pessoas tendem a pensar nas bactérias como sacos de proteínas sem organização, "ele disse." Mas isso não é verdade, e, de fato, muitas das moléculas nas bactérias estão precisamente localizadas no espaço e no tempo. Se eles não estiverem na posição certa, a célula morre. O que o trabalho de Pete está finalmente nos permitindo fazer é olhar para dentro com resolução molecular e descobrir quando e onde essas moléculas estão localizadas em relação umas às outras. "
Caulobacter crescentus, por exemplo, uma espécie bem estudada de bactérias de água doce, é conhecido por se dividir em dois tipos muito diferentes de células-filhas:uma nada livremente, enquanto o outro forma um talo e se fixa a uma superfície. Como cada célula filha obtém o que precisa para seguir seu caminho único é um mistério antigo.
Os cientistas já haviam identificado pequenas áreas em cada extremidade da célula em divisão que podem conter proteínas que desempenham papéis importantes nesta divisão celular desequilibrada. Uma das proteínas, PopZ, é encontrado em ambas as extremidades da célula em divisão, enquanto o outro, SpmX ("Spam-X") é encontrado apenas na metade que desenvolverá um talo.
Para este estudo, Saurabh e o estudante de pós-graduação Jiarui Wang marcaram proteínas em Caulobacter com marcadores fluorescentes. Então Dahlberg congelou essas amostras, realizou imagens de fluorescência de molécula única neles com a ajuda da estudante Annina Sartor, e os levou para as instalações Stanford-SLAC Cryo-EM para imagens CET dirigidas por Wah Chiu, professor em Stanford e SLAC.
Mapeando um hangout de proteína
As imagens combinadas não apenas confirmaram que ambas as proteínas estavam nas áreas que os cientistas suspeitavam, mas também revelou exatamente como eles estavam dispostos:SpmX foi incorporado na membrana interna da célula e projetou-se para o interior da célula, onde entrou em contato direto com PopZ.
"A orientação exata deste complexo de proteínas tem sido debatida nos últimos 12 anos, "Saurabh disse." Fomos capazes de observar os parceiros de proteína com resolução requintada. Agora temos uma imagem muito precisa de como essas proteínas se comunicam na célula. "
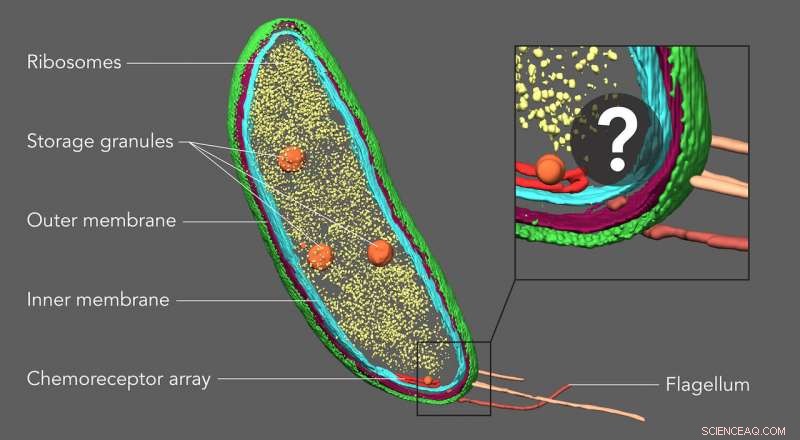
Com uma técnica chamada tomografia crioeletrônica, os cientistas podem criar imagens 3D detalhadas de células, como esta bactéria Caulobacter, e destacar seus componentes - neste caso, as membranas celulares (vermelho e azul), casca de proteína (verde), fábricas de proteínas conhecidas como ribossomos (amarelo) e grânulos de armazenamento (laranja). Mas até agora, estruturas menores e moléculas individuais não puderam ser identificadas e precisamente localizadas nessas imagens. Uma nova técnica de imagem desenvolvida em Stanford preenche essa lacuna, revelando pequenas moléculas que não são visíveis aqui. Crédito:Peter Dahlberg et al., PNAS , 8 de junho de 2020
A equipe testou a precisão do CIASM usando-o para confirmar a localização de uma proteína chamada McpA, que era conhecida por fazer parte de uma matriz quimiorreceptora na bactéria. "Proteínas extremamente sensíveis nesta matriz servem como nariz de Caulobacter, "Saurabh disse, "sentindo a química do meio ambiente para que possam se afastar de coisas desagradáveis e se mover em direção à glicose que comem."
A matriz aparece como linhas pretas paralelas em imagens CET, e marcação fluorescente das mesmas imagens identificou a localização de proteínas individuais McpA em cerca de 10 nanômetros.
Uma visão detalhada dos pontos quânticos
Em separado, estudo paralelo, publicado em 24 de abril em Angewandte Chemie , os pesquisadores usaram uma técnica semelhante para olhar para pontos quânticos únicos, com alguns resultados surpreendentes.
Os pontos quânticos são cristais em nanoescala de material semicondutor que apresentam fluorescência natural em cores determinadas por seu tamanho, forma e composição. Esses pontos são usados em pesquisas para rotular e rastrear proteínas e outros materiais biológicos, e têm aplicações potenciais na eletrônica do futuro, iluminação, Computação quântica, imagens médicas e outras áreas.
Neste estudo, o objetivo era ver como os detalhes estruturais mais finos de pontos individuais estavam relacionados a detalhes específicos de suas propriedades ópticas, disse Davis Perez, um Ph.D. aluno no laboratório de Moerner.
"Conseguimos ver alguns comportamentos surpreendentes dos pontos quânticos individuais - por exemplo, em sua resposta à excitação com luz laser, "disse ele." Mas o aspecto mais emocionante para mim é que o método que desenvolvemos para estudar os pontos quânticos também pode ser usado para estudar sistemas biológicos, como proteínas fotossintéticas, onde a energia é transferida entre grupos de proteínas, e ver como funciona a maquinaria fotossintética. "
Moerner disse que seu laboratório está trabalhando com Chiu para enfrentar esses desafios.
"É o início da combinação dos dois métodos, e estamos animados para explorar mais colaborações ligando luz e elétrons, "Chiu disse." Esta abordagem de imagem híbrida tem o potencial de descobrir estruturas de componentes moleculares envolvidos em processos biológicos essenciais em células que abrangem todos os domínios da vida. "