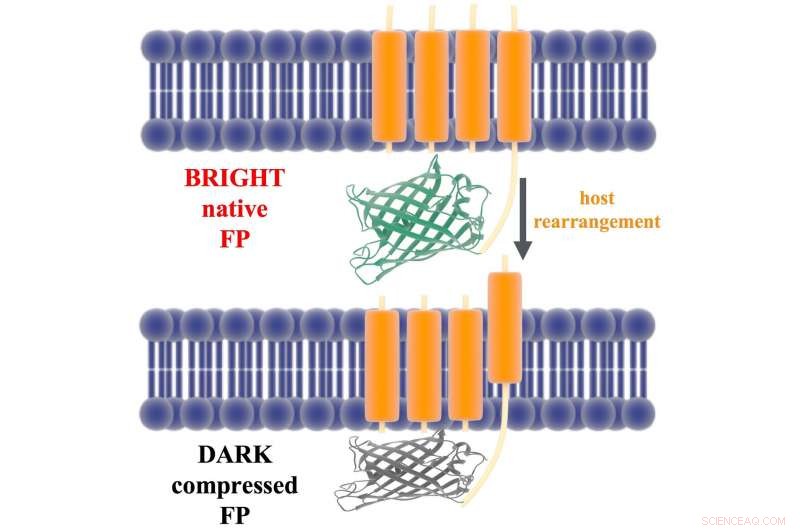
Uma ilustração mostra a corrente que liga o domínio fluorescente de uma proteína ArcLight (verde e cinza) ao seu domínio de detecção de voltagem (laranja) na membrana de um neurônio. Cientistas de Rice sugerem em um novo estudo que a despolarização da membrana move o domínio de detecção de voltagem, que, por sua vez, comprime a proteína fluorescente e a extingue. Crédito:Lena Simine
Cientistas da Rice University extinguiram efetivamente o debate sobre o mecanismo por trás de um biossensor fluorescente que monitora os neurônios detectando mudanças na voltagem.
O trabalho liderado pelo químico teórico de Rice, Peter Rossky, e pela pesquisadora de pós-doutorado Lena Simine, confirmou por meio de simulações de computador sua teoria de que um processo mecânico controla a extinção da fluorescência no ArcLight, um indicador de voltagem sintético colocado dentro de proteínas que revestem as membranas internas dos neurônios.
Por meio de seus modelos, os pesquisadores acoplaram o mecanismo e a fluorescência à intensidade dos campos elétricos que observaram no cromóforo, a parte fluorescente da proteína. Seus resultados mostraram que uma medida simples do campo em uma simulação poderia ser usada para prever se e como os novos sensores fluorescentes se comportariam antes que os pesquisadores os sintetizassem. Disse Rossky.
O estudo aparece no Jornal da American Chemical Society .
ArcLight, desenvolvido pelo neurocientista de Yale Vincent Pieribone em 2012, é uma proteína indicadora de voltagem de fluorescência geneticamente codificada. Ele contém uma mutação que torna o sinal de fluorescência mais fraco quando a voltagem aumenta e aumenta quando a voltagem cai. Isso o torna útil para rastrear sinais no sistema nervoso, expressando a proteína nos neurônios e vendo como eles se acendem.
A proteína é ligada à parede celular do neurônio por um componente sensor de voltagem que se move alguns angstroms quando um sinal de outro neurônio altera a carga elétrica na membrana. Os pesquisadores de Rice teorizaram que o movimento puxa a proteína contra a membrana, comprimindo-o e extinguindo a fluorescência.
Rossky disse que mudar a forma da proteína traz dois resíduos um nanômetro mais próximos um do outro. Isso é o suficiente para ditar como o cromóforo se livra da energia, tanto como luz (liberando fótons e fluorescência) ou como calor.
"Nós formulamos a hipótese de qual mudança de geometria ocorre na proteína como resultado da resposta da membrana, "Rossky disse." E então perguntamos, 'Isso muda a fluorescência?' E descobrimos que sim. Além disso, mostramos que monitorar uma qualidade muito mais simples - o campo elétrico ao longo de dois eixos de onde vem a fluorescência - é suficiente para descrever completamente a resposta. "
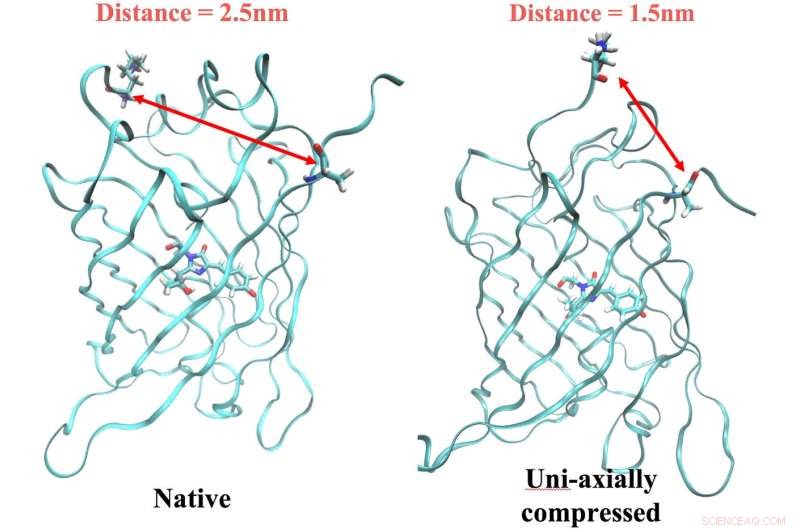
A proteína ArcLight nativa à esquerda, com resíduos de âncora com 2,5 nanômetros de distância, fluoresce quando acionado pela luz na frequência certa. Mas a fluorescência desliga quando a proteína é comprimida, o que aproxima as âncoras um nanômetro. Os pesquisadores de Rice encontraram uma ligação entre o mecanismo e um sinal elétrico na proteína que pode ser usado como um marcador ao simular novas proteínas fluorescentes com modelos de computador. Crédito:Lena Simine
ArcLight provou ser um bom modelo. Pieribone, um colaborador do Rice, disse aos participantes de uma palestra de 2014 na Rice que nem ele sabia exatamente como funcionava. A palestra inspirou Simine, que tinha acabado de chegar ao arroz, para iniciar um estudo do mecanismo.
"Eu pensei, 'Isso soa como um bom projeto para mim, '" ela disse.
Trabalhar com pesquisadores do grupo de José Onuchic no Centro de Física Teórica Biológica de Rice (CTBP) permitiu a Simine, um físico químico por formação, para aproveitar a experiência do centro na simulação de proteínas para teste.
Ela disse que um debate de uma década entre os cientistas não conseguiu determinar se as propriedades mecânicas ou elétricas das proteínas causaram sua fluorescência. Acabou sendo um pouco dos dois.
"Um artigo recente forneceu evidências computacionais de que ele é predominantemente eletrostático, e faz sentido porque a proteína é muito macia, "Simine disse." Nós também percebemos que essas mutações estão aderindo à membrana, e quando eles fazem, a orientação da proteína permite que a proteína seja comprimida. "Ela descobriu que mudanças eletrostáticas na membrana neuronal desencadearam a mudança física que extingue a fluorescência, mas também deixou um traço elétrico na proteína que pôde ser observado na simulação.
"Nós pensamos nisso e chegamos a uma coordenada de reação, "disse ela." Podemos pegar qualquer mutação da sequência desta proteína e traduzi-la em dois números que são as entradas para este modelo, os campos eletrostáticos em torno do cromóforo. É um bom, elegante teoria fenomenológica. "
O laboratório planeja testar sua técnica em proteínas fluorescentes sintetizadas de forma personalizada e simulações correspondentes para ver se sua teoria e experimentação continuam a se alinhar. Se eles fizerem, eles esperam que seus modelos sejam altamente úteis para biólogos sintéticos que fazem novas classes de marcadores fluorescentes.
"Se você quiser saber a fluorescência de uma determinada molécula, você faz o experimento, "Rossky disse." Mas se você quer saber por que funciona, esses cálculos são incrivelmente valiosos. "