Genética da batata-doce:Uma atualização abrangente da anotação do genoma do Taizhong 6
 Resumo gráfico. Crédito:Plantas Tropicais (2024). DOI:10.48130/tp-0024-0009
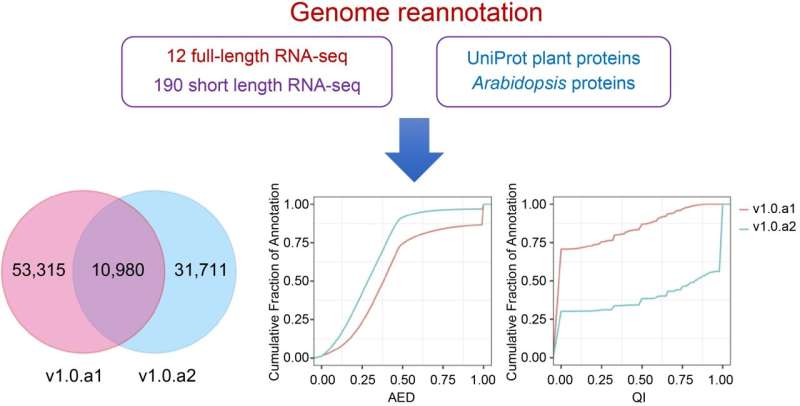
Resumo gráfico. Crédito:Plantas Tropicais (2024). DOI:10.48130/tp-0024-0009 Uma equipa de investigação melhorou substancialmente a anotação do genoma da batata-doce "Taizhong 6", introduzindo uma versão mais abrangente e detalhada, v1.0.a2. Esta atualização utiliza 12 bibliotecas de RNA completas Nanopore e 190 bibliotecas Illumina RNA-seq, resultando na identificação de 360 novos genes, modificação ou adição de 31.771 modelos de genes e nomenclatura genética refinada.
A anotação melhorada inclui perfis detalhados de expressão de miRNA, beneficiando estudos funcionais de genes na batata-doce e avançando análises genómicas em toda a família Convolvulaceae, proporcionando assim um recurso crítico para futuras pesquisas agrícolas e genéticas.
A batata-doce (Ipomoea batatas) é uma cultura vital nos países em desenvolvimento, fornecendo nutrientes essenciais e combatendo a deficiência de vitamina A em África como espécie hexaplóide. A primeira sequência do genoma, cultivar Taizhong 6, utilizou o sequenciamento Illumina, posteriormente aprimorado com tecnologias de sequenciamento de terceira geração, levando a uma montagem do genoma de alta qualidade em escala cromossômica. No entanto, permanecem desafios na obtenção de anotações precisas do genoma devido às limitações do sequenciamento de leitura curta.
Recentemente, avanços no sequenciamento de leitura longa, como Oxford Nanopore Technologies, facilitaram anotações mais precisas e permitiram insights mais profundos sobre a estrutura genética e o splicing alternativo.
Um estudo publicado em Tropical Plants em 21 de Março de 2024, baseia-se neste progresso usando um pipeline de anotação refinado que incorporou conjuntos de dados completos e extensos de RNA-seq Nanopore, melhorando a actual anotação do genoma da batata-doce.
Neste estudo, os investigadores melhoraram a anotação do genoma de I. batata para a versão 1.0.a2, utilizando uma abordagem abrangente que integra transcriptomas completos Nanopore e dados Illumina RNA-seq em vários estágios de desenvolvimento e tecidos de batata-doce.
Seu método empregou o BRAKER para previsões genéticas iniciais, enriquecido por várias dicas genômicas, seguido pela geração de modelo de consenso através do EVidenceModeler (EVM). Notavelmente, a anotação atualizada agora contém 42.751 genes codificadores de proteínas, aprimorando o modelo com UTRs de 3' e 5' e aumentando a contagem média de exon por gene.
Significativamente, esta revisão adicionou ou modificou 31.771 modelos genéticos, incorporando 8.736 isoformas de splicing alternativas, e introduziu uma nova nomenclatura genética para referência mais clara. Esta anotação mais detalhada ajuda em estudos genómicos precisos e apoia a genómica funcional avançada na batata-doce.
Além disso, a integração dos dados do miRNA e dos seus alvos oferece novos conhecimentos sobre a regulação genética, particularmente durante os diferentes estágios de desenvolvimento das raízes de armazenamento, melhorando a nossa compreensão da biologia da batata-doce e auxiliando nos esforços de melhoramento direccionado. As previsões abrangentes da função genética foram executadas usando o InterProScan e o mapeador eggNOG, fornecendo uma anotação mais rica, crucial para a investigação em curso e programas de melhoramento focados na melhoria de cultivares de batata-doce para a agricultura global.
De acordo com o investigador principal do estudo, Prof. Guopeng Zhu, "O nosso estudo contribui para uma anotação atualizada do genoma da batata-doce, o que facilitará significativamente os estudos funcionais dos genes na batata-doce e promoverá análises genómicas em toda a família Convolvulaceae."
No geral, esta estrutura genómica melhorada facilita uma genómica funcional mais profunda na batata-doce e apoia programas avançados de melhoramento, integrando dados detalhados de miRNA e previsões da função genética para melhorar as características da cultivar.
