Equipe desenvolve sistema hospedeiro-vetor eficiente para um modelo Archaeon, resolvendo conflito hospedeiro-plasmídeo baseado em CRISPR
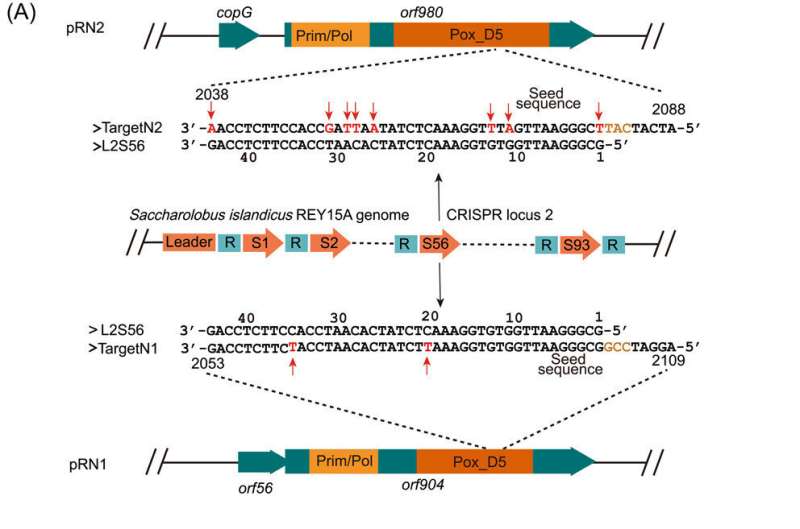 Restrição de plasmídeos derivados de pRN1 por sistemas CRISPR – Cas em Saccharolobus islandicus REY15A. (A) Alinhamento de sequência do Alvo N1/Alvo N2 e L2S56. L2S56, espaçador 56 no locus 2 CRISPR do genoma hospedeiro; Alvo N1, a sequência pRN1 correspondente ao espaçador L2S56; Alvo N2, a sequência pRN2 mostrando semelhança com o espaçador L2S56. As incompatibilidades são destacadas em vermelho e indicadas com setas vermelhas. Os trinucleotídeos na posição PAM no alvo N1 e na posição do motivo não PAM no alvo N2 estão destacados em marrom. (B) Esquema dos plasmídeos de interferência. O protoespaçador L2S56 artificial (L2S56) e o alvo N1 contêm a sequência flanqueadora 5 '5'-AGGATCCG-3' do alvo pRN1. (C) Testando a imunidade CRISPR usando dois conjuntos de plasmídeos de interferência em REY15A. Foram utilizadas quatro estirpes para transformação, incluindo as seguintes:E233S, o hospedeiro de tipo selvagem; ΔIA, derivado de E233S, sem o cas módulo genético de IA CRISPR-Cas; ΔαΔβ, derivado de E233S sem os módulos Cmr-α e Cmr-β de cas genes; e ΔαΔβΔarray, derivado de ΔαΔβ com ambas as matrizes CRISPR também excluídas. PAM, motivo adjacente do protoespaçador; PFS, sequência flanqueadora do protoespaçador. Crédito:mLife (2024). DOI:10.1002/mlf2.12107
Restrição de plasmídeos derivados de pRN1 por sistemas CRISPR – Cas em Saccharolobus islandicus REY15A. (A) Alinhamento de sequência do Alvo N1/Alvo N2 e L2S56. L2S56, espaçador 56 no locus 2 CRISPR do genoma hospedeiro; Alvo N1, a sequência pRN1 correspondente ao espaçador L2S56; Alvo N2, a sequência pRN2 mostrando semelhança com o espaçador L2S56. As incompatibilidades são destacadas em vermelho e indicadas com setas vermelhas. Os trinucleotídeos na posição PAM no alvo N1 e na posição do motivo não PAM no alvo N2 estão destacados em marrom. (B) Esquema dos plasmídeos de interferência. O protoespaçador L2S56 artificial (L2S56) e o alvo N1 contêm a sequência flanqueadora 5 '5'-AGGATCCG-3' do alvo pRN1. (C) Testando a imunidade CRISPR usando dois conjuntos de plasmídeos de interferência em REY15A. Foram utilizadas quatro estirpes para transformação, incluindo as seguintes:E233S, o hospedeiro de tipo selvagem; ΔIA, derivado de E233S, sem o cas módulo genético de IA CRISPR-Cas; ΔαΔβ, derivado de E233S sem os módulos Cmr-α e Cmr-β de cas genes; e ΔαΔβΔarray, derivado de ΔαΔβ com ambas as matrizes CRISPR também excluídas. PAM, motivo adjacente do protoespaçador; PFS, sequência flanqueadora do protoespaçador. Crédito:mLife (2024). DOI:10.1002/mlf2.12107 Um grupo de pesquisa construiu ferramentas genéticas versáteis para Saccharolobus islandicus REY15A, um dos poucos modelos de arqueas para biologia de arqueas e pesquisa em biologia CRISPR.
Essas ferramentas incluem edição eficiente de genoma, sistemas robustos de expressão de proteínas, ensaio de plasmídeos de interferência, silenciamento de genes e edição de genes baseada em CRISPR. No entanto, os vetores plasmídicos construídos para este crenarchaeon até agora são baseados apenas no plasmídeo críptico pRN2.
O estudo, publicado na mLife , foi liderado pelo Prof. Qunxin She e pelo Dr. Guanhua Yuan, ambos da Universidade de Shandong em Qingdao, China.
“Um sistema duplo hospedeiro-vetor é necessário para enriquecer a caixa de ferramentas genéticas para este modelo arqueon”, diz a Prof.
Na verdade, numa fase inicial do desenvolvimento do vector archaeal, tanto os plasmídeos pRN1 como pRN2 que coexistem no Sa. islandicus REN1H1 foram empregados para a construção de vetores de transporte para Sa. islandicus REY15A baseado nestes dois plasmídeos; Os plasmídeos derivados de pRN2 obtiveram uma alta taxa de transformação e produziram transformantes verdadeiros, enquanto os vetores baseados em pRN1 produziram apenas muito poucas colônias nas quais os plasmídeos estavam aparentemente ausentes.
"Como as matrizes CRISPR geralmente carregam espaçadores que correspondem a vários plasmídeos em Sulfolobales, suspeitamos que o genoma de Sa. islandicus REY15A possa conter um espaçador que corresponda a uma sequência em pRN1, mas não em pRN2", diz o Dr.
Depois de determinar a sequência completa do genoma de Sa. islandicus REY15A4, o genoma do hospedeiro carrega um espaçador (L2S56) mostrando apenas duas incompatibilidades com um segmento de DNA (Alvo N1) na sequência de codificação do gene da replicase pRN1. Experimentos de eficiência de transformação demonstraram que os crRNAs L2S56 foram expressos a um nível que poderia ser suficiente para induzir a imunidade I-A, mas insuficiente para desencadear a imunidade III-B para a eliminação do plasmídeo em Sa. ilhaicus REY15A.
Para obter um derivado N1 alvo funcional que escapa à imunidade CRISPR do hospedeiro, a equipe projetou três segmentos de DNA (N1a, N1b e N1c) com base no alvo pRN1, enquanto as mutações projetadas em N1a eram sinônimas, N1b e N1c tinham mutações missense. Os resultados mostraram que nenhum dos três alvos mutados foi alvo do sistema CRISPR no hospedeiro arqueal. No entanto, experiências subsequentes mostraram que N1c transporta mutações missense que podem ter inativado a proteína de replicação.
Através da construção de uma série de vetores, o Saccharolobus –E. coli pN1dAA com as mutações N1a, marcador de seleção argD, origem de replicação p15A e um marcador resistente à canamicina foram projetados com base no esqueleto pRN1, que pode alcançar uma coexistência estável com o plasmídeo pSeSD derivado de pRN2 em Sa. células REY15A de Islandicus. Isto produziu um sistema de plasmídeo duplo para estudo genético com este importante modelo arqueal.
Uma vez que o exame dos conflitos hospedeiro-plasmídeo fornece um meio útil para a identificação de sistemas plasmídeo-vetor compatíveis, uma vez que o conflito é resolvido experimentalmente, os plasmídeos projetados são então muito úteis para o desenvolvimento de sistemas hospedeiro-vetor, conforme relatado neste artigo.
