Perfeição do pêssego:redes genéticas avançadas revelam características das frutas
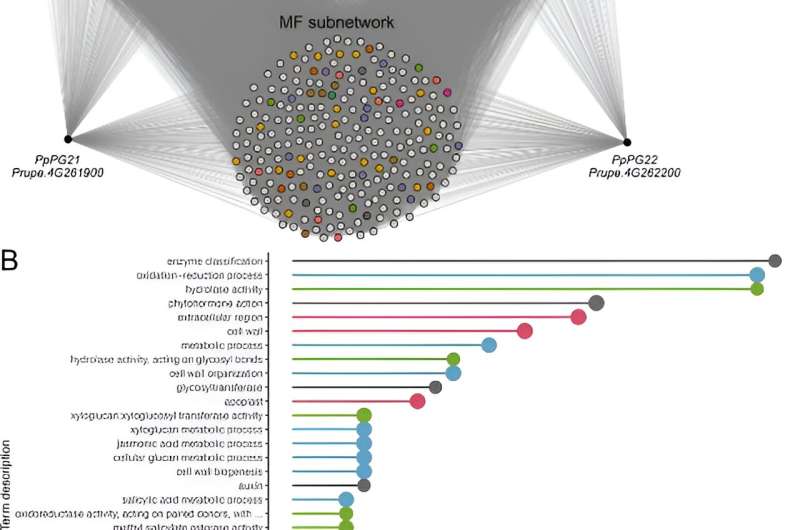 Análise de rede MF. (A) PpPG21 e PpPG22 GCNs e a rede MF. Processos biológicos de interesse são destacados na rede MF. TF:fator de transcrição, CYP450:citocromo P450. (B) Análise de enriquecimento de conjunto de genes da rede MF. Crédito:Pesquisa em Horticultura
Análise de rede MF. (A) PpPG21 e PpPG22 GCNs e a rede MF. Processos biológicos de interesse são destacados na rede MF. TF:fator de transcrição, CYP450:citocromo P450. (B) Análise de enriquecimento de conjunto de genes da rede MF. Crédito:Pesquisa em Horticultura Um estudo explorou o genoma do pêssego, criando redes de coexpressão genética (GCNs) em grande escala que prevêem funções genéticas e agilizam o processo de melhoramento do pêssego. Esta abordagem inovadora aborda a complexa tarefa de identificar genes ligados a características reprodutivas desejáveis em pêssegos.
A genética do pêssego enfrenta desafios como longos tempos de intergeração e sistemas de transformação genética limitados, tornando complexa a identificação e validação de genes. As abordagens tradicionais muitas vezes ficam aquém da previsão das funções genéticas devido a essas limitações.
As redes de coexpressão gênica (GCNs), que mapeiam as relações entre as expressões gênicas, oferecem uma solução promissora. Ao aproveitar o princípio “culpado por associação”, os GCNs podem prever funções genéticas com base em padrões de expressão compartilhados. Devido a estes desafios, existe uma necessidade premente de ferramentas inovadoras como os GCNs para melhorar a nossa compreensão da genética do pêssego.
Pesquisadores do Institut de Recerca i Tecnologia Agroalimentàries (IRTA) e do Centre de Recerca en Agrigenòmica (CRAG) publicaram um estudo na revista Horticulture Research em 2 de janeiro de 2024. O estudo apresenta uma nova ferramenta que aproveita os GCNs para prever funções genéticas em pêssegos, revolucionando potencialmente o melhoramento de frutas.
O estudo criou quatro GCNs a partir de 604 bibliotecas Illumina RNA-Seq e avaliou seu desempenho usando um algoritmo de aprendizado de máquina baseado no princípio “culpado por associação”. Dentre as redes, a COO300 apresentou o melhor desempenho, abrangendo 21.956 genes. Para validar o COO300, os pesquisadores realizaram dois estudos de caso.
No primeiro estudo de caso foram analisados os genes PpPG21 e PpPG22, envolvidos no amaciamento do pêssego. Os genes co-expressos formaram a rede melting flesh (MF), que foi enriquecida com termos relacionados à organização da parede celular e sinalização de fitohormônios. A rede MF incluiu genes associados à desmontagem da parede celular e fitohormônios relacionados ao amadurecimento, demonstrando a capacidade do COO300 de prever com precisão as funções dos genes.
No segundo estudo de caso, foi examinado o fator de transcrição PpMYB10.1, que regula o acúmulo de antocianinas. A rede de cor da fruta (FC) identificou genes envolvidos no metabolismo e regulação das antocianinas. Ao comparar a rede FC com ortólogos de videira, os pesquisadores identificaram redes regulatórias conservadas, validando ainda mais as capacidades preditivas do COO300.
Iban Eduardo, pesquisador líder do CRAG, afirmou:"O desenvolvimento da rede COO300 representa um avanço significativo na genética do pêssego. Ao prever com precisão as funções dos genes, esta ferramenta não apenas melhora nossa compreensão da biologia do pêssego, mas também abre caminho para estratégias de reprodução mais direcionadas e eficientes."
O estudo oferece aos criadores uma ferramenta poderosa para melhorar as variedades de pêssego. Ao decodificar as funções dos genes, espera-se que o PeachGCN v1.0 e seus scripts, acessíveis a todos, impulsionem avanços no sabor, longevidade e nutrição dos pêssegos. Esta visão genómica é um prenúncio de mudança, sinalizando uma mudança para a reprodução baseada em dados na agricultura.
