Uma maneira melhor de encontrar agulhas de vírus de RNA em palheiros de banco de dados
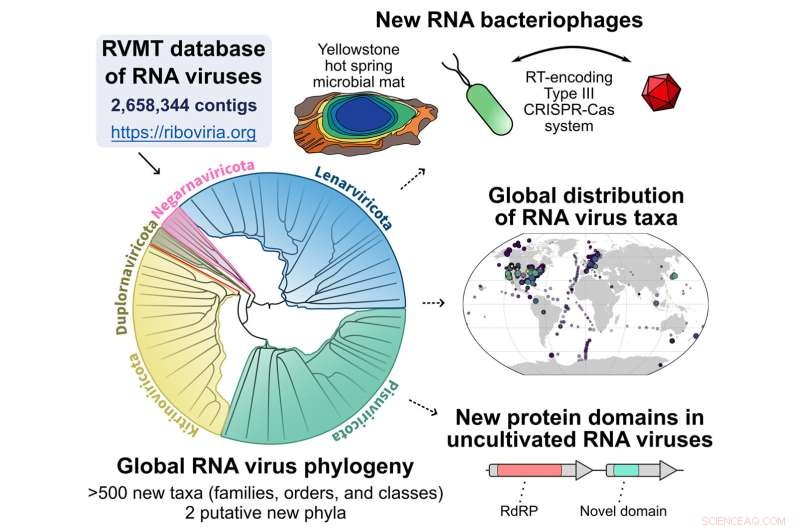
Visão geral gráfica do pipeline começando com o banco de dados RNA Virus MetaTranscriptomes (RVMT) para descobrir a expansão da diversidade de vírus RNA. Crédito:Simon Roux
Certa vez, um zoológico ofereceu um livro de colorir com ursos polares em cenas de inverno que vinham com giz de cera em vários tons de branco. Para pesquisadores que procuram sequências de vírus de RNA em grandes conjuntos de dados, seu trabalho pode ser semelhante a encontrar um único floco de neve em uma página colorida desse livro.
Publicado on-line em 28 de setembro de 2022, em
Cell , uma equipe liderada por pesquisadores da Universidade de Tel Aviv em Israel, do Centro Nacional de Informações sobre Biotecnologia e do Departamento de Energia dos EUA (DOE) Joint Genome Institute (JGI), uma instalação do DOE Office of Science User Facility localizada no Lawrence Berkeley National Laboratory ( Berkeley Lab) descrevem um pipeline computacional que pode verificar especificamente esses flocos de neve ou sequências de vírus de RNA. Usando esse fluxo de trabalho, a equipe vasculhou mais de 5.000 conjuntos de dados de sequências de RNA (metatranscriptomas) gerados a partir de diversas amostras ambientais em todo o mundo, resultando em um aumento de cinco vezes na diversidade de vírus de RNA.
"O mundo dos vírus ao nosso redor é vasto e agora temos os meios para explorá-lo", disse Eugene Koonin, pesquisador sênior do NCBI e um dos autores principais do artigo sobre a diversidade viral descoberta. "Embora os desafios técnicos da análise de dados nessa escala sejam formidáveis".
Peneiras computacionais para filtrar sequências Há mais micróbios no planeta do que partículas em um punhado de sujeira, e os vírus superam amplamente os micróbios. Avanços nas tecnologias de sequenciamento e ferramentas computacionais revelaram uma diversidade de vírus que infectam não apenas culturas, animais e humanos, mas também micróbios cuja presença ou ausência pode afetar os ciclos de nutrientes do planeta.
Enquanto a maioria das informações genéticas do organismo é codificada no DNA, com o RNA entregando as instruções dentro do DNA para a célula, os vírus de RNA armazenam suas informações genéticas no RNA sem um estágio de DNA. "Eu diria que os vírus de RNA globalmente são ainda menos conhecidos do que os vírus de DNA", disse Simon Roux, cientista do JGI e um dos co-líderes do projeto. “Mas, assim como os vírus de DNA, os vírus de RNA infectam micróbios em todo o mundo e levam à morte celular e/ou mudanças profundas na fisiologia celular durante a infecção”.
Embora todos os vírus de RNA tenham um gene que codifica uma enzima chamada RNA polimerase dirigida por RNS (RdRP), necessária para replicar a replicação do genoma do RNA, detectá-la tem sido um desafio. Encontrar os flocos de neve do vírus RNA na tempestade de dados genômicos envolveu o desenvolvimento de peneiras computacionais especiais para filtrar sequências que provavelmente não continham a sequência RdRP.
O trabalho resultou de uma colaboração de três vias que começou em 2019, lembrou Uri Neri, da Universidade de Tel Aviv, um dos co-líderes do projeto e primeiro autor do estudo. Membros das equipes de Tel Aviv e NCBI, que já estavam trabalhando juntos na mineração de vírus procarióticos, aprenderam com Nikos Kyrpides, da JGI, que seu grupo de Ciência de Dados de Microbiomas também estava trabalhando na mineração de vírus RNA. Após algumas reuniões virtuais das três equipes, ficou claro que um esforço colaborativo maior seria muito mais eficaz na obtenção de resultados de maior qualidade em comparação com esforços individuais menores. Este é também o tipo de espírito comunitário sinérgico e colaborativo que o JGI defende e promove ativamente.
A equipe usou todos os conjuntos de dados de metatranscriptomas publicamente disponíveis do sistema Integrated Microbial Genomes &Microbiomes (IMG/M) do JGI. "Depois analisamos muitas outras amostras e refinamos nossa metodologia", disse Neri. "Nossa equipe cresceu e o escopo do projeto também." Para esse fim, enfatizou Kyrpides, as contribuições dos numerosos usuários da ciência do JGI na coleta e envio de suas amostras de microbioma para sequenciamento no JGI não podem ser exageradas. Sua cooperação e apoio, disse ele, e em vários casos, sua permissão para usar dados de sequência ainda não publicados, foram absolutamente críticos para o sucesso desse esforço, assim como o reconhecimento de sua contribuição.
Tanto Roux quanto Koonin observaram que a infinidade de sequências de vírus de RNA descobertas "muda significativamente a visão global da diversidade de vírus", embora não nas classificações de nível superior de grupos de vírus (filos). As novas sequências estão preenchendo algumas lacunas nos vírus existentes grupos enquanto também adiciona novas ramificações. Além disso, os vírus de RNA não parecem estar distribuídos uniformemente pelo mundo.
Um grupo expandido é de vírus associados a bactérias; até agora, a maioria dos vírus de RNA conhecidos foram associados a eucariotos. Junto com a expansão dos vírus de RNA associados a bactérias está a descoberta de que "algumas bactérias usam o CRISPR para se defender contra o RNA", observou Roux, "embora não esteja claro por que isso é tão raramente detectado".
Desenvolver abordagens para reconciliar Big Data 'real' Para a equipe, o trabalho computacional que levou à descoberta da abundância de vírus de RNA é apenas o começo. "Costumo dizer que apenas identificar uma sequência como viral não é nem metade da história." disse Neri. "Investimos muito de nossos esforços nas análises pós-descoberta - da melhor maneira possível, tentamos descrever os domínios de proteína que cada vírus carrega e quem é seu provável hospedeiro. Tornamos todas essas informações totalmente gratuitas e abertamente disponível para a comunidade científica mais ampla."
Uri Gophna, da Universidade de Tel Aviv, e Koonin observaram que outras pesquisas paralelas relataram "expansões dramáticas" semelhantes do viroma de RNA global. "Agora precisamos comparar e reconciliar as descobertas, chegando a um único conjunto de dados não redundante", disse Koonin. "Espero que em breve possamos estimar o tamanho real do vírus de RNA. No entanto, isso agora é Big Data real, estamos lidando com bilhões de sequências e, em breve, com trilhões. O desenvolvimento de abordagens eficientes e automatizadas para analisar e classificar dados de sequência nessa escala é essencial."
+ Explorar mais Uma ferramenta automatizada para avaliar a qualidade dos dados de vírus
