Resumo gráfico. Crédito:Célula Molecular (2022). DOI:10.1016/j.molcel.2022.06.023
Usando uma nova técnica inovadora, cientistas da Duke-NUS Medical School e seus colaboradores identificaram milhares de sequências de DNA anteriormente desconhecidas no genoma humano que codificam microproteínas e peptídeos potencialmente críticos para a saúde e doenças humanas.
"Muito do que entendemos sobre os 2% conhecidos do genoma que codifica proteínas vem da busca por longas cadeias de sequências de nucleotídeos codificadoras de proteínas, ou longos quadros de leitura abertos", explicou a bióloga computacional Dr. Sonia Chothani, pesquisadora da Programa de Distúrbios Cardiovasculares e Metabólicos (CVMD) da Duke-NUS e primeiro autor do estudo. "Recentemente, no entanto, os cientistas descobriram pequenos quadros de leitura aberta (smORFs) que também podem ser traduzidos do RNA em pequenos peptídeos, que têm papéis no reparo do DNA, formação muscular e regulação genética".
Os cientistas têm tentado identificar smORFs e os pequenos peptídeos que codificam, uma vez que a interrupção desses smORFs pode causar doenças. No entanto, as abordagens atualmente disponíveis são muito limitadas.
"Muitos dos conjuntos de dados atuais não fornecem informações detalhadas o suficiente para identificar smORFs no RNA", acrescentou Dr. Chothani. "A maioria também vem de análises de células humanas imortalizadas que são propagadas - às vezes por décadas - para estudar a fisiologia, função e doença das células. No entanto, essas linhagens de células nem sempre são representações precisas da fisiologia humana".
Publicando em
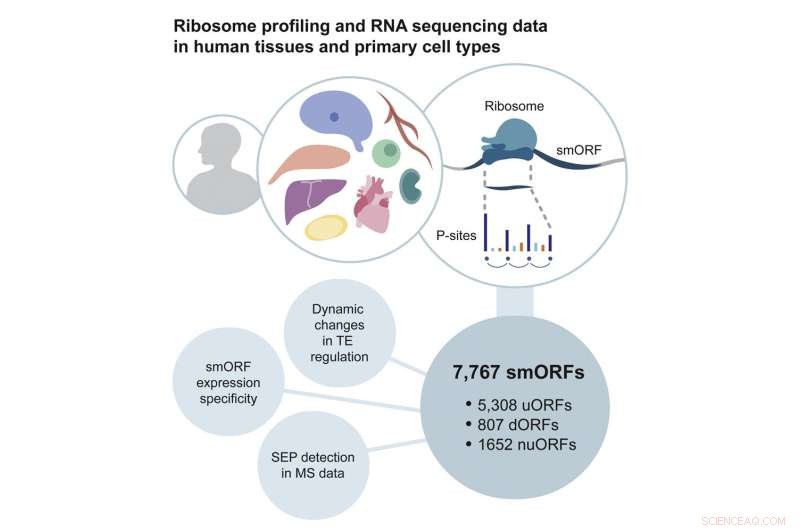
Célula Molecular , Chothani e seus colegas em Cingapura, Alemanha, Reino Unido e Austrália descrevem uma metodologia que desenvolveram para abordar esses problemas. Eles rastrearam conjuntos de dados de perfis de ribossomos atualmente disponíveis para fitas curtas de RNA com seções periódicas de três bases, cobrindo mais de 60% do comprimento do RNA. Eles então conduziram seu próprio sequenciamento de RNA e perfil de ribossomo para gerar um recurso de dados combinado de seis tipos de células e cinco tipos de tecido, como do coração e do cérebro, derivados de centenas de pacientes.
As análises desses dados identificaram cerca de 8.000 smORFs. Curiosamente, eles eram altamente específicos para os tecidos em que foram encontrados, o que significa que esses smORFs podem desempenhar uma função específica para seu ambiente. A equipe também identificou 603 microproteínas codificadas por alguns desses smORFs.
"O genoma está repleto de smORFs", disse o professor assistente Owen Rackham, autor sênior do estudo do Programa CVMD. “Nosso mapa abrangente e espacialmente resolvido de smORFs humanos destaca componentes funcionais negligenciados do genoma, identifica novos participantes na saúde e na doença e fornece um recurso para a comunidade científica como uma plataforma para acelerar as descobertas”.
O professor Patrick Casey, vice-reitor sênior de pesquisa da Duke-NUS, disse:"Com o sistema de saúde evoluindo para não apenas tratar doenças, mas também preveni-las, identificar novos alvos potenciais para pesquisa de doenças e desenvolvimento de medicamentos pode abrir caminhos para novas soluções. Esta pesquisa do Dr. Chothani e sua equipe, publicada como um recurso para a comunidade científica, traz importantes insights para o campo."
+ Explorar mais Encontrar os menores genes pode trazer grandes benefícios
