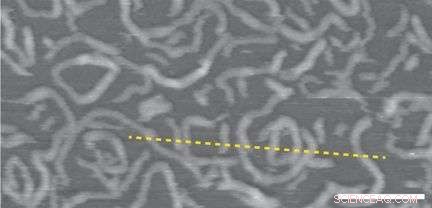
A topografia e as medidas de altura de poli (APPV-DNA). Crédito: Nature Nanotechnology , DOI:10.1038 / NNANO.2015.190
(Phys.org) —Nova pesquisa permite aos cientistas esculpir polímeros em formas bidimensionais e tridimensionais, semelhante a como os polipeptídeos se dobram em formas tridimensionais funcionais. Esta capacidade é particularmente vantajosa para polímeros conjugados, polímeros que têm um sistema de elétrons-pi em rede, porque eles estão conduzindo. Imobilizar e moldar polímeros condutores é uma etapa importante na construção de circuitos moleculares.
Um grupo de cientistas da Universidade Aarhus, na Dinamarca, o Wyss Institute em Harvard, e o Instituto Max Plank na Alemanha, sintetizaram, caracterizado, e imobilizou um polímero conjugado usando origami de DNA. Seu polímero foi capaz de ser moldado e moldado em várias formas bidimensionais e tridimensionais, mantendo suas propriedades físicas. O trabalho deles aparece em Nature Nanotechnology .
Knudsen, et al. sintetizou um polímero de escova conjugado, (2, 5-dialcoxi) parafenileno vinileno (APPV) que é funcionalizado com sequências de DNA de fita simples (ssDNA) de nove nucleotídeos para servir de ligação ao origami de DNA. APPV possui grupos hidroxila ao longo de sua estrutura que estão ligados a unidades de fenileno. Esses grupos hidroxila estão disponíveis para funcionalização com ssDNA sintético.
O ssDNA recozido com fitas complementares que se estendem a partir de uma plataforma de DNA, mantendo assim o polímero no lugar. Essa técnica é conhecida como origami de DNA porque as fitas complementares de DNA que se estendem a partir do origami de DNA podem ser adaptadas a qualquer formato ou desenho e devem guiar o polímero com seu ssDNA complementar para assumir a mesma forma.
Neste experimento, APPV-DNA foi caracterizado por cromatografia de permeação em gel, Espectroscopia UV-Vis, espectroscopia de fluorescência, XPS, e AFM. A cromatografia de permeação em gel mostrou que o tamanho do polímero variou de 340 kDa a 3, 300 kDa. Este e os estudos de AFM indicaram a presença de pedaços de polímero menores e mais longos. O XPS mostrou que menos de dois terços das unidades de fenileno, contendo grupos hidroxila, onde funcionalizado com ssDNA. Adicionalmente, Os estudos AFM forneceram informações sobre o potencial de superfície, indicando que o polímero APPV-DNA tem uma transferência de carga maior do que o substrato de óxido de silício, mas menor do que ouro ou nanotubos de carbono.
O polímero foi então imobilizado em origami de DNA em várias formas bidimensionais e tridimensionais, e a transferência de carga, bem como a integridade do polímero foram testadas. O primeiro teste envolveu origami de DNA linear, Em forma de U, e aos 90 o ângulos. Estudos de potencial de superfície indicaram que o polímero APPV-DNA imobilizado exibiu habilidades de transferência de carga semelhantes em todas as conformações. A flexibilidade do polímero foi verificada submetendo-o a formas de origami de DNA que esticariam a estrutura:onda, Escadaria, e circular.
Finalmente, o polímero APPV-DNA foi formado em uma estrutura cilíndrica tridimensional feita de anéis empilhados de hélices duplas. Os anéis empilhados são mantidos juntos usando fios de grampo. Estudos TEM confirmaram a forma do cilindro, mas o polímero não fornece contraste adequado para caracterização completa usando TEM. AFM ou qualquer outra técnica de microscopia de varredura também não funcionará para este tipo de estrutura. A interação entre a ponta e a molécula pode danificar a estrutura tridimensional "macia" do polímero.
A fim de obter uma representação tridimensional do cilindro APPV-DNA, Knudsen, et al. usado DNA-PAINT. Usando o excesso de ssDNA de nove nucleotídeos que não se ligou à estrutura de origami do DNA, Knudsen, et al. fez fitas complementares com um rótulo fluorescente. Eles então usaram o DNA-PAINT para visualizar o padrão da fita e renderizar uma imagem tridimensional.
Esta pesquisa demonstra a capacidade de controlar a conformação bidimensional e tridimensional de um polímero conjugado, que tem implicações promissoras para o projeto de circuitos moleculares.
© 2015 Phys.org