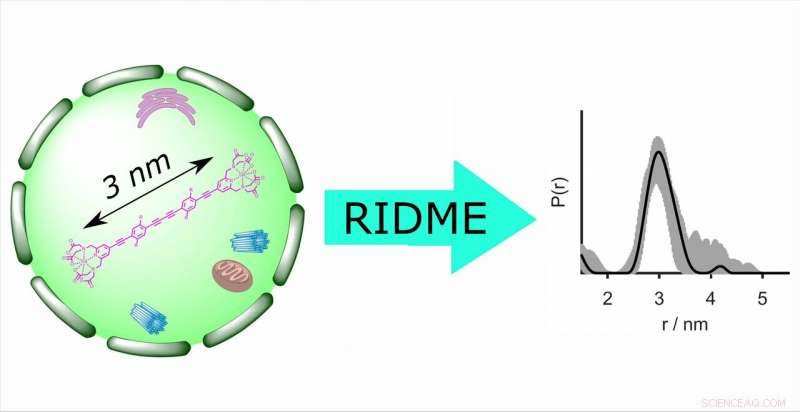
A determinação da distância na célula por EPR revela informações estruturais essenciais sobre biomacromoléculas sob condições nativas. Pela primeira vez, a técnica de EPR pulsado RIDME (reforço da modulação dipolar induzida por relaxamento) foi utilizada para medições de distância dentro das células. Ele fornece uma sensibilidade cinco vezes melhor em comparação com a abordagem de ressonância elétron-elétron duplo usada anteriormente. Crédito:Grupo de pesquisa Professor Malte Drescher, Universidade de Constança
Em um artigo conjunto, pesquisadores da Universidade de Konstanz, A Universidade de Bielefeld e a ETH Zurich demonstram pela primeira vez que a técnica de ressonância paramagnética eletrônica (EPR) RIDME (realce da modulação dipolar induzida por relaxamento) pode ser aplicada para determinar distâncias entre marcadores de spin baseados em gadolínio (III) nas células. A determinação da distância na célula por ressonância paramagnética eletrônica (EPR) revela informações estruturais essenciais sobre as biomacromoléculas, incluindo sua conformação, bem como processos de dobramento e desdobramento.
Métodos convencionais para determinação de distâncias na célula, como ressonância elétron-elétron dupla (DEER ou PELDOR) são principalmente muito menos sensíveis do que RIDME, fornecem profundidades de modulação até cinco vezes menores, têm certas limitações no que diz respeito à largura de banda de excitação e são tecnicamente mais exigentes. Como uma técnica de frequência única que faz uso de spin flips induzidos por relaxamento para determinar a distância entre dois rótulos de spin, ou seja, entre dois elétrons desemparelhados, O RIDME supera todas essas desvantagens.
Essa técnica permite que os pesquisadores trabalhem com moléculas em condições nativas, como Professor Malte Drescher e autor principal, Dr. Mykhailo Azarkh, ambos da Universidade de Konstanz, enfatizam:“Começamos analisando a conformação de uma proteína dentro da célula. Com técnicas menos sensíveis, somos forçados a inserir e marcar uma grande quantidade de proteínas para poder observá-la, o que não é absolutamente o que acontece na natureza. Idealmente, queremos trabalhar com concentrações fisiologicamente relevantes. Uma vez que RIDME é muito mais sensível do que DEER, permite-nos fazer exatamente isso. Estamos agora em condições de resolver questões que de outra forma não seríamos capazes de resolver ".
O desempenho do RIDME na célula foi avaliado na banda Q usando réguas moleculares rígidas marcadas com Gd (III) -PyMTA e microjuntas em oócitos de Xenopus laevis (sapo africano com garras). Em outras palavras, os pesquisadores usaram um sistema de modelo onde a distância precisa entre os rótulos de spin já era conhecida, permitindo-lhes verificar as medições RIDME. O artigo resultante, intitulado "Gd (III) –Gd (III) Aumento da Modulação Dipolar Induzida por Relaxamento para Determinação da Distância de Ressonância Paramagnética Eletrônica In-Cell" foi publicado online no Journal of Physical Chemistry Letters em 13.03.2019.
A determinação da distância RIDME in-cell foi desenvolvida e testada como parte do projeto em andamento financiado pelo ERC "SPICE - Espectroscopia em células", para o qual Malte Drescher, Professor Heisenberg de Espectroscopia de Sistemas Complexos da Universidade de Konstanz, e sua equipe de pesquisa foi reconhecida com uma bolsa ERC Consolidator no valor de aproximadamente dois milhões de euros em 2017. Seu objetivo é desenvolver novas abordagens para espectroscopia que lhes permitam explorar estruturas biológicas maiores e mais complexas no nível molecular da célula.
O próximo passo nesta linha de pesquisa será identificar outros marcadores de spin adequados e desenvolver RIDME para aplicação em moléculas onde a distância entre os marcadores de spin é desconhecida. Um foco particular de atenção estará nas moléculas associadas a doenças neurodegenerativas, como Alzheimer e Parkinson.