IA decifra novo código regulador de genes em plantas e faz previsões precisas para genomas recém-sequenciados
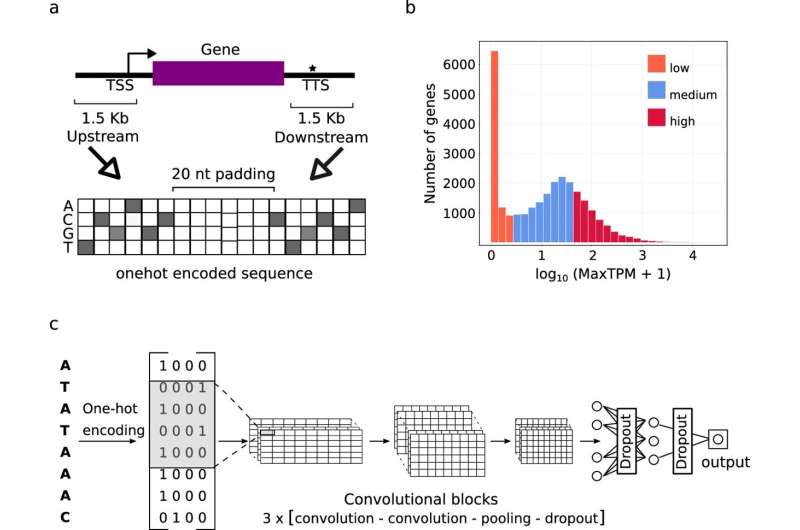 Os modelos de predição de expressão gênica exigiram a extração da sequência gênica proximal dos genomas de referência das plantas cultivadas, estimativa e classificação dos níveis de transcrição e conversão da sequência nucleotídica por meio de codificação one-hot para gerar dados de treinamento para a modelagem em uma rede neural convolucional. Crédito:Nature Communications (2024). DOI:10.1038/s41467-024-47744-0
Os modelos de predição de expressão gênica exigiram a extração da sequência gênica proximal dos genomas de referência das plantas cultivadas, estimativa e classificação dos níveis de transcrição e conversão da sequência nucleotídica por meio de codificação one-hot para gerar dados de treinamento para a modelagem em uma rede neural convolucional. Crédito:Nature Communications (2024). DOI:10.1038/s41467-024-47744-0 A tecnologia de sequenciamento de genoma fornece milhares de novos genomas de plantas anualmente. Na agricultura, os investigadores fundem esta informação genómica com dados observacionais (medindo várias características das plantas) para identificar correlações entre variantes genéticas e características das culturas, como contagem de sementes, resistência a infecções fúngicas, cor ou sabor dos frutos.
No entanto, a compreensão de como a variação genética influencia a atividade genética a nível molecular é bastante limitada. Esta lacuna no conhecimento dificulta a criação de “culturas inteligentes” com melhor qualidade e impacto ambiental negativo reduzido, alcançado pela combinação de variantes genéticas específicas de função conhecida.
Pesquisadores do Instituto IPK Leibniz e do Forschungszentrum Jülich (FZ) fizeram um avanço significativo para enfrentar este desafio. Jedrzej Jakub Szymanski, a equipe de pesquisa internacional treinou modelos interpretáveis de aprendizagem profunda, um subconjunto de algoritmos de IA, em um vasto conjunto de dados de informações genômicas de várias espécies de plantas.
“Esses modelos não só foram capazes de prever com precisão a atividade genética a partir de sequências, mas também identificar quais partes da sequência contribuem para essas previsões”, explica o chefe do grupo de pesquisa “Análise e Modelagem de Rede” do IPK. A tecnologia de IA aplicada pelos pesquisadores é semelhante à usada na visão computacional, que envolve o reconhecimento de características faciais em imagens e a inferência de emoções.
Em contraste com abordagens anteriores baseadas no enriquecimento estatístico, aqui os pesquisadores combinaram a identificação de características de sequência com a determinação do número de cópias de mRNA no quadro de um modelo matemático que foi treinado levando em consideração informações biológicas sobre a estrutura do modelo genético e homologia de sequência, portanto, gene evolução.
"Ficamos realmente impressionados com a eficácia. Em poucos dias de treinamento, redescobrimos muitas sequências regulatórias conhecidas e descobrimos que cerca de 50% das características identificadas eram inteiramente novas. Esses modelos generalizaram de forma excelente para espécies de plantas nas quais não foram treinados, tornando-os eles são valiosos para analisar genomas recém-sequenciados", diz o Dr. Szymanski.
"E demonstramos especificamente sua aplicação em diversos cultivares de tomate com dados de sequenciamento de leitura longa. Identificamos variações específicas na sequência reguladora que explicaram as diferenças observadas na atividade genética e, consequentemente, variações na forma, cor e robustez. Esta é uma melhoria notável em relação a associações estatísticas classicamente usadas de polimorfismos de nucleotídeo único."
A equipe compartilhou abertamente seus modelos e forneceu uma interface web para uso. "Curiosamente, muito esforço foi feito para degradar o desempenho do nosso modelo. Para evitar resultados excessivamente otimistas devido à descoberta de atalhos pela IA, foi necessário um mergulho profundo na biologia da regulação genética para eliminar qualquer viés potencial, reduzir o vazamento de dados e o overfitting", diz Fritz Forbang Peleke, o principal pesquisador de aprendizado de máquina e primeiro autor do estudo, que foi publicado na revista Nature Communications .
Simon Zumkeller, coautor e biólogo evolucionista da FZ Jülich, afirma:"Com as análises apresentadas podemos investigar e comparar a regulação genética nas plantas e inferir a sua evolução. Para aplicações práticas, o método também fornece uma nova base. Estamos nos aproximando da identificação rotineira de elementos reguladores de genes em genomas de plantas conhecidos e recentemente sequenciados, em vários tecidos e sob diferentes condições ambientais”.
