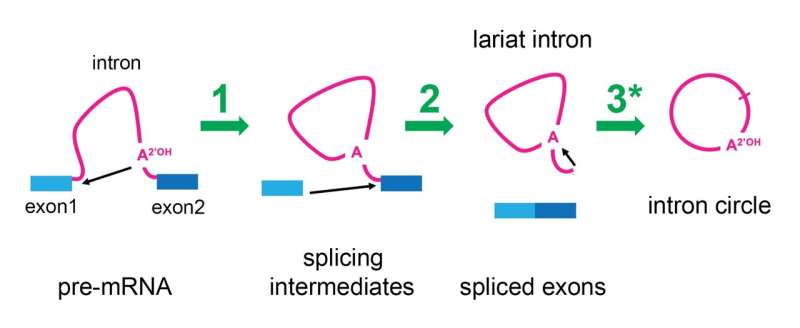
 É mostrado o caminho de emenda. O RNA pré-mensageiro (pré-mRNA) possui éxons (azul) e íntrons (rosa). O spliceossomo (não mostrado) era conhecido por catalisar duas reações químicas (setas pretas) em um processo de duas etapas (setas verdes marcadas como 1 e 2) que une os éxons e remove o íntron como um laço. Este estudo demonstra que após a conclusão do splicing, o spliceossomo ainda está ativo e pode converter o íntron do laço em um círculo usando uma terceira reação (seta verde 3) marcada por um asterisco. Crédito:Manuel Ares, UC Santa Cruz
É mostrado o caminho de emenda. O RNA pré-mensageiro (pré-mRNA) possui éxons (azul) e íntrons (rosa). O spliceossomo (não mostrado) era conhecido por catalisar duas reações químicas (setas pretas) em um processo de duas etapas (setas verdes marcadas como 1 e 2) que une os éxons e remove o íntron como um laço. Este estudo demonstra que após a conclusão do splicing, o spliceossomo ainda está ativo e pode converter o íntron do laço em um círculo usando uma terceira reação (seta verde 3) marcada por um asterisco. Crédito:Manuel Ares, UC Santa Cruz Embora você possa não apreciá-los, ou mesmo não ter ouvido falar deles, em todo o seu corpo, inúmeras máquinas microscópicas chamadas spliceossomas estão trabalhando arduamente. Enquanto você se senta e lê, eles estão juntando fiel e rapidamente as informações quebradas em seus genes, removendo sequências chamadas "íntrons" para que seus RNAs mensageiros possam produzir as proteínas corretas necessárias para suas células.
Os íntrons são talvez um dos maiores mistérios do nosso genoma. São sequências de DNA que interrompem as informações sensíveis de codificação de proteínas em seus genes e precisam ser “separadas”. O genoma humano tem centenas de milhares de íntrons, cerca de 7 ou 8 por gene, e cada um é removido por um complexo especializado de proteínas de RNA chamado "spliceossomo", que corta todos os íntrons e une as sequências codificantes restantes, chamadas éxons. Não se sabe como esse sistema de genes quebrados e o spliceossoma evoluíram em nossos genomas.
Ao longo de sua longa carreira, Manny Ares, ilustre professor de biologia molecular, celular e do desenvolvimento da UC Santa Cruz, assumiu como missão aprender o máximo que puder sobre o splicing de RNA.
“Eu adoro o spliceossomo”, disse Ares. "Eu só quero saber tudo o que o spliceossomo faz, mesmo que não saiba por que ele está fazendo isso."
Em um novo artigo publicado na revista Genes and Development , Ares relata uma descoberta surpreendente sobre o spliceossomo que pode nos dizer mais sobre a evolução de diferentes espécies e a forma como as células se adaptaram ao estranho problema dos íntrons. Os autores mostram que após o spliceossomo terminar de emendar o mRNA, ele permanece ativo e pode se envolver em reações adicionais com os íntrons removidos.
Esta descoberta fornece a indicação mais forte que temos até agora de que os spliceossomos poderiam ser capazes de reinserir um íntron de volta no genoma em outro local. Esta é uma capacidade que anteriormente não se acreditava que os spliceossomos possuíssem, mas que é uma característica comum dos "íntrons do Grupo II", primos distantes do spliceossomo que existem principalmente em bactérias.
Acredita-se que o spliceossomo e os íntrons do Grupo II compartilhem um ancestral comum que foi responsável pela disseminação dos íntrons por todo o genoma, mas enquanto os íntrons do Grupo II podem se separar do RNA e depois diretamente de volta ao DNA, os "íntrons spliceossômicos" que são encontrados em a maioria dos organismos de nível superior requer o spliceossomo para o splicing e não se acredita que sejam reinseridos no DNA. No entanto, a descoberta do laboratório de Ares indica que o spliceossomo ainda pode estar reinserindo íntrons no genoma hoje. Esta é uma possibilidade intrigante a considerar porque os intrões que são reintroduzidos no ADN acrescentam complexidade ao genoma, e compreender mais sobre a origem destes intrões poderia ajudar-nos a compreender melhor como os organismos continuam a evoluir.
Com base em uma descoberta interessante
Os genes de um organismo são feitos de DNA, no qual quatro bases, adenina (A), citosina (C), guanina (G) e timina (T) são ordenadas em sequências que codificam instruções biológicas, como como produzir proteínas específicas no corpo. precisa. Antes que essas instruções possam ser lidas, o DNA é copiado em RNA por um processo conhecido como transcrição, e então os íntrons desse RNA precisam ser removidos antes que um ribossomo possa traduzi-lo em proteínas reais.
O spliceossomo remove os íntrons usando um processo de duas etapas que resulta no RNA do íntron tendo uma de suas extremidades unida ao meio, formando um círculo com uma cauda que se parece com um "lariat" ou laço de cowboy. Essa aparência os levou a serem chamados de "íntrons lariat". Recentemente, pesquisadores da Universidade Brown que estavam estudando as localizações dos locais de união nesses laços fizeram uma observação estranha – alguns íntrons eram na verdade circulares em vez de terem formato de laço.
Esta observação chamou imediatamente a atenção de Ares. Algo parecia estar interagindo com os íntrons do laço depois que eles foram removidos da sequência de RNA para mudar sua forma, e o spliceossomo era seu principal suspeito.
“Achei isso interessante por causa dessa ideia muito antiga sobre a origem dos íntrons”, disse Ares. “Há muitas evidências de que as partes de RNA do spliceossomo, os snRNAs, estão intimamente relacionadas aos íntrons do Grupo II”.
Como o mecanismo químico de splicing é muito semelhante entre os spliceossomos e seus primos distantes, os íntrons do Grupo II, muitos pesquisadores teorizaram que, quando o processo de auto-splicing se tornou muito ineficiente para que os íntrons do Grupo II se completassem de forma confiável por conta própria, partes de esses íntrons evoluíram para se tornarem o spliceossomo. Embora os íntrons do Grupo II fossem capazes de se inserir diretamente de volta no DNA, não se pensava que os íntrons spliceossomais que exigiam a ajuda de spliceossomos fossem inseridos de volta no DNA.
“Uma das questões que estava faltando nesta história em minha mente era:é possível que o spliceossomo moderno ainda seja capaz de pegar um íntron em forma de laço e inseri-lo em algum lugar do genoma?” Ares disse. “Ainda é capaz de fazer o que o complexo ancestral fez?”
Para começar a responder a esta questão, Ares decidiu investigar se era de facto o spliceossoma que estava a fazer alterações nos intrões do laço para remover as suas caudas. Seu laboratório retardou o processo de splicing em células de levedura e descobriu que, depois que o spliceossomo liberou o mRNA do qual havia terminado o splicing dos íntrons, ele se pendurou nos laços de íntrons e os remodelou em círculos verdadeiros. O laboratório Ares foi capaz de reanalisar dados publicados de sequenciamento de RNA de células humanas e descobriu que os spliceossomos humanos também tinham essa capacidade.
“Estamos entusiasmados com isso porque, embora não saibamos o que esse RNA circular pode fazer, o fato de o spliceossomo ainda estar ativo sugere que ele pode ser capaz de catalisar a inserção do íntron lariat de volta ao genoma”, disse Ares.
Se o spliceossomo for capaz de reinserir o íntron no DNA, isso também acrescentaria um peso significativo à teoria de que os spliceossomos e os íntrons do Grupo II compartilharam um ancestral comum há muito tempo.
Testando uma teoria
Agora que Ares e seu laboratório mostraram que o spliceossomo tem a capacidade catalítica de hipoteticamente colocar íntrons de volta no DNA como seus ancestrais fizeram, o próximo passo é os pesquisadores criarem uma situação artificial na qual eles "alimentam" uma fita de DNA para um spliceossomo que ainda está ligado a um íntron em forma de laço e ver se eles conseguem realmente fazer com que ele insira o íntron em algum lugar, o que apresentaria uma "prova de conceito" para esta teoria.
Se o spliceossomo for capaz de reinserir íntrons no genoma, é provável que seja um evento muito raro em humanos, porque os spliceossomos humanos têm uma demanda incrivelmente alta e, portanto, não têm muito tempo para gastar com os íntrons removidos. Em outros organismos onde o spliceossomo não está tão ocupado, entretanto, a reinserção de íntrons pode ser mais frequente. Ares está trabalhando em estreita colaboração com o professor de engenharia biomolecular da UCSC, Russ Corbett-Detig, que recentemente liderou uma busca sistemática e exaustiva por novos íntrons nos genomas disponíveis de todas as espécies que contêm íntrons, publicada na revista Proceedings of the National Academy. de Ciências (PNAS ) ano passado.
O artigo da PNAS mostrou que eventos de “explosão” de íntrons muito atrás na história evolutiva provavelmente introduziram milhares de íntrons em um genoma de uma só vez. Ares e Corbett-Detig estão agora trabalhando para recriar artificialmente um evento de explosão, o que lhes daria informações sobre como os genomas reagiram quando isso aconteceu.
Ares disse que sua parceria interdisciplinar com a Corbett-Detig abriu as portas para que eles realmente investigassem alguns dos maiores mistérios sobre os íntrons que provavelmente seriam impossíveis de serem totalmente compreendidos sem seus conhecimentos combinados.
“É a melhor maneira de fazer as coisas”, disse Ares. "Quando você encontra alguém que tem o mesmo tipo de perguntas em mente, mas um conjunto diferente de métodos, perspectivas, preconceitos e ideias estranhas, isso fica mais emocionante. Isso faz você sentir que pode sair e resolver um problema como este, o que é muito complexo."
