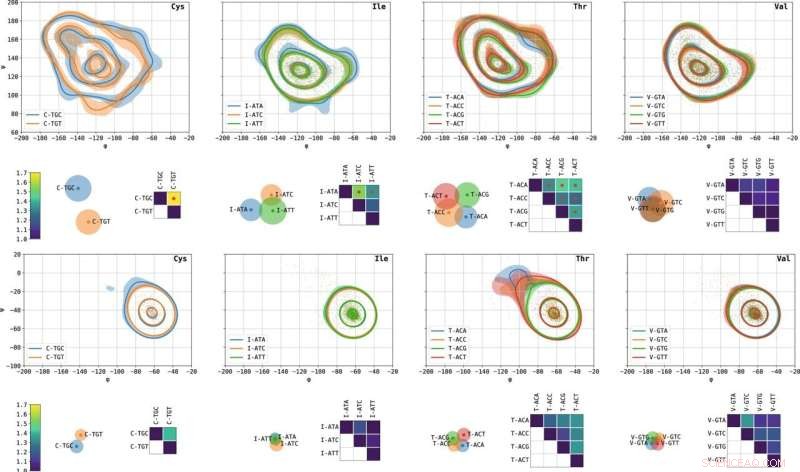
Gráficos de Ramachandran específicos de códons de aminoácidos selecionados e distâncias entre eles. Da esquerda para a direita estão cisteína, isoleucina, treonina e valina. Os gráficos de contorno representam as linhas de nível contendo 10, 50 e 90% da massa de probabilidade. As regiões sombreadas representam intervalos de confiança de 10% a 90% calculados em 1.000 bootstraps aleatórios. Os modos β- (superior) e α- (inferior) são descritos. As matrizes mostram L1 distâncias entre pares de gráficos de Ramachandran códon-específicos, normalizados para que a autodistância seja 1. Os pontos vermelhos indicam pares com distribuições de ângulo diedro significativamente diferentes com base em seu valor p. Os gráficos de dispersão que visualizam as matrizes de distância foram obtidos por uma variante de escalonamento multidimensional (MDS). Cada ponto representa um códon; distâncias euclidianas de pares entre os pontos se aproximam de L1 distância entre os códons correspondentes. Os círculos aproximam os raios de incerteza. Quanto mais dois círculos se sobrepõem, menos distinguíveis são os gráficos de Ramachandran específicos de códons correspondentes. Crédito:Comunicação da Natureza (2022). DOI:10.1038/s41467-022-30390-9
Um estudo que integra ideias biológicas e novas ferramentas da ciência da computação descobriu novas associações entre codificação genética e estrutura de proteínas, o que poderia potencialmente mudar a maneira como pensamos sobre a produção de proteínas no ribossomo - a "linha de montagem de proteínas" da célula. A pesquisa, liderada pelo professor Alex Bronstein, Dra. Ailie Marx e Ph.D. estudante Aviv Rosenberg, foi publicado em
Nature Communications .
As proteínas, as moléculas complexas que desempenham papéis críticos em praticamente todos os mecanismos biológicos, são produzidas pelos ribossomos em um processo chamado tradução. O ribossomo decodifica as "instruções genéticas" recebidas para sintetizar cadeias de aminoácidos - os blocos de construção das proteínas. Quando os aminoácidos são sequencialmente ligados em uma longa cadeia, eles se dobram em uma estrutura tridimensional única que confere à proteína suas propriedades biológicas e funcionalidade. Erros na tradução podem levar a erros de dobragem e, posteriormente, distúrbios fisiológicos, leves e graves.
As instruções de produção de proteínas são entregues ao ribossomo como códons, sequências de três "letras" do código genético de nucleotídeos, que especificam a identidade e a ordem dos aminoácidos a serem adicionados pelo ribossomo à cadeia proteica. Por exemplo, o códon UUU sinaliza para a adição do aminoácido fenilalanina, enquanto o códon UAC instrui para a adição de tirosina. Desta forma, a sequência de códons codifica a sequência única de aminoácidos característica de cada proteína. Esse mapeamento de códons genéticos para aminoácidos usados na tradução é comum a todas as criaturas vivas do planeta e é considerado um mecanismo primitivo.
Como se tudo isso não fosse complicado o suficiente, é importante ressaltar que existem 61 códons que são decodificados em apenas 20 aminoácidos. Em outras palavras, todos, exceto dois aminoácidos, são codificados por múltiplos códons.
É aqui que a presente pesquisa entra em cena. Com base em experimentos realizados nas décadas de 1960 e 1970, o dogma aceito afirma que as proteínas não carregam "memória" do códon específico do qual cada aminoácido foi traduzido, desde que a identidade do aminoácido permaneça inalterada. Esses primeiros experimentos de dobramento de proteínas usaram desnaturantes químicos para desdobrar proteínas totalmente formadas e, em seguida, demonstraram que, após a remoção desses produtos químicos, a cadeia de proteínas poderia se redobrar espontaneamente para recuperar sua estrutura e função originais. Esses experimentos sugeriram que apenas a sequência de aminoácidos, e não a sequência de códons específica, determina a estrutura de uma proteína. Em vista desse dogma, mutações que alteram a codificação genética sem alterar o aminoácido são amplamente denominadas como "silenciosas" e consideradas inconsequentes para a estrutura e função das proteínas.
A equipe de pesquisa do Technion descobriu uma associação entre a identidade do códon e a estrutura local da proteína traduzida, o que sugere que esse pode não ser o caso geral e que as proteínas podem de fato "lembrar" as instruções específicas das quais foram sintetizadas. A equipe de pesquisa analisou milhares de estruturas tridimensionais de proteínas usando ferramentas dedicadas que desenvolveram, que integram métodos avançados de ciência da computação, aprendizado de máquina e estatística. Dessa forma, eles compararam com precisão as distribuições de ângulos formados nessas estruturas sob diferentes códigos genéticos sinônimos. Suas descobertas mostram que, para certos códons, há uma dependência estatística significativa entre a identidade do códon e a estrutura local da proteína, na posição do aminoácido codificado por esse códon.
Os pesquisadores enfatizam que as descobertas ainda não são capazes de esclarecer a direção da relação causal, o que significa que ainda não é possível dizer se uma mudança na codificação genética pode causar uma mudança na estrutura da proteína local ou se mudanças estruturais podem causar codificação diferente, por exemplo, através de processos evolutivos. Esta questão é a base para um estudo de pesquisa subsequente que está sendo realizado pelo grupo. According to Dr. Marx, a biologist by training and education, "If we find in subsequent research that the codon indeed has a causal effect on protein folding, this is likely to have a huge impact on our understanding of protein folding, as well as on future applications, such as engineering new proteins."
Dr. Marx emphasizes that the discovery presented in the article would not have been possible without Prof. Bronstein's computer and analysis skills. "This research is truly interdisciplinary, because biology alone cannot cope with such vast quantities of data without the help of data science, and computer scientists cannot themselves perform research of this kind, since they lack familiarity with the complex biological processes being probed. Therefore, our research highlights the huge advantage of interdisciplinary research that integrates skills from different fields to create a whole that is greater than the sum of its parts."
