Tecnologia que simula interações moleculares complexas pode levar a melhores tratamentos para câncer e COVID-19
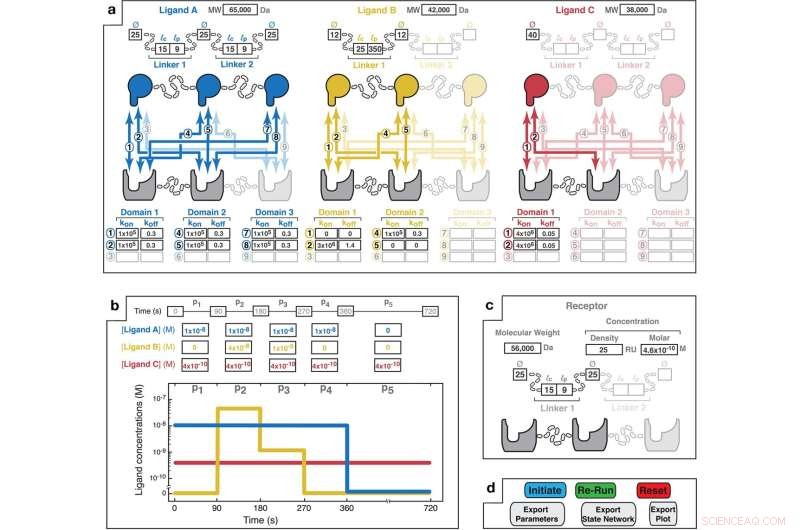
A interface de projeto de entrada do MVsim fornece especificação de parâmetros interativos para sistemas de interação multivalente e multimolecular. um Uma interface de apontar e clicar permite ao usuário selecionar o número de ligantes (até três) e valências do(s) ligante(s) e receptor (até trivalente) que compõem o sistema multivalente. Com base no desenho escolhido, o usuário especifica a estrutura de cada um dos ligantes, inserindo o peso molecular aplicável (MW); os diâmetros do domínio de ligação (Ø); os comprimentos de contorno (lc dos ligantes (ou seja, a distância máxima de ponta a ponta; por exemplo, 3,5 Å e 1,5 Å por aminoácido para uma bobina aleatória e hélice alfa, respectivamente); e os comprimentos de persistência (lp) do ligantes. Além disso, as interações combinatórias aplicáveis (numeradas de 1 a 9) exclusivas para cada pareamento receptor-ligante são destacadas. Os campos de parâmetro permitem a entrada de constantes de taxa monovalentes para cada interação de pares. As interações não vinculativas podem ser indicadas com k ligado e k desligado valores de zero (por exemplo, como ilustrado com Ligando B em amarelo para interações "1" e "5"). b Um campo de entrada permite que o usuário especifique padrões das concentrações totais de ligantes em massa. Uma fase de associação ocorre durante períodos de concentração de ligante em massa diferente de zero (por exemplo, 90-270 s para Ligando B). As fases de dissociação ocorrem quando o ligante é removido da solução em massa (por exemplo, 360-720 s para o Ligando A). Aqui, o Ligante C é especificado como continuamente presente na solução durante os 720 s do curso de tempo da interação. A exibição gráfica permite a visualização do padrão de pulso de concentração em massa especificado. c Parâmetros de entrada do usuário para o receptor. A concentração do receptor pode ser especificada como uma densidade de superfície que mimetiza SPR (medida em RU; onde 1 RU é igual a ~1 pg/mm
2
) ou uma concentração molar. A topologia do receptor é especificada na mesma forma descrita acima para os ligantes. d O MVsim A guia do controlador permite a iniciação, iteração e exportação de simulações de vinculação. "Initiate" executa uma simulação. "Re-executar" executa uma simulação abreviada usada quando nenhuma alteração foi feita na valência ou topologia do sistema. "Redefinir" reinicia o aplicativo e limpa os parâmetros de entrada do usuário de todos os campos. Crédito:Comunicação da Natureza (2022). DOI:10.1038/s41467-022-32496-6
Uma equipe liderada por engenheiros biomédicos da Universidade de Minnesota Twin Cities desenvolveu um aplicativo universalmente acessível que pode simular interações moleculares complexas, o que permitirá aos pesquisadores projetar melhores tratamentos para doenças como câncer e COVID-19.
O artigo se baseia em um estudo que os pesquisadores publicaram em 2019. Agora, eles expandiram a tecnologia para simular interações moleculares ainda mais complexas, facilitaram o uso do aplicativo para não especialistas e aplicaram suas descobertas para esclarecer como o SARS -CoV-2 vírus infecta o corpo.
O estudo foi publicado na
Nature Communications , e o aplicativo, chamado MVsim, está disponível gratuitamente para outros pesquisadores no GitHub.
O simulador prevê a força, velocidade e seletividade de interações multivalentes, que envolvem moléculas que possuem múltiplos sítios de ligação e podem ser usadas para desenvolver medicamentos para doenças, principalmente câncer e COVID-19.
“As interações multivalentes são realmente importantes em sistemas biológicos naturais, e agora estão começando a ser exploradas criativamente para criar novas drogas terapêuticas que alavancam suas propriedades de ligação únicas”, disse Casim Sarkar, autor sênior do artigo e professor da Universidade de Minnesota. Departamento de Engenharia Biomédica.
"Com drogas multivalentes, você pode, em princípio, atingir células muito especificamente de uma maneira que não é possível com drogas monovalentes padrão, mas há muitas variáveis a serem consideradas em seu design e muito do trabalho no campo até agora foi feito através de tentativa e erro experimental", acrescentou Sarkar. "Agora, usando o MVsim, somos capazes de fazer boas previsões que podem ser usadas para projetar mais racionalmente essas terapias".
Muitos medicamentos contra o câncer não apenas se ligam às células tumorais, mas também às células que não deveriam ter como alvo, o que muitas vezes cria efeitos colaterais indesejados para o paciente. Ao otimizar a especificidade das interações multivalentes usando o MVsim, os pesquisadores podem projetar medicamentos que visam mais especificamente as células de um tumor, minimizando a ligação a outras células do corpo.
Outro exemplo é o vírus SARS-CoV-2. Os cientistas sabem que o vírus está evoluindo para infectar melhor nossas células e escapar de nosso sistema imunológico, mas os mecanismos moleculares por trás de como o vírus faz isso são relativamente desconhecidos. Usando sua tecnologia MVsim, os pesquisadores da Universidade de Minnesota foram capazes de explorar esse processo mais profundamente, descobrindo as taxas em que os domínios de ligação individuais dentro da proteína multivalente do vírus alternam entre um estado de infecção celular e um estado de evasão imune.
“Essencialmente, temos um microscópio computacional que nos permite olhar sob o capô e ver o que proteínas multivalentes, como a proteína spike SARS-CoV-2, estão fazendo em nível molecular”, explicou Sarkar. "Este nível de detalhe molecular é difícil de capturar com um experimento físico. Um dos verdadeiros poderes do MVsim é que podemos não apenas aprender mais sobre como esses sistemas funcionam, mas também podemos usar essa ferramenta para projetar novas interações multivalentes para doenças como câncer e COVID-19."
Os pesquisadores já identificaram possíveis maneiras de limitar a infectividade das variantes atuais e futuras do SARS-CoV-2, que planejam testar em breve.
+ Explorar mais Engineered multivalent self-assembled binder protein against SARS-CoV-2 RBD
