Novo modelo captura a velocidade irregular de proteínas de cópia de DNA em bactérias
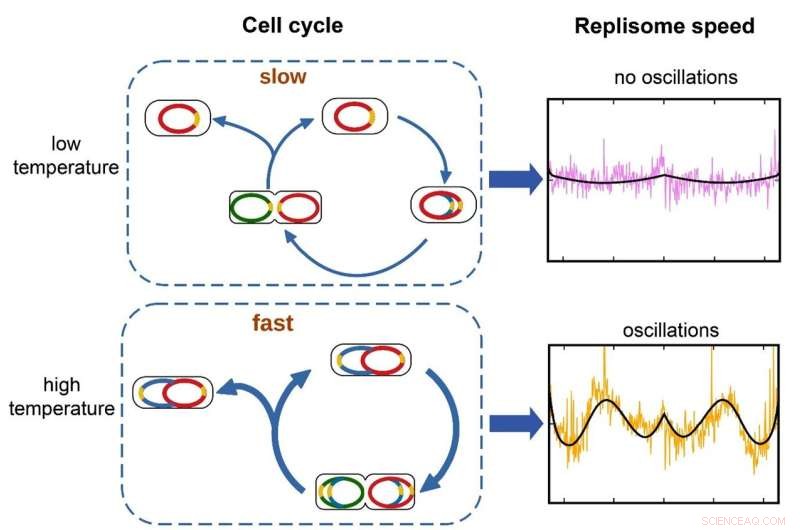
Quando a taxa de crescimento é rápida, como quando as bactérias são cultivadas em alta temperatura, aparecem oscilações na velocidade de resposta. Isso pode ser devido a múltiplas cópias do genoma sendo replicações ao mesmo tempo. Em taxas de crescimento lentas, quando apenas um genoma é copiado por vez, as oscilações desaparecem. Crédito:Instituto de Ciência e Tecnologia de Okinawa
A divisão celular é fundamental para a vida, permitindo que os organismos cresçam, reparem tecidos e se reproduzam. Para uma célula se dividir, todo o DNA dentro da célula (o genoma) deve primeiro ser copiado, em um processo chamado replicação do DNA. Mas a dinâmica precisa dos replissomas – a maquinaria proteica que copia o DNA – tem sido difícil para os cientistas determinarem.
Agora, pesquisadores do Instituto de Ciência e Tecnologia de Okinawa (OIST) no Japão desenvolveram um novo modelo que pode determinar variações na velocidade com que os replisomas copiam genomas bacterianos. O modelo, combinado com experimentos, mostra que certas seções de DNA são copiadas mais rapidamente do que outras e revela uma ligação intrigante entre velocidade de replicação e taxa de erro. A pesquisa foi publicada em
eLife em 25 de julho de 2022.
"As máquinas que copiam o DNA são incríveis - são muito rápidas e muito precisas", disse Simone Pigolotti, professora associada da OIST que chefia a Unidade de Complexidade Biológica. "Compreender essas máquinas pode nos dizer o que é importante para as células - quais erros são toleráveis, quais não são, quão rápida deve ser a replicação."
O modelo baseia-se em medir a quantidade de diferentes localizações de DNA dentro de uma população de células bacterianas que estão em constante divisão. Nas bactérias, para iniciar a replicação do DNA, dois replissomas se ligam ao DNA em um ponto de origem definido e seguem em direções opostas ao longo da alça do DNA, copiando o DNA até se encontrarem do outro lado. Isso significa que o DNA mais próximo do ponto de origem é copiado primeiro, enquanto o DNA mais próximo do ponto de terminação é copiado por último.
"Se você deixar uma população de bactérias crescer livremente, então, a qualquer momento, a maioria das células estará no processo de divisão celular. Como a replicação do DNA sempre começa no mesmo local, isso significa que se você sequenciar todo o DNA , haverá uma maior abundância de DNA que está mais próximo do ponto de origem e uma quantidade muito menor de DNA que está mais próxima do ponto final", explicou o Prof. Pigolotti.
No estudo, pesquisadores da Unidade de Química e Engenharia de Ácidos Nucleicos do OIST cultivaram bactérias Escherichia coli (E. coli) em diferentes temperaturas. A Seção de Sequenciamento então sequenciou o DNA da bactéria.
Ao analisar as características da curva de distribuição, os pesquisadores foram capazes de determinar a velocidade exata da maquinaria da proteína. Eles descobriram que, à medida que a temperatura aumentava, a velocidade de replicação aumentava. Ainda mais interessante, os pesquisadores descobriram que os replissomas variavam sua velocidade em diferentes pontos ao longo do genoma.
Uma razão potencial para sua velocidade flutuante, especula o Prof. Pigolotti, é que pode haver limites nos recursos necessários para a replicação, como nucleotídeos – os blocos de construção do DNA.
Em E. coli, quando as condições são boas, uma única célula bacteriana pode se dividir a cada 25 minutos. Mas o processo de replicação do DNA leva mais tempo – cerca de 40 minutos. Portanto, para manter altas taxas de crescimento, várias cópias do genoma são replicadas ao mesmo tempo, o que aumenta o número de replissomas em ação. A competição por nucleotídeos poderia então fazer com que os replissomas diminuíssem.
Evidências adicionais corroboram essa hipótese. Em baixas temperaturas e em culturas pobres em nutrientes, quando a taxa de crescimento da bactéria é baixa e apenas um genoma seria copiado por vez, essas flutuações na velocidade de replicação desaparecem.
Curiosamente, os pesquisadores também descobriram que as oscilações observadas na velocidade de replicação também correspondiam às oscilações na taxa de mutação documentadas em outros estudos. Quando eles sobrepuseram os dois padrões, descobriram que as áreas do genoma que foram copiadas mais rapidamente também tinham uma taxa de mutação mais alta.
"Isso parece intuitivo - se pensarmos em uma ação, como digitar em um teclado, quanto mais rápido digitarmos, maior a probabilidade de cometermos um erro", disse o Prof. Pigolotti. "Então, achamos que quando os replisomes são mais rápidos, sua taxa de erro é maior."
Para o Prof. Pigolotti, o próximo passo é determinar como a velocidade de replicação muda em cepas mutantes de E. coli, como aquelas que não possuem proteínas que auxiliam na replicação. Ele também está curioso para ver se o padrão se mantém em outras cepas de bactérias.
"É uma direção de pesquisa realmente emocionante", disse o Prof. Pigolotti. "E todo o trabalho foi feito em colaboração com outras unidades daqui. É o tipo de colaboração interdisciplinar que só pode acontecer na OIST."
+ Explorar mais Duas proteínas retardam o trem de replicação do DNA em Drosophila
