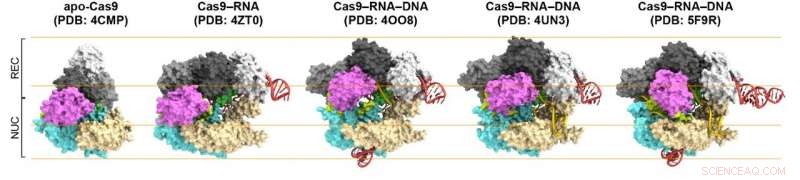
 p Figura 1. Estruturas do Cas9. Da esquerda para a direita:Cas9 sozinho (apo-Cas9), Cas9 ligado ao RNA (Cas9-RNA), Cas9 – RNA ligado ao seu alvo de DNA de fita simples (Cas9 – RNA – DNA), Cas9 – RNA ligado a um duplex parcial de DNA (Cas9 – RNA – DNA) e Cas9 – RNA ligado ao seu alvo de DNA de fita dupla (Cas9 – RNA – DNA). Crédito:Kanazawa University
p Figura 1. Estruturas do Cas9. Da esquerda para a direita:Cas9 sozinho (apo-Cas9), Cas9 ligado ao RNA (Cas9-RNA), Cas9 – RNA ligado ao seu alvo de DNA de fita simples (Cas9 – RNA – DNA), Cas9 – RNA ligado a um duplex parcial de DNA (Cas9 – RNA – DNA) e Cas9 – RNA ligado ao seu alvo de DNA de fita dupla (Cas9 – RNA – DNA). Crédito:Kanazawa University
p Pesquisadores da Universidade de Kanazawa e da Universidade de Tóquio relatam em
Nature Communications a visualização da dinâmica de 'tesouras moleculares' - o principal mecanismo da técnica de engenharia genética CRISPR-Cas9. p Uma das técnicas usadas em engenharia genética - o processo de modificar artificialmente o genoma de um organismo vivo - envolve o chamado sistema de nuclease CRISPR-Cas9. Usando este sistema, o DNA de uma célula pode ser cortado em um local desejado, onde os genes podem ser deletados ou adicionados. A seleção do local a ser cortado é feita por uma molécula de 'RNA guia' ligada à proteína Cas9. Agora, uma equipe de pesquisadores liderada por Mikihiro Shibata da Universidade de Kanazawa e Osamu Nureki da Universidade de Tóquio visualizou a dinâmica do complexo CRISPR-Cas9, em particular como ele corta o DNA, fornecendo informações valiosas sobre o mecanismo de clivagem de DNA mediado por CRISPR-Cas9.
p Para seus estudos de visualização, os cientistas usaram microscopia de força atômica de alta velocidade (HS-AFM), um método para imagens de superfícies. Uma superfície é sondada movendo um pequeno cantilever sobre ela; a força experimentada pela sonda pode ser convertida em uma medida de altura. Uma varredura de toda a superfície resulta em um mapa de altura da amostra. A configuração experimental de alta velocidade de Shibata e colegas permitiu extremamente rápido, varreduras repetidas - conversíveis em filmes - das biomoléculas que participam da ação de tesoura molecular.
p Primeiro, os cientistas compararam Cas9 sem e com RNA anexado (Cas9-RNA). Eles descobriram que o primeiro era capaz de adotar com flexibilidade várias conformações, enquanto o último tem um fixo, estrutura de dois lóbulos, destacando a capacidade de estabilização conformacional do RNA guia. Então, Shibata e colegas observaram como o complexo Cas9 – RNA estabilizado tem como alvo o DNA. Eles confirmaram que ele se liga a um local do motivo adjacente do protoespaçador pré-selecionado (PAM) no DNA. Um PAM é uma sequência curta de nucleotídeos localizada próximo ao local alvo do DNA, que é complementar ao RNA guia.
p Os filmes de alta velocidade da equipe de pesquisa revelaram ainda que a segmentação ('interrogatório de DNA') é alcançada por meio da difusão 3-D do complexo Cas9-RNA. Finalmente, os pesquisadores conseguiram visualizar a dinâmica do próprio processo de clivagem:eles observaram como a região da 'tesoura molecular' sofre flutuações conformacionais depois que Cas9-RNA desenrola localmente o DNA de fita dupla (Filme 1 [URL]).
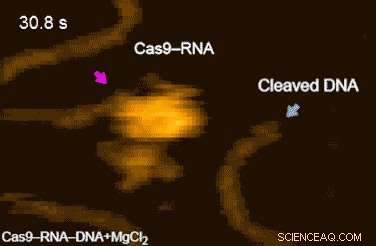 p Figura 2. Filmes HS-AFM de clivagem de DNA por Cas9-RNA. As flutuações do domínio da nuclease são indicadas por setas magenta. Os produtos de clivagem liberados do Cas9 – RNA são indicados por setas azuis. Crédito:Kanazawa University
p Figura 2. Filmes HS-AFM de clivagem de DNA por Cas9-RNA. As flutuações do domínio da nuclease são indicadas por setas magenta. Os produtos de clivagem liberados do Cas9 – RNA são indicados por setas azuis. Crédito:Kanazawa University
p O trabalho de Shibata avança nossa compreensão do mecanismo de edição do genoma CRISPR-Cas9. Nas palavras dos pesquisadores:"... este estudo fornece detalhes sem precedentes sobre a dinâmica funcional do CRISPR-Cas9, e destaca o potencial de HS-AFM para elucidar os mecanismos de ação de nucleases efetoras guiadas por RNA de sistemas CRISPR-Cas distintos. "
p
CRISPR-Cas9
p CRISPR, abreviatura de "repetições palindrômicas curtas com espaçamento regular em cluster", refere-se a um conjunto de sequências de DNA bacteriano contendo fragmentos do DNA de vírus que atacaram a bactéria anteriormente. Esses fragmentos são usados pela bactéria para evitar novos ataques dos mesmos vírus. "Cas" refere-se a genes associados a CRISPR; "Cas9" é uma proteína associada a CRISPR com dois domínios de nuclease (A nuclease é uma enzima capaz de clivar ácidos nucleicos, moléculas orgânicas presentes no DNA e no RNA).
p Nos últimos anos, foi desenvolvida uma técnica de engenharia genética em que um complexo CRISPR-Cas9 atua como 'tesoura molecular'; a nuclease Cas9 se liga a uma molécula de RNA guia que contém informações sobre o sítio de DNA a ser alvo. Usando microscopia de força atômica de alta velocidade, Mikihiro Shibata da Universidade de Kanazawa e colegas agora estudaram a dinâmica do complexo CRISPR-Cas9 em grande detalhe.
p
Força atômica microscópica
p A microscopia de força atômica (AFM) é uma técnica de imagem na qual a imagem é formada pela varredura de uma superfície com uma ponta muito pequena. O movimento de varredura horizontal da ponta é controlado por meio de elementos piezoelétricos, enquanto o movimento vertical é convertido em um perfil de altura, resulting in a height distribution of the sample's surface. As the technique does not involve lenses, its resolution is not restricted by the so-called diffraction limit. In a high-speed setup, AFM can be used to produce movies of a sample's evolution in real time. High-speed AFM has been used successfully to study protein dynamics, for example myosin V walking on an actin filament, the photo-induced conformational change of bacteriorhodopsin, and the degradation of cellulose. Shibata and colleagues have now applied the high-speed AFM technique for visualizing the dynamics of DNA cleavage by CRISPR-Cas9.

