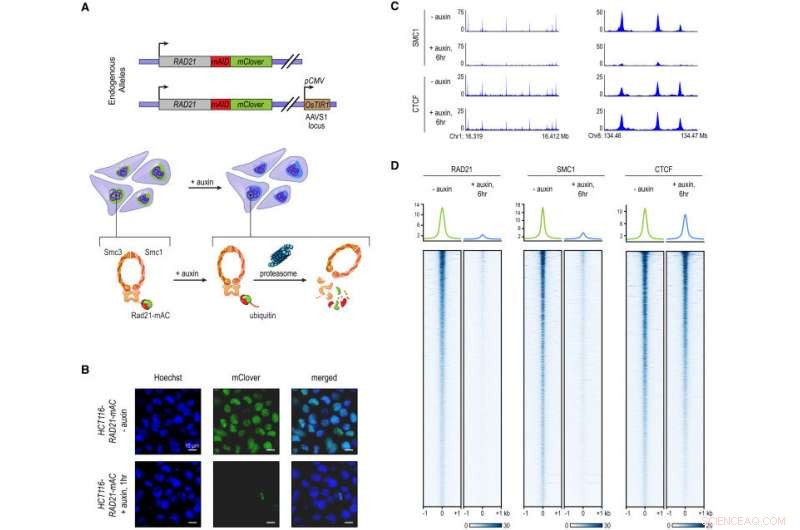
 p A marcação de RAD21 endógeno com um degradador indutível por auxina permite o rápido, Perda de Coesina Quase Completa. (A) Em células HCT-116-RAD21-mAC, ambos os alelos RAD21 são marcados com degrons indutíveis por auxina e um repórter mClover, e o gene OsTIR1 está integrado no locus AAVS1. O tratamento com auxina leva à degradação proteassomal de RAD21. (B) Imagens de células vivas após coloração com Hoechst 33342 para marcar núcleos. A fluorescência nuclear do mClover correspondente ao RAD21 marcado foi perdida após 1 hora de tratamento com auxina. (C) Sinal SMC1 e CTCF ChIP-seq com e sem tratamento com auxina. (D) RAD21, SMC1, e sinal CTCF ChIP-seq (à esquerda, meio, direita) em todos os picos chamados para cada uma das proteínas em células RAD21-mAC não tratadas. Superior:Enriquecimento médio para cada proteína. Após a degradação de RAD21, o complexo de coesina não se liga mais à cromatina. A ligação de CTCF não é afetada. Crédito: Célula . DOI:10.1016 / j.cell.2017.09.026
p A marcação de RAD21 endógeno com um degradador indutível por auxina permite o rápido, Perda de Coesina Quase Completa. (A) Em células HCT-116-RAD21-mAC, ambos os alelos RAD21 são marcados com degrons indutíveis por auxina e um repórter mClover, e o gene OsTIR1 está integrado no locus AAVS1. O tratamento com auxina leva à degradação proteassomal de RAD21. (B) Imagens de células vivas após coloração com Hoechst 33342 para marcar núcleos. A fluorescência nuclear do mClover correspondente ao RAD21 marcado foi perdida após 1 hora de tratamento com auxina. (C) Sinal SMC1 e CTCF ChIP-seq com e sem tratamento com auxina. (D) RAD21, SMC1, e sinal CTCF ChIP-seq (à esquerda, meio, direita) em todos os picos chamados para cada uma das proteínas em células RAD21-mAC não tratadas. Superior:Enriquecimento médio para cada proteína. Após a degradação de RAD21, o complexo de coesina não se liga mais à cromatina. A ligação de CTCF não é afetada. Crédito: Célula . DOI:10.1016 / j.cell.2017.09.026
p Uma equipe multi-institucional abrangendo Baylor College of Medicine, Rice University, A Universidade de Stanford e o Broad Institute of MIT e Harvard criaram o primeiro mapa 4-D de alta resolução de dobra do genoma, rastrear todo o genoma humano à medida que ele se dobra ao longo do tempo. O relatório, o que pode levar a novas maneiras de entender as doenças genéticas, aparece na capa de
Célula . p
Fazendo conexões
p Por décadas, pesquisadores suspeitaram que, quando uma célula humana responde a um estímulo, Elementos de DNA que estão distantes no genoma se encontram rapidamente, formando loops ao longo do cromossomo. Ao reorganizar esses elementos de DNA no espaço, a célula é capaz de alterar quais genes estão ativos.
p Em 2014, a mesma equipe de cientistas mostrou que era possível mapear esses loops. Mas os primeiros mapas eram estáticos, sem a capacidade de observar a mudança dos loops. Não estava claro se, no espaço lotado do núcleo, Os elementos do DNA poderiam se encontrar com rapidez suficiente para controlar as respostas celulares.
p "Antes, poderíamos fazer mapas de como o genoma se dobrou quando estava em um determinado estado, mas o problema com uma imagem estática é que, se nada mudar, é difícil descobrir como as coisas funcionam, "disse Suhas Rao, primeiro autor do novo estudo. "Nossa abordagem atual é mais como fazer um filme; podemos assistir as dobras à medida que desaparecem e reaparecem."
p
Um anel para governar todos eles
p Para acompanhar o processo de dobra ao longo do tempo, a equipe de pesquisa começou interrompendo a coesina, um complexo de proteínas em forma de anel localizado nos limites de quase todos os loops conhecidos. Em 2015, a equipe propôs que a coesina cria laços de DNA no núcleo da célula por um processo de extrusão.
p "A extrusão funciona como o ajustador do comprimento da alça em uma mochila, "explicou o Dr. Erez Lieberman Aiden, diretor do Center for Genome Architecture do Baylor College of Medicine e autor sênior do novo estudo. "Quando você passa a correia pelos dois lados, ele forma um loop. O DNA parece estar fazendo a mesma coisa - exceto que os anéis de coesina parecem desempenhar o papel de ajustadores. "
p Aiden disse que uma previsão crucial do modelo de 2015 é que todos os loops devem desaparecer na ausência de coesina. Na nova pesquisa, Aiden, Rao e seus colegas testaram essa suposição.
p "Descobrimos que, quando interrompemos a coesina, milhares de loops desapareceram, "disse Rao, um estudante de medicina na Universidade de Stanford, Hertz Fellow e membro do laboratório Aiden. "Então, quando colocamos a coesina de volta, todos esses loops voltam - geralmente em questão de minutos. Isso é precisamente o que você poderia prever a partir do modelo de extrusão, e sugere que a velocidade com que a coesina se move ao longo do DNA está entre as mais rápidas para qualquer proteína humana conhecida. "
p
Loops versus grupos
p Mas nem tudo aconteceu como os pesquisadores esperavam. Em alguns casos, os loops fizeram exatamente o oposto do que os pesquisadores previram.
p "Enquanto víamos milhares de loops em todo o genoma ficarem mais fracos, notamos um padrão engraçado, "disse Aiden, também um McNair Scholar, Hertz Fellow e pesquisador sênior do Centro de Física Biológica Teórica da Rice University. "Havia alguns loops estranhos que estavam realmente se tornando mais fortes. Então, conforme colocamos a coesina de volta, a maioria dos loops se recuperou totalmente - mas esses loops estranhos novamente fizeram o oposto - eles desapareceram! "
p Ao examinar como os mapas mudaram ao longo do tempo, a equipe percebeu que a extrusão não era o único mecanismo que unia os elementos do DNA. Um segundo mecanismo, chamado compartimentalização, não envolveu coesina.
p "O segundo mecanismo que observamos é bastante diferente da extrusão, "explicou Rao." A extrusão tende a reunir os elementos do DNA dois de cada vez, e apenas se estiverem no mesmo cromossomo. Este outro mecanismo pode conectar grandes grupos de elementos uns aos outros, mesmo que estejam em cromossomos diferentes. E parece ser tão rápido quanto a extrusão. "
p Eric Lander, diretor do Broad Institute, um co-autor do estudo, disse, "Estamos começando a entender as regras pelas quais os elementos do DNA se reúnem no núcleo. Agora que podemos rastrear os elementos conforme eles se movem ao longo do tempo, os mecanismos subjacentes estão começando a ficar mais claros. "
