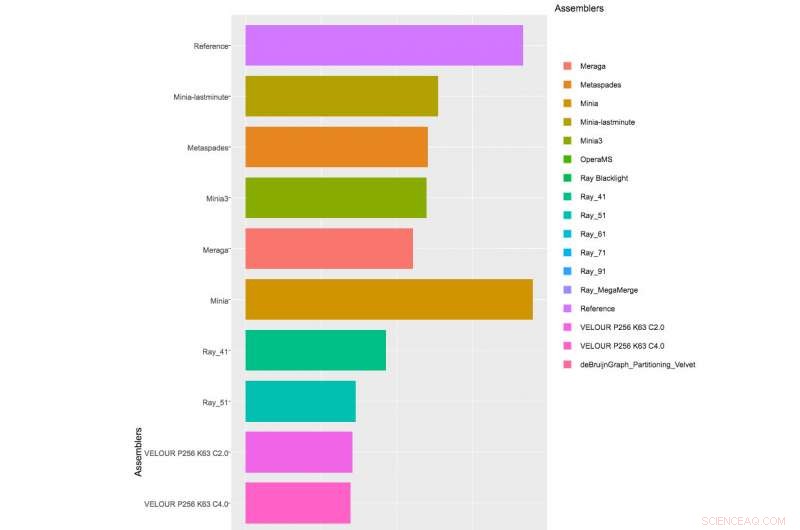
Tabela mostrando resultados parciais de montadores aplicados ao 1º Desafio CAMI, Conjunto de dados 1. Clique aqui para ver a tabela completa.
Eles estão por todas as partes, mas invisível a olho nu. Os micróbios são invisíveis, forças influentes por trás da regulamentação dos principais processos ambientais, como o ciclo do carbono, no entanto, a maioria deles permanece desconhecida. Por mais de uma década, o Instituto Conjunto do Genoma do Departamento de Energia dos EUA (DOE JGI), um DOE Office of Science User Facility, tem permitido aos pesquisadores estudar micróbios não cultivados, incapazes de crescer em laboratório, usando abordagens de última geração, como sequenciamento genômico de alto rendimento de comunidades ambientais ("metagenômica") e o desenvolvimento de ferramentas computacionais para descobrir e caracterizar comunidades microbianas do ambiente. Para lidar com a montagem de metagenomas em um conjunto de segmentos de DNA sobrepostos que juntos representam uma região de consenso de DNA ou contigs, em seguida, colocando esses contigs em caixas de genoma, e, finalmente, a realização de perfis taxonômicos de caixas de genoma, analistas de todo o mundo desenvolveram uma série de diferentes ferramentas computacionais, no entanto, até agora não havia consenso sobre como avaliar seu desempenho.
Publicado em 2 de outubro, Em 2017 Métodos da Natureza , uma equipe incluindo pesquisadores do DOE JGI descreveu os resultados do Desafio de Avaliação Crítica da Interpretação de Metagenoma (CAMI), o primeiro de sempre, avaliação de benchmarking organizada pela comunidade de ferramentas computacionais para metagenomas. O Desafio CAMI foi liderado por Alexander Sczyrba, chefe do grupo de Metagenômica Computacional na Universidade de Bielefeld e ex-bolsista de pós-doutorado do DOE JGI, e Alice McHardy, chefe do Laboratório de Biologia Computacional de Pesquisa de Infecção no Helmholtz Center for Infection Research.
"É muito difícil para os pesquisadores descobrir qual programa usar para um determinado conjunto de dados e análise com base nos resultados de documentos de método, "disse McHardy." Os conjuntos de dados e medidas de avaliação usados nas avaliações variam amplamente. Outro problema é que os desenvolvedores geralmente gastam muito tempo comparando o estado da arte ao avaliar o desempenho de um novo software dessa maneira. A CAMI quer mudar essas coisas e envolver a comunidade na definição de padrões e melhores práticas para avaliação e na aplicação desses princípios em desafios de benchmarking. "
O CAMI Challenge aconteceu ao longo de três meses em 2015. Para avaliar as ferramentas computacionais, os organizadores desenvolveram 3 conjuntos de dados de metagenoma simulados usando mais de 300 esboços de genomas de isolados bacterianos e de archaea sequenciados e montados pelo DOE JGI, que faziam parte do projeto Genomic Encyclopedia of Bacteria and Archaeal publicado recentemente em Nature Biotechnology . Esses genomas foram compartilhados com o consórcio CAMI Challenges antes de serem divulgados ao público para facilitar o benchmarking objetivo de diferentes ferramentas. Os conjuntos de dados também incluíram cerca do mesmo número de genomas do Instituto Max Planck em Colônia, Alemanha, junto com elementos circulares e vírus. Os conjuntos de dados simulados eram um único conjunto de dados de amostra de 15 bilhões de bases (Gb), um conjunto de dados de 40 Gb com 40 genomas e 20 elementos circulares, e um conjunto de dados de série temporal de 75 Gb composto por cinco amostras e incluindo centenas de genomas e elementos circulares.
"JGI tem um grande interesse em benchmarking de ferramentas e tecnologias que avancem na análise de metagenômicas e melhorem a qualidade dos dados que fornecemos aos usuários. Tendo publicado o primeiro estudo sobre o uso de conjuntos de dados simulados para benchmarking de ferramentas metagenômicas do JGI, é ótimo ver como essa metodologia se expandiu ao longo dos anos e agora, por meio deste estudo, evoluindo para um modelo de esforços comunitários padronizados no campo, "disse Nikos Kyrpides, Chefe do programa DOE JGI Prokaryote Super.
"A JGI está muito empenhada não apenas em benchmarking de protocolos de laboratório, mas também fluxos de trabalho computacionais, "acrescentou o chefe do DOE JGI Microbial Genomics, Tanja Woyke." Isso torna a nossa participação em esforços comunitários essenciais, como o CAMI, tão importante. "
Com mais de 40 equipes inscritas para o Desafio, e os organizadores do CAMI receberam 215 inscrições de 25 programas em todo o mundo, embora apenas 17 equipes estivessem dispostas a ter suas implementações de software publicadas. Os organizadores do CAMI avaliaram as ferramentas computacionais em 3 categorias. Meia dúzia de montadores e pipelines de montagem foram avaliados na montagem de sequências de genoma geradas a partir de tecnologias de sequenciamento de leitura curta. No desafio de binning, five genome binners and 4 taxonomic binners were evaluated on criteria including the tools' efficacy in recovering individual genomes. Finalmente, 10 taxonomic profilers with various parameter settings were evaluated on how well they could predict the identities and relative abundances of the microbes and circular elements. The benchmarking results are available on https://data.cami-challenge.org/results.
The CAMI organizers are already planning future benchmarking challenges, for example to evaluate and aid method development for long read sequencing technologies. "CAMI is an ongoing initiative, " noted Sczyrba. "We are currently further automating the benchmarking and comparative result visualizations. And we invite everyone interested to join and work with CAMI on providing comprehensive performance overviews of the computational metagenomics toolkit, to inform developers about current challenges in computational metagenomics and applied scientists of the most suitable software for their research questions."