Novos nanodispositivos de RNA em células vivas podem detectar e analisar múltiplos sinais complexos
 p O ácido ribonucléico (RNA) é usado para criar circuitos lógicos capazes de realizar vários cálculos. Em novos experimentos, Green e seus colegas incorporaram portas lógicas de RNA em células bacterianas vivas, que agem como pequenos computadores. Crédito:Jason Drees para o Biodesign Institute
p O ácido ribonucléico (RNA) é usado para criar circuitos lógicos capazes de realizar vários cálculos. Em novos experimentos, Green e seus colegas incorporaram portas lógicas de RNA em células bacterianas vivas, que agem como pequenos computadores. Crédito:Jason Drees para o Biodesign Institute
p O nexo interdisciplinar de biologia e engenharia, conhecida como biologia sintética, está crescendo em um ritmo rápido, abrindo novas perspectivas que dificilmente poderiam ser imaginadas há pouco tempo. p Em uma nova pesquisa, Alex Green, professor do Instituto de Biodesign da ASU, demonstra como células vivas podem ser induzidas a realizar cálculos na forma de pequenos robôs ou computadores.
p Os resultados do novo estudo têm implicações significativas para o design inteligente de medicamentos e a distribuição inteligente de medicamentos, produção de energia verde, tecnologias de diagnóstico de baixo custo e até mesmo o desenvolvimento de nanomáquinas futurísticas capazes de caçar células cancerosas ou desligar genes aberrantes.
p "Estamos usando interações RNA-RNA muito previsíveis e programáveis para definir o que esses circuitos podem fazer, "diz Green." Isso significa que podemos usar um software de computador para projetar sequências de RNA que se comportam da maneira que queremos em uma célula. Isso torna o processo de design muito mais rápido. "
p O estudo aparece na edição online avançada da revista
Natureza .
p
RNA Designer
p A abordagem descrita usa circuitos compostos de ácido ribonucléico ou RNA. Esses projetos de circuito, que se assemelham a circuitos eletrônicos convencionais, automontar em células bacterianas, permitindo que eles percebam as mensagens recebidas e respondam a elas produzindo uma saída computacional específica, (nesse caso, uma proteína).
p No novo estudo, circuitos especializados conhecidos como portas lógicas foram projetados no laboratório, em seguida, incorporado em células vivas. Os minúsculos interruptores do circuito são acionados quando as mensagens (na forma de fragmentos de RNA) se ligam às suas sequências de RNA complementares no circuito celular, ativando a porta lógica e produzindo uma saída desejada.
p Os interruptores de RNA podem ser combinados de várias maneiras para produzir portas lógicas mais complexas, capazes de avaliar e responder a várias entradas, assim como um simples computador pode pegar várias variáveis e realizar operações sequenciais como adição e subtração para chegar a um resultado final.
p O novo estudo melhora drasticamente a facilidade com que a computação celular pode ser realizada. A abordagem apenas de RNA para a produção de nanodispositivos celulares é um avanço significativo, já que os esforços anteriores exigiam o uso de intermediários complexos, como proteínas. Agora, as peças de ribocomputação necessárias podem ser prontamente projetadas no computador. As propriedades simples de emparelhamento de bases das quatro letras de nucleotídeos do RNA (A, C, G e U) asseguram a previsível automontagem e funcionamento dessas peças dentro de uma célula viva.
p O trabalho de Green nesta área começou no Wyss Institute em Harvard, onde ajudou a desenvolver o componente central usado nos circuitos celulares, conhecido como switch toehold de RNA. O trabalho foi realizado enquanto Green era um pós-doutorando trabalhando com o especialista em nanotecnologia Peng Yin, junto com os biólogos sintéticos James Collins e Pamela Silver, que são todos co-autores do novo artigo. “Os primeiros experimentos foram em 2012, "Green diz." Basicamente, as chaves de apoio tiveram um desempenho tão bom que queríamos encontrar uma maneira de explorá-las da melhor forma para aplicativos de celular. "
Este vídeo demonstra os princípios básicos do switch toehold de RNA. Crédito:Arizona State University p Depois de chegar na ASU, O primeiro aluno de graduação de Green, Duo Ma, trabalhou em experimentos no Instituto Biodesign, enquanto outro pós-doutorado, Jongmin Kim continuou um trabalho semelhante no Wyss Institute. Ambos também são co-autores do novo estudo.
p
Chip Pentium da natureza
p A possibilidade de usar DNA e RNA, as moléculas da vida, para realizar cálculos semelhantes aos de computador foi demonstrado pela primeira vez em 1994 por Leonard Adleman, da University of Southern California. Desde então, o rápido progresso avançou consideravelmente no campo, e recentemente, tal computação molecular foi realizada dentro de células vivas. (As células bacterianas são geralmente utilizadas para este propósito, pois são mais simples e fáceis de manipular.)
p A técnica descrita no novo artigo tira vantagem do fato de que o RNA, ao contrário do DNA, é de fita simples quando é produzida nas células. Isso permite que os pesquisadores projetem circuitos de RNA que podem ser ativados quando uma fita de RNA complementar se liga a uma sequência de RNA exposta no circuito projetado. Esta ligação de fitas complementares é regular e previsível, com nucleotídeos A sempre emparelhados com U e C sempre emparelhados com G.
p Com todos os elementos de processamento do circuito feitos usando RNA, que pode assumir um número astronômico de sequências potenciais, o verdadeiro poder do método recém-descrito reside em sua capacidade de realizar muitas operações ao mesmo tempo. Essa capacidade de processamento paralelo permite uma computação mais rápida e sofisticada, ao mesmo tempo em que faz uso eficiente dos recursos limitados da célula.
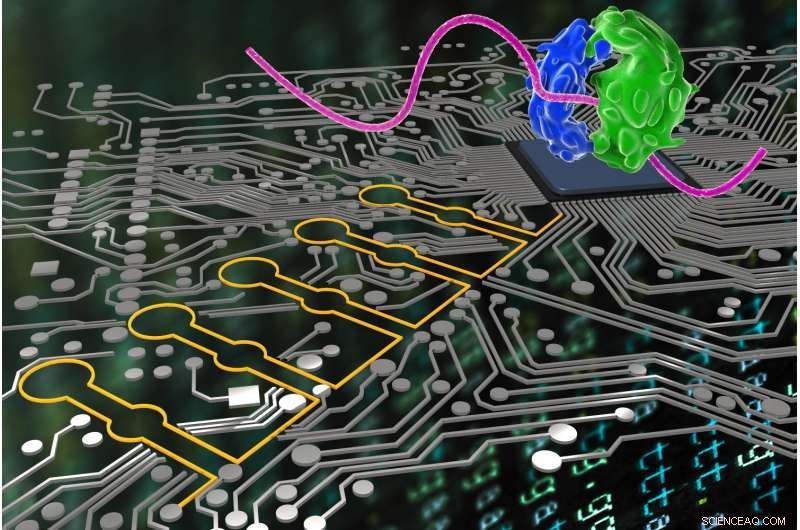 p Semelhante a como os cientistas da computação usam linguagem lógica para fazer com que seus programas tornem E precisos, OU e NÃO decisões em direção a uma meta final, "Dispositivos de ribocomputação" (estilizados aqui em amarelo) desenvolvidos por uma equipe do Wyss Institute agora podem ser usados por biólogos sintéticos para detectar e interpretar vários sinais em células e instruir logicamente seus ribossomos (estilizados em azul e verde) para produzir diferentes proteínas. Crédito:Wyss Institute da Harvard University
p Semelhante a como os cientistas da computação usam linguagem lógica para fazer com que seus programas tornem E precisos, OU e NÃO decisões em direção a uma meta final, "Dispositivos de ribocomputação" (estilizados aqui em amarelo) desenvolvidos por uma equipe do Wyss Institute agora podem ser usados por biólogos sintéticos para detectar e interpretar vários sinais em células e instruir logicamente seus ribossomos (estilizados em azul e verde) para produzir diferentes proteínas. Crédito:Wyss Institute da Harvard University
p
Resultados lógicos
p No novo estudo, portas lógicas conhecidas como AND, OR e NOT foram projetados. Uma porta AND produz uma saída na célula apenas quando duas mensagens de RNA A AND B estão presentes. Uma porta OR responde a A OU B, enquanto uma porta NOT bloqueará a saída se uma determinada entrada de RNA estiver presente. A combinação dessas portas pode produzir uma lógica complexa capaz de responder a várias entradas.
p Usando interruptores de base de RNA, os pesquisadores produziram os primeiros dispositivos de ribocomputação capazes de AND de quatro entradas, OR de seis entradas e um dispositivo de 12 entradas capaz de realizar uma combinação complexa de AND, Lógica OR e NOT conhecida como expressão de forma normal disjuntiva. Quando a porta lógica encontra as sequências de ligação de RNA corretas que levam à ativação, um interruptor de suporte é aberto e o processo de tradução em proteína ocorre. Todas essas funções de detecção de circuito e saída podem ser integradas na mesma molécula, tornando os sistemas compactos e mais fáceis de implementar em uma célula.
p A pesquisa representa a próxima fase do trabalho em andamento usando os interruptores de base de RNA altamente versáteis. Em trabalhos anteriores, Green e seus colegas demonstraram que um barato, Uma matriz de chaves de base de RNA baseada em papel pode atuar como uma plataforma altamente precisa para o diagnóstico do vírus Zika. A detecção de RNA viral pela matriz ativou os interruptores do pé, desencadeando a produção de uma proteína, que é registrado como uma mudança de cor na matriz.
p O princípio básico do uso de dispositivos baseados em RNA para regular a produção de proteínas pode ser aplicado a praticamente qualquer entrada de RNA, inaugurando uma nova geração de precisão, diagnósticos de baixo custo para uma ampla gama de doenças. A abordagem sem células é particularmente adequada para ameaças emergentes e durante surtos de doenças no mundo em desenvolvimento, onde recursos médicos e pessoal podem ser limitados.
p
O computador dentro
p De acordo com Green, o próximo estágio da pesquisa se concentrará no uso da tecnologia de base de RNA para produzir as chamadas redes neurais dentro das células vivas - circuitos capazes de analisar uma gama de entradas excitatórias e inibitórias, calculando a média deles e produzindo uma saída, uma vez que um determinado limite de atividade é atingido, da mesma forma que um neurônio calcula a média dos sinais recebidos de outros neurônios. Em última análise, pesquisadores esperam induzir as células a se comunicarem umas com as outras por meio de sinais moleculares programáveis, formando um ambiente verdadeiramente interativo, rede semelhante a um cérebro.
p "Porque estamos usando RNA, uma molécula universal de vida, sabemos que essas interações também podem funcionar em outras células, portanto, nosso método fornece uma estratégia geral que pode ser transportada para outros organismos, "Green diz, aludindo a um futuro em que as células humanas se tornem entidades totalmente programáveis com extensas capacidades biológicas.

